R语言之决策树CART、C4.5算法
决策树是以树的结构将决策或者分类过程展现出来,其目的是根据若干输入变量的值构造出一个相适应的模型,来预测输出变量的值。预测变量为离散型时,为分类树;连续型时,为回归树。
常用的决策树算法:
| 算法 | 简介 | R包及函数 |
|---|---|---|
| ID3 | 使用信息增益作为分类标准 ,处理离散数据,仅适用于分类树 。 | rpart包 rpart() |
| CART | 使用基尼系数作为分类标准,离散、连续数据均可,适用于分类树,回归树。 | rpart包 rpart() |
| C4.5 | 使用信息增益和增益率相结合作为分类标准,离散、连续数据均可,但效率较低,适用于分类树 | RWeka包 J48() |
| C5.0 | 是C4.5用于大数据集的拓展,效率较高 | C50包 C5.0() |
建模过程:
1)选定一个最佳变量将全部样本分为两类,并且实现两类中的纯度最大化,其分类标准有“信息增益”,“增益率”,“基尼系数”等。具体理论可参考周志华老师的《机器学习》一书。
2)对每一个分类子集重复步骤1)。
3)重复步骤1)、2),直到没有分类方法能将不纯度下降到给定的阈值以下。
4)根据步骤3)得出的模型预测所属类别。
用到的程序包rpart, raprt.plot. RWake, sampling, partykit。 建模所用数据为UCI机器学习数据库里的威斯康星州乳腺癌数据集和汽车数据集mtcars。仅以CART和C4.5算法函数的调用为例,C5.0算法的调用可用C50包中的C5.0()函数。
一、CART算法
步骤:
1、数据预处理,建立好训练集和预测集。代码如下:可参见《R语言实战》。
> loc<-"http://archive.ics.uci.edu/ml/machine-learning-databases/"
> ds<-"breast-cancer-wisconsin/breast-cancer-wisconsin.data"
> url<-paste(loc,ds,sep="")
> data<-read.table(url,sep=",",header=F,na.strings="?")
> names(data)<-c("ID","clumpThickness","sizeUniformity",
+ "shapeUniformity","maginalAdhesion",
+ "singleEpithelialCellSize","bareNuclei",
+ "blandChromatin","normalNucleoli",
+ "mitosis","class")
> data$class[data$class==2]<-"良性"
> data$class[data$class==4]<-"恶性"
> data<-data[-1]
> set.seed(1234)
> train<-sample(nrow(data),0.7*nrow(data))
> tdata<-data[train,]
> vdata<-data[-train,] 2、用rpart()构建树
rpart()函数的格式:
rpart(formula,data,weights,subsets,na.action=na.rpart,method,parms,control…)
| 参数 | 参数解释 |
|---|---|
| formula | 如y1~y2+y3+y4或者简写形式y1~.(表示输入变量为除y的所有变量)。 |
| data | 训练集。 |
| subset | 从data中选取子集建模。 |
| na.action | 缺失值处理,默认为na.rpart。表示剔除缺失y值或者缺失所有输入变量的数据。 |
| method | 选择决策树的类型。”anova”对应回归树。“class”用于分类变量,对应分类树。“poisson”用于二分类变量,对应分类树。“exp”用于生存分析。 |
| parms | 此参数只适用于分类变量。若method=”class”,则parms=list(split,prior,loss).其中split可取“information”(信息增益分类规则),“gini” (基尼分类规则)。不同的分类规则分别对应上述不同的算法。 |
| control | 控制树的各种参数。control=list(minsplit,minbucket=round(minsplit/3),…) |
| minsplit | 每个节点中所含样本最小数,默认为10.当训练集样本数较少时,需自己设置此参数大小,否则可能因为样本数较少与此参数默认值冲突,导致画出来的树没有分支。 |
| minbucket | 叶节点中所含样本最小数. |
| cp | 复杂度参数,通常设定阈值用来剪枝。 |
利用rpart()函数对上述的训练集建决策树。
> library(rpart)
> dtree<-rpart(class~.,data=tdata,method="class", parms=list(split="information")) #使用ID3算法,若split="gini",则为CART算法。
> printcp(dtree)
Classification tree:
rpart(formula = class ~ ., data = tdata, method = "class", parms = list(split = "information"))
Variables actually used in tree construction:
[1] bareNuclei clumpThickness shapeUniformity sizeUniformity
Root node error: 160/489 = 0.3272
n= 489
CP nsplit rel error xerror xstd
1 0.800000 0 1.00000 1.00000 0.064846
2 0.046875 1 0.20000 0.27500 0.039549
3 0.012500 3 0.10625 0.16875 0.031567
4 0.010000 4 0.09375 0.16875 0.0315673、prune()剪枝,提高模型的泛化能力
格式: prune(tree,cp,…)
> dtree$cptable #剪枝前的复杂度 nsplit分枝数 rel error训练集对应的误差 xerror 10折交叉验证误差 xstd交叉验证误差的标准差
CP nsplit rel error xerror xstd
1 0.800000 0 1.00000 1.00000 0.06484605
2 0.046875 1 0.20000 0.27500 0.03954867
3 0.012500 3 0.10625 0.16875 0.03156665
4 0.010000 4 0.09375 0.16875 0.03156665
> tree<-prune(dtree,cp=0.0125)
> tree$cptable #剪枝后的复杂度及分枝数
CP nsplit rel error xerror xstd
1 0.800000 0 1.00000 1.00000 0.06484605
2 0.046875 1 0.20000 0.27500 0.03954867
3 0.012500 3 0.10625 0.16875 0.031566654、画出树图rpart.plot()
格式 rpart.plot(tree,type,fallen.leaves=T,branch,…)
| 参数 | 参数的解释 |
|---|---|
| tree | 画图所用的树模型。 |
| type | 可取1,2,3,4.控制图形中节点的形式。 |
| fallen.leaves | 默认值为T,在图的地段显示终端节点。 |
| branch | 控制图的外观。如branch=1,获得垂直树干的决策树。 |
树状图代码如下:
> opar<-par(no.readonly = T)
> par(mfrow=c(1,2))
> library(rpart.plot)
> rpart.plot(dtree,branch=1,type=2, fallen.leaves=T,cex=0.8, sub="剪枝前")
> rpart.plot(tree,branch=1, type=4,fallen.leaves=T,cex=0.8, sub="剪枝后")
> par(opar)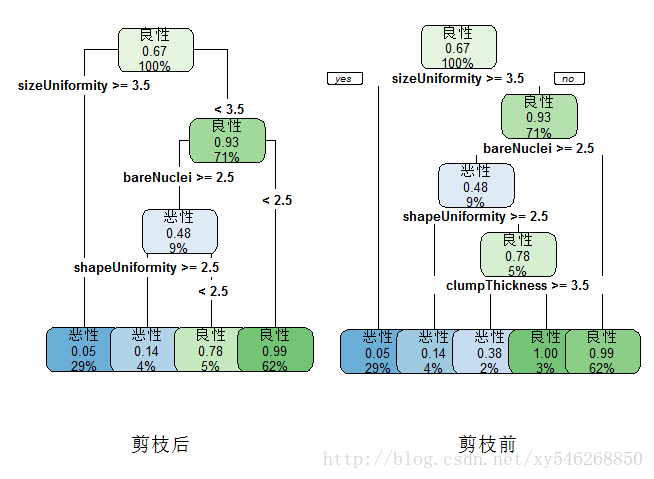
5、利用预测集检验模型效果predict()
格式 predict(fit,newdata,type,…)
> predtree<-predict(tree,newdata=vdata,type="class") #利用预测集进行预测
> table(vdata$class,predtree,dnn=c("真实值","预测值")) #输出混淆矩阵
预测值
真实值 恶性 良性
恶性 79 2
良性 7 122从混淆矩阵可以看出此模型准确率为(79+122)/(79+2+7+122)=95.71%
二、C4.5算法
RWeka包里的J48()可实现C4.5算法的调用。调用格式:
J48(formula,data,subset,na.action,control=Weka_control(u=T,c=0.25,M=2,R=T,N=3,B=T),options=NULL)
| 参数 | 参数的解释 |
|---|---|
| U | 默认值为TURE,表示不剪枝。 |
| C | 对剪枝过程设置的置信阈值。 |
| M | 默认值为2,表示叶节点最小样本量。 |
| R | 默认值为TURE,表示按错误率递减剪枝。 |
| N | 默认值为3,表示在R=T的情况下,交叉验证的折叠次数。 |
| B | 默认值为T,表示是否建立二叉树。 |
对R自带的数据集mtcars的变量mpg进行分类预测。
1、对mpg进行分组,创建训练集和预测集,代码如下:
> y<-quantile(mtcars$mpg,c(.8,.6,.4,.2)) #对mpg的数值取分位数
> mtcars$group[mtcars$mpg1]&mtcars$mpg>=y[2]]<-"A"
> mtcars$group[mtcars$mpg2]&mtcars$mpg>=y[3]]<-"B"
> mtcars$group[mtcars$mpg3]&mtcars$mpg>=y[4]]<-"C"
> mtcars$group[mtcars$mpg4]]<-"D"
> mtcars$group<-factor(mtcars$group) #将要预测的变量定义为因子,否则J48()函数无法识别
> a<-round(3/4*sum(mtcars$group=="A"))
> b<-round(3/4*sum(mtcars$group=="B"))
> c<-round(3/4*sum(mtcars$group=="C"))
> d<-round(3/4*sum(mtcars$group=="D"))
library(sampling)
> sub<-strata(mtcars,stratanames="group",size=c(a,b,c,d),method="srswor") #对分组变量采用分层抽样,保证样本的代表性
> tdata<-mtcars[sub$ID_unit,] #训练集
> vdata<-mtcars[-sub$ID_unit,] #预测集 2、创建分类树J48() `
> library(RWeka)
> ctree<-J48(group~.,data=tdata,control=Weka_control(M=2))
> print(ctree)
J48 pruned tree
------------------
mpg <= 19.7
| mpg <= 14.7: D (5.0)
| mpg > 14.7
| | mpg <= 17.8: C (4.0)
| | mpg > 17.8: B (4.0)
mpg > 19.7: A (10.0)
Number of Leaves : 4
Size of the tree : 7
> library(partykit)
> plot(ctree,type="simple") #画出树图,结果如下,由于训练集的数据较少,只有23条数据,最后的树图仅以mpg作为分支变量,也直接影响了最后的准确率。
3、预测predict()
> pretree<-predict(ctree,newdata=vdata)
> table(pretree,vdata$group,dnn=c("预测值","真实值")) #输出混淆矩阵
真实值
预测值 A B C D
A 4 1 0 0
B 0 0 0 0
C 0 0 3 1
D 0 0 0 0
> pretree
[1] A A C C C A A A C
Levels: A B C D
> vdata$group
[1] A A C C C A A B D
Levels: A B C D准确率为77%。因为数据较少,所以准确率会稍低。
