python机器学习手写算法系列——DBSCAN聚类
本文,就像本系列的其他文章一样。旨在通过阅读原论文+手写代码的方式,自己先把算法搞明白,然后再教其他人。手写代码除了可以验证自己是否搞明白以外,我会对中间过程做图。这样,我可以通过图直观的验证算法是否正确。而这些图,又成为写文章时候的很好的素材。
什么是 DBSCAN
DBSCAN,全称是 Density-Based Scan。 故名思意,就是通过密度扫描。DBSCAN是一种聚类算法,和KMeans相比,他不需要指定cluster的数量。他的主要参数有两个,半径和邻居的数量。Scikit-Learn中,半径用 ϵ \epsilon ϵ(epsilon)表示,邻居的数量用min-samples表示。我们这里也借用sklearn的表示方式。这样大家使用sklearn的时候不会搞混。
当然,除了sklearn,在weka,R,elki等库里,也有DBSCAN的实现。他在教科书里也经常被提及,并有很多成功的实际运用。许多基于密度的聚类算法,也都受了DBSCAN的启发。实践证明改算法是有效的,并在2014年获得了SIGKDD的test-of-time大奖。
和KMeans比较
为什么有了KMeans,还要有DBSCAN,肯定是KMeans有解决不了的问题。
比如,我们画两个月亮。
import numpy as np
from matplotlib import pyplot as plt
from matplotlib.patches import Circle
from sklearn.cluster import DBSCAN, KMeans
from sklearn.datasets import make_moons
from sklearn.metrics.pairwise import euclidean_distances
X, y = make_moons(n_samples=1000, noise=0.05, random_state=42)
plt.figure(figsize=(12, 6))
plt.scatter(X[:,0], X[:,1])
plt.show()
km = KMeans(n_clusters=2)
km.fit(X)
plt.figure(figsize=(12, 6))
# Plot the clusters
plt.scatter(X[:, 0], X[:, 1], c=km.labels_)
plt.title(f"Kmeans (k=2)")
plt.show()
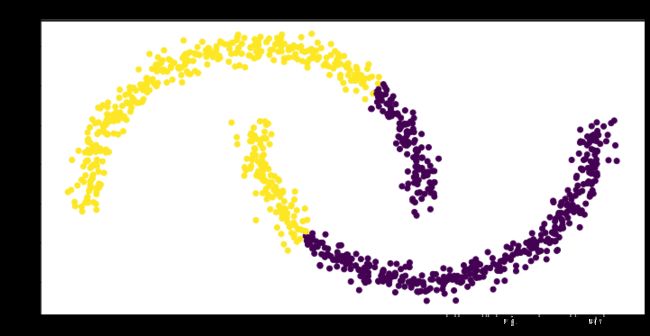
这里,我们可以看到,我们本来希望两个月亮能分别对应两个簇(cluster),但是事与愿违。这是因为,KMeans是按照距离来聚类的。一个月亮的边缘,距离其中心的距离,可能大于其距离另一个月亮中心的距离。这样,这些点就被另一个月亮带跑了。
DBSCAN 算法
第一步:找核心(core)点
这时,DBSCAN就出现了。他不是基于距离的,而是基于密度的。他把高密度的区域都连起来,形成一个簇。
这里,我们就需要半径( ϵ \epsilon ϵ)和最小邻居数量(min-samples)来确定高密度区了。
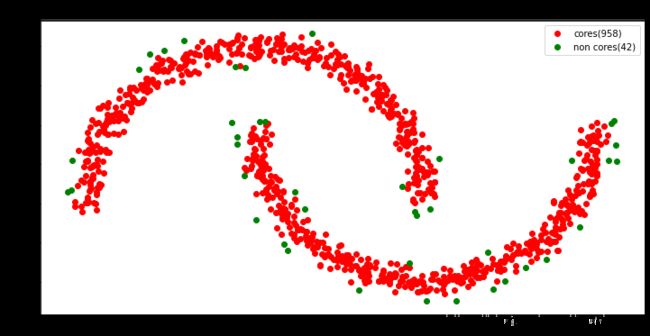
首先,我们要找到一些中心点。他们的半径( ϵ \epsilon ϵ)之内,有min-samples个点。我们这里把epsilon和min-samples设置为0.1和10。
这时找到的这些中心点,叫做core。
# set the parameters
epsilon = 0.1
min_samples = 10
# In most of the cluster algorithms, the first step is to calculate the distance matrix.
distance_matrix=euclidean_distances(X)
neighbour_predicate_matrix = distance_matrix<=epsilon
neighbour_counts_array=neighbour_predicate_matrix.sum(axis=0)
core_predicates = neighbour_counts_array>=min_samples
indices = np.arange(0, 1000)
core_indices = indices[core_predicates]
non_core_indices = indices[~core_predicates]
n_cores = sum(core_predicates)
n_non_cores = 1000-n_cores
plt.figure(figsize=(12, 6))
# cores
plt.scatter(X[core_indices,0], X[core_indices,1], c='r', label=f"cores({n_cores})" )
plt.scatter(X[non_core_indices,0], X[non_core_indices,1], c='g', label=f"non cores({n_non_cores})")
plt.legend()
plt.title("Cores and Non-cores")
plt.show()
第二步:连接核心点
用了这些core,我们就要把他们都连接起来了。
labels = -np.ones((1000,), dtype=np.int)
cluster_id=0
loop_index=0
while sum(labels<0) > 0:
#unlabelled_predicates = (labels==-1)
unlabelled_core_predicates = ((core_predicates) & (labels==-1))
unlabelled_core_indices = indices[unlabelled_core_predicates]
if len(unlabelled_core_indices) == 0:
break
first_index=unlabelled_core_indices[0]
neighbourhood_predicates = neighbour_predicate_matrix[first_index]
neighbourhood_indices = indices[core_predicates & neighbourhood_predicates]
while(len(neighbourhood_indices)>0):
fig, ax = plt.subplots(figsize=(12, 6))
fig.facecolor='white'
circle = Circle((X[first_index, 0], X[first_index, 1]), 0.1, facecolor='none',
edgecolor=(0, 0.8, 0.8), linewidth=3, alpha=0.5)
ax.add_patch(circle)
# plt.figure(figsize=(10, 8))
# cores
plt.scatter(X[core_indices,0], X[core_indices,1], c='r')
plt.scatter(X[non_core_indices,0], X[non_core_indices,1], c='g')
# clustered
plt.scatter(X[labels>=0,0], X[labels>=0,1], c=labels[labels>=0])
# and its neighourhood
plt.scatter(X[neighbourhood_indices, 0], X[neighbourhood_indices, 1], c="tab:blue", marker='o')
# draw first
plt.scatter(X[first_index, 0], X[first_index, 1], c="tab:cyan", marker='o')
plt.title(f"Cluster {cluster_id}, Loop {loop_index}")
plt.show()
# plt.savefig(f"tmp/{loop_index}.png")
plt.clf()
labels[neighbourhood_indices] = cluster_id
unlabelled_predicates = (labels==-1)
neighbourhood_predicates_matrix = neighbour_predicate_matrix[neighbourhood_indices]
neighbourhood_predicates = neighbourhood_predicates_matrix.any(axis=0)
neighbourhood_indices = indices[(core_predicates & neighbourhood_predicates & unlabelled_predicates)]
loop_index+=1
# break
cluster_id+=1
我把这里打印的64张fig做成了gif。我们看到。算法先找到一个core,然后找到他半径( ϵ \epsilon ϵ)之内的core,把他们聚为一簇。然后,再找这个core的相邻的core,并置为一簇。以此类推,知道找不到新的core为止。这时,在剩下的core里,有发起一个新的簇。用上面的方法继续找,直到找不到为止。
第三步:连接非核心点,并找出异常点
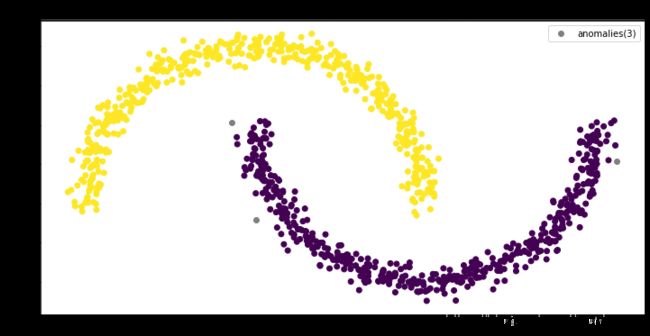
剩下的非核心点,我们可以直接认为都是异常点,也可以看他们是否在核心点的旁边,如果是,则继承核心点的簇,否则为异常点。这个完全看需要了。
clusters = np.unique(labels)
for cluster_id in [c for c in clusters if c>-1]:
# find the points that are already clusterd to cluster_id
cluster_predicates = labels==cluster_id
# find the neighours of the above
neighbours_matrix = neighbour_predicate_matrix[cluster_predicates]
neighbours_predicates = neighbours_matrix.any(axis=0)
# remove the already clustered points from the neighourhood.
clustered_non_core_indices=indices[neighbours_predicates & ~cluster_predicates]
# label the rest neighours.
labels[clustered_non_core_indices] = cluster_id
plt.figure(figsize=(12, 6))
# cores
#plt.scatter(X[core_indices,0], X[core_indices,1], c='r')
#plt.scatter(X[non_core_indices,0], X[non_core_indices,1], c='g')
anomaly_predicates = labels==-1
# clustered
plt.scatter(X[~anomaly_predicates,0], X[~anomaly_predicates,1], c=labels[labels>=0])
# anomalies
n_anomalies = sum(anomaly_predicates)
plt.scatter(X[anomaly_predicates,0], X[anomaly_predicates,1], c='gray', label=f'anomalies({n_anomalies})')
plt.legend()
plt.title("DBSCAN (from scratch) with epsilon=0.1 and min_samples=10")
plt.show()
和scikit-learn比较
def cluster_and_plot(epsilon, min_samples):
dbscan = DBSCAN(eps=epsilon, min_samples=min_samples)
dbscan.fit(X)
plt.figure(figsize=(12, 6))
# Plot the clusters
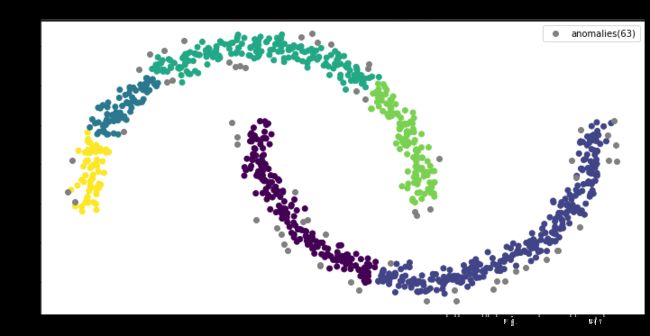
plt.scatter(X[dbscan.labels_>-1, 0], X[dbscan.labels_>-1, 1], c=dbscan.labels_[dbscan.labels_>-1])
# plot anomolies
anomaly_predicates = dbscan.labels_==-1
n_anomalies = sum(anomaly_predicates)
plt.scatter(X[anomaly_predicates, 0], X[anomaly_predicates, 1], c='gray', label=f'anomalies({n_anomalies})')
plt.title(f"SCAN with epsilon={epsilon} and min_samples={min_samples}")
plt.legend()
plt.show()
cluster_and_plot(0.1, 10)
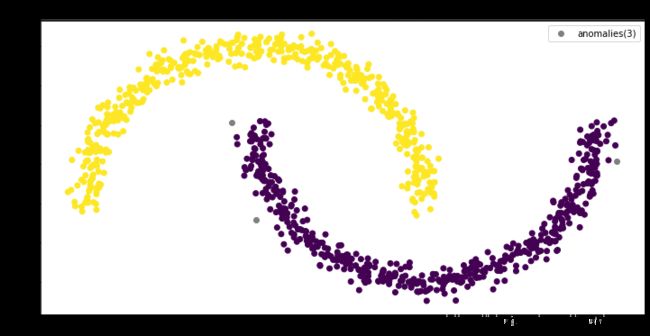
我们看到,scikit-learn的结果和我们手写算法的结果是一致的。证明我们对算法的理解正确。
参数选择的启发
根据DBSCAN的作者们最新的论文,min-samples参数建议为2 * 维度。当效果不好的时候,可以增大。
ϵ \epsilon ϵ比较难设置。根据算法的设计, ϵ \epsilon ϵ越小越好。当然,这个也受距离算法的影响。专家经验也可以运用到 ϵ \epsilon ϵ的选择。比如在GPS地图上, ϵ \epsilon ϵ设置为1公里。
这里,我们对上面的数据做了几个实验。我们保持min-samples=4(2*dim)不变,然后调整 ϵ \epsilon ϵ。我们看到当 ϵ \epsilon ϵ为0.03和0.04的时候,效果并不好。然后,我们增大 ϵ \epsilon ϵ到0.06,这时两个月亮被正确的分开了。接着,我们正大 ϵ \epsilon ϵ,我们看到异常点的数量在下降。
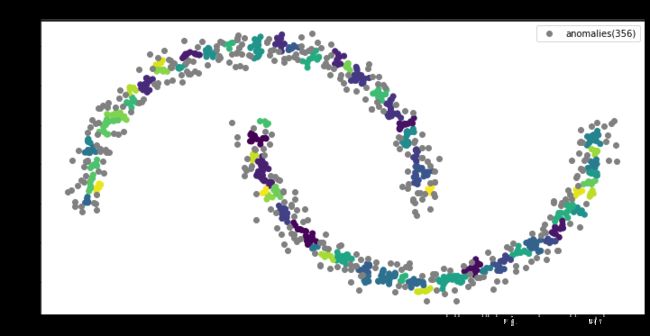

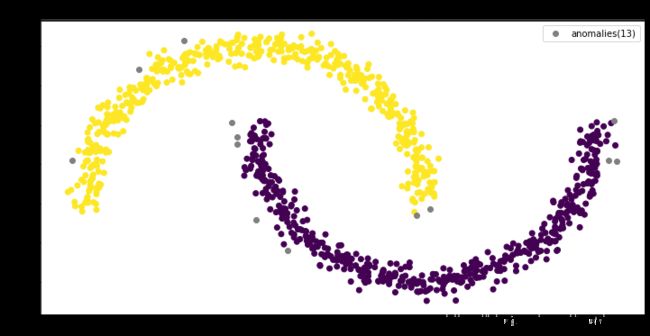
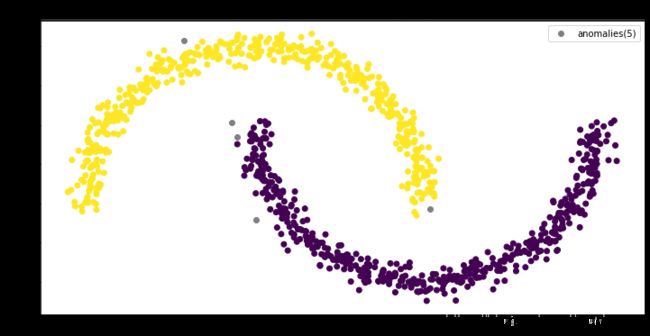
我想,在实际项目中,还是要根据实际情况区设置算法的参数的。比如,我们现在用DBSCAN去找信用卡行为异常。这时,银行可能要验证我们的算法,我们需要给银行一个最异常的客户。这时,我们就可以增大 ϵ \epsilon ϵ,以减少异常点。
但是如果反过来,另一个项目,预算也有效,他们只想找出最典型的客户,也是越少越好。这时别说异常点了,可能非核心点都要去掉。
总结
DBSCAN是一个神奇的算法,他即是聚类算法,又是异常检测算法。和KMeans不同,他把连续的区域聚为一类。我们应该根据实际需要选择算法及参数。当然,前提是你首先要了解这些算法。
代码地址
https://github.com/EricWebsmith/machine_learning_from_scrach
参考资料
书: Hands-on Machine Learning with Scikit-Learn and Tensorflow
论文: DBSCAN Revisited, Revisited: Why and How You Should (Still) Use DBSCAN