【机器学习案例实践】使用SVM对Iris数据集特征提取及分类
【机器学习案例实践】使用SVM对Iris数据集特征提取及分类
实验简介
本实验使用的数据集很经典,实验本身是基于SVM支持向量机技术对数据集Iris进行特征分类。实验采取Sklearn函数库来实现SVM,并使用SVM对提取好的特征进行分类,结果的展示方面进行了数据可视化保证观测结果清晰可见。
首先,Iris数据集的中文名是安德森鸢尾花卉数据集,Iris包含150个样本,对应数据集的每行数据。每行数据包含每个样本的四个特征和样本的类别信息,所以iris数据集是一个150行5列的二维表形式的样本,被应用于多类模型的实验当中。
还需要进一步介绍数据集的内容:数据集当中,每个样本包含了花萼长度、花萼宽度、花瓣长度、花瓣宽度四个特征(前4列),我们需要建立一个分类器,分类器可以通过样本的四个特征来判断样本属于山鸢尾、变色鸢尾还是维吉尼亚鸢尾('Iris-setosa', 'Iris-versicolor' , 'Iris-virginica')。当然数据集默认为str分类的,位于第五列,而由于在分类中类别标签必须为数字量,所以应将Iris.data中的第5列的类别(字符串)转换为数字形式。这是需要进行人工清洗的一个过程。
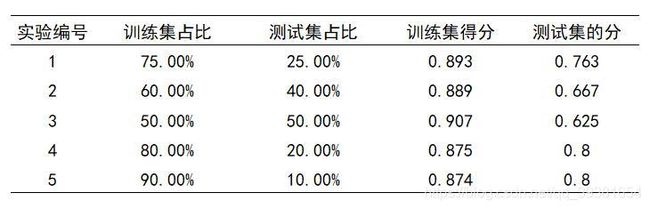
数据清洗完毕后就开始利用数据集进行分类器的构建,Sklearn非常方便可以根据需要将原始数据集按照比例依据类别标签划分成训练集和测试集,本次实验过程中分别按照默认比例和个人设置比例作了5组对照实验。划分好训练集和测试集之后,就可以开始训练SVM模型了,对核函数和参数的选择将直接影响到模型的泛化误差和预测准确度,在此处按照之前写过的文章分成四组进行对照。在训练完模型之后,采用可视化方法将预测区域和样本呈现出来,可以比较直观地观测到模型的预测精度。具体对照组的对照情况和具体实验过程将在后文中写出。
实验环境
软件环境:
Windows 10操作系统,Pycharm IDE环境。
硬件环境:
12线程CPU,16G内存,6G显存
第三方函数库:
- Sklearn函数库(用于构建SVM模型和数据集划分)= Sci-Kit learn库
- Numpy函数库(用于基本数据清洗和对数据格式进行处理会用于数据可视化过程中)
- Matplotlib函数库(用于数据可视化过程便于对比实验结果)
实验目标
我们现在得到了一组兰花数据集,在这组数据中包含三种不同种类的兰花,我们打算使用SVM对其进行分类,并通过这组实验来评价SVM的分类效果和参数设置对模型泛化误差的影响。
实验过程
首先数据集是.data格式的文件,根据观察和了解得知,其中包括4维特征值,和1维的标志位,而标志位还是以字符串形式给出的,所以需要先对数据集进行清洗和特征提取,为了方便后期可视化展示,在本次实验过程中只取其中前两维特征来进行实验。
数据清洗可以通过字典过滤来实现,使用loadtxt函数中的参数converters来实现字典转换。将第五列特征特征提取过程使用的是numpy库当中的split函数,该函数可以实现将前四维特征与后面的部分分离,这里用到一个以前不太常见的切片写法(4,),在这里特别记录一下。具体代表的含义为以4,5列中心点未划分划为两个array保存在两个参数中(注意结果为两个输出)。所以我们可以通过这个函数来实现将数据和标志分离,并保持其一一对应,相对位置不变。
为了实验能够有显著的结果,而且为了保证训练集和测试集能有着良好的数据一致性,使用train_test_split函数将原始数据集对训练集和测试及进行划分,该方法是有默认分配比例的(训练集:测试集=7.5:2.5),但也可以认为指定参数来进行修改。本次试验分别取了5组比例进行对照。由于,数据集包含两部分(数据和标签),所以当分离结束后,需要使用4个变量来接收返回值(train_data, test_data, train_label, test_label)。
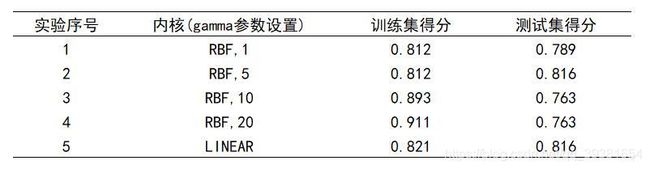
当以上对数据的处理结束,就可以开始构建SVM分类器了,Sklearn提供了很好的分类器实现,我将在这里介绍其参数配置,以及实验的对照设计。分类器的实现总共包含3个主要参数(训练对应关系decision_function_shape,学习步长c和核函数的选择kernel),当选择的核为rbf高斯核的时候还会需要配置gamma径向基核速度参数。本次试验中步长给定为5且对应关系为一对多ovr,这两个变量作为控制变量保持不变。对照组分别使用高斯核(不同gamma)和线性核,进行对照设计实验。
设计好对照组之后,将不同对照组的实验数据跑出进行对比。同时给出可视化展示。
实验代码
第三方函数库的导入部分:
# Author:JinyuZ1996
# Creation date:2020/8/10 10:24
# -*- coding:utf-8 -*-
from sklearn import svm
import numpy as np
import matplotlib.pyplot as plt
import matplotlib
from sklearn.model_selection import train_test_split函数定义部分:(本实验对函数封装不足是一个缺点后期改进)
# 函数定义部分
# 定义转换器字典用于转换对应兰花数字(相当于loadtxt函数转换器参数converters的字典)
def Sort_dic(type):
it = {b'Iris-setosa': 0, b'Iris-versicolor': 1, b'Iris-virginica': 2}
return it[type]
具体实现部分:(事实上我重点做了以下本实验的可视化部分并设置了对照)
关于接下来分类器定义这一部分:我需要详细讲一下参数问题,虽然上文中讲到了一部分,单还不够详细:
首先关于核函数的选取,kernel='linear'时,为线性核,C越大分类效果越好,但有可能会过拟合(defaul C=1)。而当kernel='rbf'时(default),为高斯核,gamma值越小,分类界面越连续;gamma值越大,分类界面越“散”,分类效果越好,但有可能会过拟合(在后面的图中能看出来)。
其次关于决策函数对应关系问题,当decision_function_shape='ovr'时,为one v rest(一对多),即一个类别与其他类别进行划分,而当decision_function_shape='ovo'时,为one v one(一对一),即将类别两两之间进行划分,用二分类的方法模拟多分类的结果。
# 具体实现部分
# 读取数据集的数据并进行简单清洗
path = 'Iris.data'
# converters是数据转换器定义,将第5列的花名格式str转化为0,1,2三种数字分别代表不同的类别兰花类别(这是一步数据清洗过程)
data = np.loadtxt(path, dtype=float, delimiter=',', converters={4:Sort_dic})
# 将数据和标签列划分开来
# split函数的参数意义(数据,分割位置(这里用了一种不常见的写法表示前四列为一组记作x,后面剩余部分为一组记作y),
# axis = 1(代表水平分割,以每一个行记录为切割对象) 或 0(代表垂直分割,以属性为切割对象))。
x, y = np.split(data, indices_or_sections=(4,), axis=1) # x为数据,y为标签
# 为便于后边画图显示,只选取前两维度。若不用画图,可选取前四列x[:,0:4]就选中所有特征了
x = x[:,0:2] # 标记一下,这个切片的意思是提取前两列的每一行[每一行,0,1两列]
# Sklearn库函数train_test_split可以实现将数据集按比例划分为训练集和测试集
train_data, test_data, train_label, test_label = train_test_split(x, y, random_state=1, train_size=0.8,test_size=0.2)
# 目前x为数据y为标签(即标注样本属于哪一类)
# 定义SVM分类器,希望大家还记得之前我们讲过的rbf核是什么
# C越大分类效果越好,但有可能会过拟合,gamma是高斯核参数,而后面的dfs制定了类别划分方式,ovr是一对多方式。
classifier = svm.SVC(C=5, kernel='rbf',gamma=20 ,decision_function_shape='ovr')
# 这里分类器的参数关系我会在实验报告中给出,比较复杂代码注释中不做详述
classifier.fit(train_data, train_label.ravel()) # 用训练集数据来训练模型。(ravel函数在降维时默认是行序优先)
# 计算svc分类器的准确率
print("Training_set_score:", format(classifier.score(train_data, train_label),'.3f'))
print("Testing_set_score:", format(classifier.score(test_data, test_label),'.3f'))
# 绘制图形将实验结果可视化(注意现在我们就挑了前二维特征来画图,好画,事实上该数据集有四维特征呢,不好画)
# 首先确定坐标轴范围,通过二维坐标最大最小值来确定范围
# 第1维特征的范围(花萼长度)
x1_min = x[:, 0].min()
x1_max = x[:, 0].max()
# 第2维特征的范围(花萼宽度)
x2_min = x[:, 1].min()
x2_max = x[:, 1].max()
# mgrid方法用来生成网格矩阵形式的图框架
x1, x2 = np.mgrid[x1_min:x1_max:200j, x2_min:x2_max:200j] # 生成网络采样点(其实是颜色区域),先沿着x1向右扩展,再沿着x2向下扩展
grid_test = np.stack((x1.flat, x2.flat), axis=1) # 再通过stack()函数,axis=1,生成测试点,其实就是合并横与纵等于计算x1+x2
grid_value = classifier.predict(grid_test) # 用训练好的分类器去预测这一片面积内的所有点,为了画出不同类别区域
grid_value = grid_value.reshape(x1.shape) # (大坑)使刚刚构建的区域与输入的形状相同(裁减掉过多的冗余点,必须写不然会导致越界读取报错,这个点的bug非常难debug)
# 设置两组颜色(高亮色为预测区域,样本点为深色)
light_camp = matplotlib.colors.ListedColormap(['#FFA0A0', '#A0FFA0', '#A0A0FF'])
dark_camp = matplotlib.colors.ListedColormap(['r', 'g', 'b'])
fig = plt.figure(figsize=(10, 5)) # 设置窗体大小
fig.canvas.set_window_title('SVM -2 feature classification of Iris') # 设置窗体title
# 使用pcolormesh()将预测值(区域)显示出来
plt.pcolormesh(x1, x2, grid_value, cmap=light_camp)
plt.scatter(x[:, 0], x[:, 1], c=y[:, 0], s=30, cmap=dark_camp) # 加入所有样本点,以深色显示
plt.scatter(test_data[:, 0], test_data[:, 1], c=test_label[:, 0], s=30, edgecolors='white', zorder=2,cmap=dark_camp)
# 单独再把测试集样本点加一个圈,更加直观的查看命中效果
# 设置图表的标题以及x1,x2坐标轴含义
plt.title('SVM -2 feature classification of Iris')
plt.xlabel('length of calyx')
plt.ylabel('width of calyx')
# 设置坐标轴的边界
plt.xlim(x1_min, x1_max)
plt.ylim(x2_min, x2_max)
plt.show()关于数据可视化部分,我再做一些补充,大家会发现我在这里使用了grid,这个来自numpy库函数,详细解释一下:
要想理解mgrid的参数,我们先假设现在要实现一个目标函数f(x,y) = x + y。x轴范围1~3,y轴范围4~6,当绘制图像时主要分四步进行:(这里参考的是https://blog.csdn.net/u012679707/article/details/80501358博主的解释,同样的数据集,我向前辈借鉴了数据可视化方法,站在巨人的肩膀上学习)
step1:x方向上的扩展(即沿x轴向右扩展):
[1 1 1]
[2 2 2]
[3 3 3]
step2:y方向上的扩展(即沿y轴向下扩展):
[4 5 6]
[4 5 6]
[4 5 6]
step3:定位(xi,yi)(其实就是合并两个矩阵):
[(1,4) (1,5) (1,6)]
[(2,4) (2,5) (2,6)]
[(3,4) (3,5) (3,6)]
step4:将(xi,yi)代入F(x,y) = x+y表示出来
因此这里x1, x2 = np.mgrid[x1_min:x1_max:200j, x2_min:x2_max:200j]后的结果可以看做是将两个矩阵拼合之后,创造了一片布满点的区域出来。
实验结果分析及结论
首先,我们先来看一下数据集划分对照的结果,我们这时候规定C=5,核为rbf核,ovr模式,gamma=10。
根据划分结果看,在接近75~80%:25~20%的训练集测试集比例时,结果开始趋于稳定,同时为了避免测试集数据过少缺乏一般性,可以认为默认的7.5:2.5的比例已经具备相对较好的观测水平(接下来的实验对照组均是以7.5:2.5来进行的)。
接下来开始对不同参数的分类器模型进行对照观测:
我们和之前对手写输入识别的数据计处理方法一样,主要针对的是径向基核的实验效果(因为速度快),当然也加入了一组线性核做对照。单从数据来看的话似乎,gamma取5的时候测试集表现出了很好的预测效果,但是竟然超过了训练集得分,我们认为这是不可靠的,事实上很好理解,gamma过低会导致径向基核欠拟合(当然过高也会过拟合),所以结果不可靠,不能认为是得到了很好的预测结果和模型参数。事实上,这一点也可以从下文的图中直观地感受到,因为我们绘制了预测区域,而这个区域和我们最终预测的部分重合度很低,是欠拟合的。
所以,我们应该关注的是,当gamma达到10-20的时候,测试集得分趋于稳定,我们认为在这个区间上的gamma值才是相对可靠的,这一点也可以通过下文的图来印证。有趣的是,我引入了一组线性核发现表现并不是很差(无论是在训练集还是在测试集),也许当实际应用需要的时候,也会选择牺牲linear核的错误率来提高分类速度。这在现实应用中往往是可以接受的。我在我的前一篇文章也就是手写输入识别案例报告中对这一部分也是有所提及的。
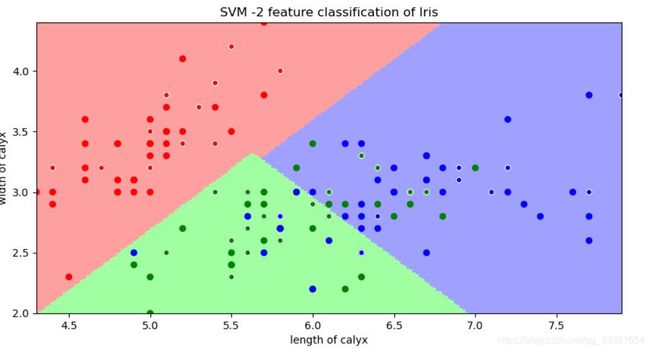
接下来展示不同的参数对照组的预测效果图像,一方面这可以帮助我们更直观的观察结果,另一方面这也是为了印证我刚刚所得出的结论:(三种不同的花类别用三种颜色代表,高亮区域是我们用grid构造的预测区域(也就是我们的模型),样本点的颜色代表着其数据集中的标签颜色,有白色环的为测试集样本)
本实验使用的数据集
https://download.csdn.net/download/qq_39381654/12710878