磁共振影像分析之: 基于FSL的VBM分析(1)
磁共振影像分析之: 基于FSL的VBM分析(1)
基本概念
在开始之前, 先给出几个基本概念:
磁共振成像
磁共振成像(Magnetic Resonance Imaging, MRI)是一种基于核磁共振原理和射频波探测组织结构和功能的医学影像技术. 值得一提的是, 磁共振成像技术没有辐射, 对人体没有影响.
结构磁共振(structural magnetic resonance imaging) 包括T1\T2和DTI三种数据, 据说T1观察解剖结构较好。T2显示组织病变较好。
传统的形态学测量方法对全脑或者感兴趣区域(ROI)计算灰质的体积. 比较费时, VBM将每个人的大脑都形成一个模板, 避免了人与人之间较大的解剖学差异, 并将结果经过平滑处理, 使得每个体素都代表它和邻居的平均值, 然后对不同个体间进行体素水平的比较.
VBM的基本概念:
VBM (voxel-based morphometry )可以在体素水平上定量检测出脑组织的密度和体积,反映不同群体或者个体局部脑区的脑组织成分与特征的差异. 目前, VBM已经广泛应用与描绘大脑结构的细微变化.
1995年, Wright首先提出了基于体素的形态学分析(VBM)思想
2000年, Ashburner提出了标准的VBM方法
2001年, Good等人提出了优化VBM的算法.
VBM方法简单高效, 有标准的处理流程, 包括很多软件提供自动处理功能. VBM的预处理流程可以大致总结为:
1) 从原始图像产生平均模板,也就是后文中提到的subject-specific模板
2)配准:将所有的原始图像通过仿射变换配准到1)生成的模板上
3)组织分割:灰质,白质和脑脊液
4)保持体积调制
5) 平滑处理, 用各向同性的高斯核
6)统计分析
FSL的VBM处理命令
如果想要从形态学上比较组间大脑局部灰质(voxel级别)的差别, 还有比如针对大脑功能的研究, 想要得到大脑功能上的差别是否由灰质差别造成的, 面对这样的问题就可以使用VBM.
由于人脑个体之间存在解剖学上的差异, 不能直接比较, 首先要将原始NifTI图像配准到标准模板, 包括必要的非线性对齐, 使得相同解剖位置的组织在空间上相对应. 这里的标准模板通常是用被试数据生成的subject-specific模板. 将所有图像匹配到标准模板上, 也就意味着将脑组织的空间位置\大小和分辨率和模板一致.
将配准后的大脑图像分割成灰质\白质和脑脊液以及非脑成分, 调制分割后的图像, 以得到能反映脑组织体积的图像.
上述两个步骤, 就是分割和配准的整体顺序不一定是完全分开. FSL给出的pipeline就是“大脑提取->线性配准->分割灰质白质和脑脊液->灰质的非线性配准->生成subject-specific模板”这样的流程.
得到配准的结果后, 为了增加参数检测的有效性, 需要做平滑, 经过平滑后每个体素的信号强度是周期体素的加权平均值.
这里有个配准度的问题, 要比较全脑voxel-by-voxel灰质差异, 你需要所有被试都是可比较的,也就是match的, 这就是为何要做非线性配准的原因. 但是如果配准结果太完美了, 所有被试的NifTi都和标准模板特别match, 那么做体素之间的差别分析就失去了意义. 这就是为何要限制非线性修正的自由度. 也就是在配准的前提下, 保留原大脑的基本形态结构.
也正是这个原因,请一定要谨记VBM分析方法是有一定的限制的. 有的时候可能会很难决定结果是由于灰质的厚度或者形态直接造成的, 还是由于不同的皮质折叠(gyrification) 模式间接造成的. 事实上, gyri/sulci 没有对齐, 甚至是皮层的不同折叠模式都会造成灰质分布的差异. 此外, 理论上结果还会与非线性对齐的自由度设置和光滑操作算法相关.
完成平滑的工作后, 就要进入统计建模和假设检验的环节. 通常采用广义线性模型并进行方差分析(analysis of variance, ANOVA). 将灰质的总体积\年龄\性别作为协变量, 以去除其对统计结果的影响,比较不同组之间的灰质体积差异, 用FWE多重比较检验,得到组间存在显著差异的统计结果图.
1 数据准备
首先, 在这里下载数据:
下载后解压, 创建文件夹 mkdir my_fsl_vbm并把所有的T1数据拷贝进去. VBM做比较时候, 如果分组不止一个, 而且每组的个数不相同, 比如数据最少的组里一共有N个被试, 那么其他组里随机选择N个被试, 生成study-specific模板, 将选做生成模板的数据名称单独放进template_list文件. 文件中就是各个被试的文件名称, 最好用组名的缩写开头.例如
CON_001.nii.gz
CON_002.nii.gz
…
CON_008.nii.gz
PAT_001.nii.gz
PAT_002.nii.gz
PAT_003.nii.gz
…
PAT_008.nii.gz
在进行vbm分析之前,根据被试间统计模型(cross-suject statistical model) , 生成design.mat和design.con.
在FSL提供的数据中, 包含了这两个文件, 在seg_struc/vbm/stats
design.mat中是:
/NumWaves 2
/NumPoints 16
/PPheights 1.000000e+00 1.000000e+00
/Matrix
1.000000e+00 0.000000e+00
1.000000e+00 0.000000e+00
1.000000e+00 0.000000e+00
1.000000e+00 0.000000e+00
1.000000e+00 0.000000e+00
1.000000e+00 0.000000e+00
1.000000e+00 0.000000e+00
1.000000e+00 0.000000e+00
0.000000e+00 1.000000e+00
0.000000e+00 1.000000e+00
0.000000e+00 1.000000e+00
0.000000e+00 1.000000e+00
0.000000e+00 1.000000e+00
0.000000e+00 1.000000e+00
0.000000e+00 1.000000e+00
0.000000e+00 1.000000e+00
design.mat中数据排列应该和ls命令得到的文件名排列一样, 前面的6个[1.0 0.0 ]代表的是con开头的对照组, 后面8个[0,1]对应的是pat开头的病人组.
2 提取大脑影像
下面用fslvbm_1_bet命令提取大脑组织, 并做线性配准. 参数-b代表采用标准bet命令, -N代表采用鲁棒性更好的大脑组织提取方式(bet+standard_space_roi)命令.
fslvbm_1_bet -b 或者
fslvbm_1_bet -N fslvbm_1_bet命令的输入输出如图:

运行完后, 可以得到快视的html. 在控制台中输入firefox ./slicesdir/index.html &就可以查看处理结果了.
3 生成study-specific模板
下面就可以生成刚才提到的study-specific template. 首先所有提取后的脑图像应该进行segmentation, 分割成灰质\白质和脑脊液(GM, WM, CSF) . 之后template_list上的灰质图像经过仿射变换对齐到GM ICBM-152模板上, 并进行合并和平均. 平均后的图像之后对x轴坐标去负值, 翻转得到一个镜像文件, 然后原图像和镜像图像再做一次平均得到first-pass, study-specific 灰质仿射模板( template_GM_init 文件). 之后, template_list上的灰质图像再非线性配准到这个生成的灰质仿射模板上, 然后组合成4D图像(“template_4D_GM”文件) , 之后再做平均, 平均之后再次在x轴翻转, 得到镜像图像, 最后, 平均图像和镜像图像再次做平均, 得到一个在标准坐标系下左右对称的study-specific 非线性灰质模板.(图像精度是 2∗2∗2mm3 2 ∗ 2 ∗ 2 m m 3 )
fslvbm_2_template -a或者
fslvbm_2_template -n参数-a的含义是, -n的含义是
fslvbm_2_template命令的输入输出关系如下图:
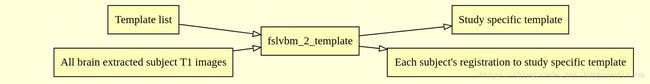
运行后, 还是可以在struc文件夹下查看快视图像. 运行后shell中得到如下输出:
con_1623_struc
…
pat_2662_struc
pat_2996_struc
Running segmentation: ID=19929
Running initial registration: ID=21814
Creating first-pass template: ID=23673
Running registration to first-pass template: ID=24090
Creating second-pass template: ID=27369
Study-specific template will be created, when complete, check results with:
fslview struc/template_4D_GM
and turn on the movie loop to check all subjects, then run:
fslview /home/dl/Software/FSL/data/standard/tissuepriors/avg152T1_gray struc/template_GM
to check general alignment of mean GM template vs. original standard space template.从输出的这些信息中, 可以得到这一步的执行过程:
1) 先进行segmentation, 分离灰质\白质和脑脊液
2) 之后进行第一遍对齐(registration), 用每个被试切割后的灰质和标准灰质模板得到的结果进行对齐. 对齐后得到的结果, 排成一列求平均, 得到的均值灰质图像 template_GM. 再将 template_GM的x坐标值取反, 也就相当于沿着y-z坐标轴平面做镜像, 得到图像 template_GM_flipped. 将 template_GM和 template_GM_flipped求平均, 从而得到左右脑对称的图像: template_GM_init. 至此完成了first-pass.
3) 将得到的subject-specific模板template_GM_init作为新的模板, 将每个被试的灰质图像对齐到新模板上, 对结果进行上一步相同的操作, 得到最终的subject-specific灰质模板.
将提示中的图像打开, 将study-specific模板和标准模板比较
fsleyes /home/dl/Software/FSL/data/standard/tissuepriors/avg152T1_gray struc/template_GM下图是生成的study-specific模板:
未完待续…
参考:
基于体素的形态学分析及结构网络对埃森克人格的研究, 卢凤梅, 电子科技大学, 2012
FSL帮助: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FSLVBM
MR影像体素形态学的阿尔茨海默病自动分类方法, 郭圣文, 东南大学学报
