gbdt 算法比随机森林容易_机器学习(集成算法1:随机森林分类器-红酒数据集案例、随机森林回归器-波士顿数据集填充缺失值案例)...
UCI机器学习练习数据集网站: http:// archive.ics.uci.edu/ml/ datasets/Skin+Segmentation
竞赛中备青睐的算法:随机森林、梯度提升树(GBDT)、XGBoost等集成树
集成算法应用领域:市场营销建模/统计客户来源、保留和流失/预测疾病的风险和病患者的易感性
一、知识点聚焦
1 、bagging: 随机森林分类器/随机森林回归器
【bagging使用的两个必要条件:基分类器尽量独立!弱分类器的预测准确准确率要>50% !】
>核心思想:并行建立n个模型,通过一定策略组合合成一个强分类器(基分类器为决策树,[相关剪枝参数])
>核心参数:
- n_estimators: 基分类器的个数;
- random_state: 空值森林生成的模式(注意:并不能控制森林中的每一颗树的生成模式)
- booststrap: 有放回的随机抽样。默认为True(通常不会改变这个参数)
- oob_score: 袋外数据可以作为测试样本,测试模型的效果(设置为True时)
>重要属性和接口
- .estimators_:查看森林中每一颗树的情况
- .oob_score_: 袋外数据的预测准确率
- .feature_importance_: 特征重要性
>常用接口
- score : 测试集的预测准确率
- apply:样本在森林中的叶子节点的索引
- predict:样本预测结果
- fit:训练模型的接口
- predict_proba: 样本被预测为不同类别的概率(随机分类器)
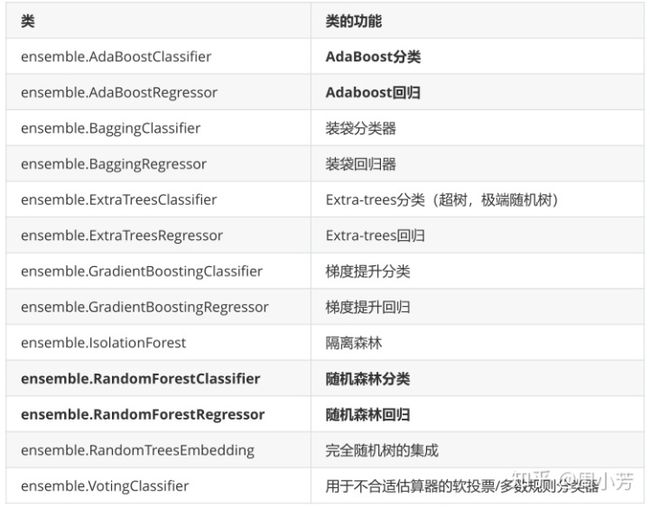
3、集成算法都包含哪些算法呢?
二、随机森林分类器(红酒数据集案例)
随机森林是在多个决策树基分类器基础上构建的,自然模型效果要比决策树更好,当然模型越复杂意味着计算效率越低。决策树VS随机森林
#导包
import matplotlib.pyplot as plt
%matplotlib inline
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.datasets import load_wine
from sklearn.model_selection import train_test_split
#导数据集,切分数据集
wine = load_wine()
wine.data
wine.target
wine.feature_names
Xtrain, Xtest, Ytrain, Ytest = train_test_split(wine.data,wine.target,test_size=0.3)
Xtrain.shape
#对比随机森林分类器vs决策树
clf = DecisionTreeClassifier(random_state = 0)
rfc = RandomForestClassifier(random_state = 0)
clf = clf.fit(Xtrain,Ytrain)
rfc = rfc.fit(Xtrain,Ytrain)
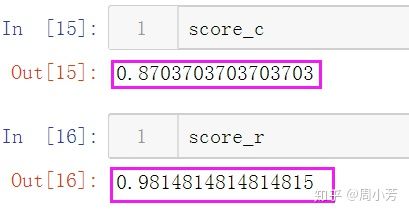
score_c = clf.score(Xtest,Ytest)
score_r = rfc.score(Xtest,Ytest)
score_c
score_r一组样本可能说服力不大,下面看看交叉验证的结果吧~
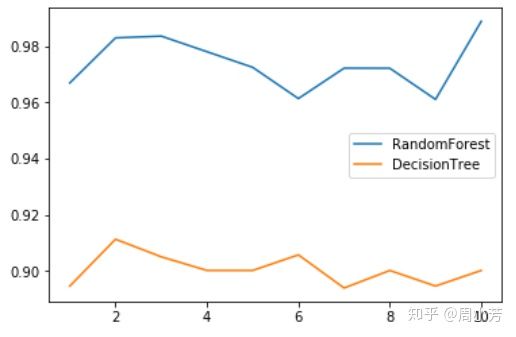
#交叉验证
from sklearn.model_selection import cross_val_score
label = "RandomForest"
for model in [RandomForestClassifier(n_estimators=25),DecisionTreeClassifier()]:
score = cross_val_score(model,wine.data,wine.target,cv=10)
# print("{}:".format(label)),print(score.mean())
print(f"{label}"),print(score.mean())
plt.plot(range(1,11),score,label = label)
plt.legend()
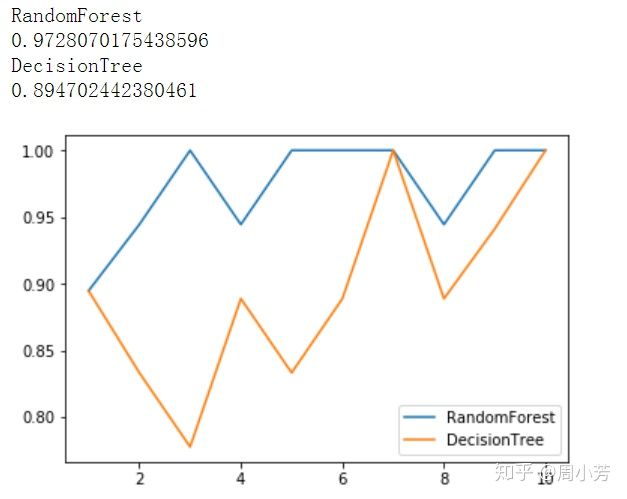
label = "DecisionTree"为了观察更稳定的结果,下面进行十组交叉验证
(单个决策树的准确率越高,随机森林的准确率也越高)
#十组交叉验证
rfc_l = []
clf_l = []
for i in range(10):
rfc = RandomForestClassifier(n_estimators=25)
rfc_s = cross_val_score(rfc,wine.data,wine.target,cv=10).mean()
rfc_l.append(rfc_s)
clf = DecisionTreeClassifier()
clf_s = cross_val_score(clf,wine.data,wine.target,cv=10).mean()
clf_l.append(clf_s)
#绘制结果曲线
plt.plot(range(1,11),rfc_l,label = "RandomForest")
plt.plot(range(1,11),clf_l,label = "DecisionTree")
plt.legend()
plt.show()n_estimators的学习曲线
#绘制n_estimators的学习曲线
superpa = []
for i in range(200):
rfc = RandomForestClassifier(n_estimators=i+1)
rfc_s = cross_val_score(rfc,wine.data,wine.target,cv=10).mean()
superpa.append(rfc_s)
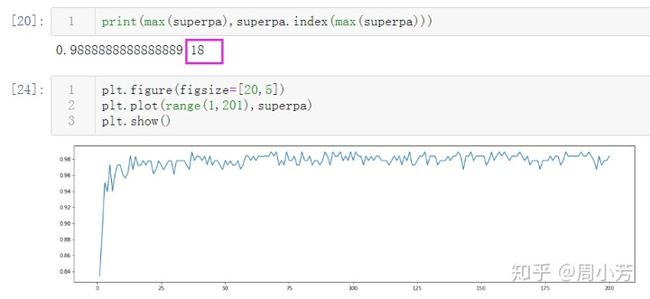
print(max(superpa),superpa.index(max(superpa)))
plt.figure(figsize=[20,5])
plt.plot(range(1,201),superpa)
plt.show()思考:随机森林用了神马方法,来保证集成的效果一定好于单个分类器?
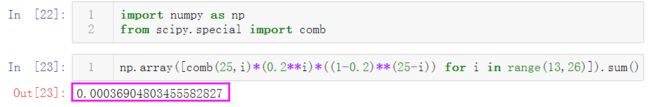
答:随机森林的本质是一种袋装算法,袋装集成算法是对基评估器的预测结果进行平均或者多数表决原则来决定集成评估器的结果。假设建立了25颗树的森林,只有当13颗以上的树判断错误时,随机森林才会判错。假如一颗树的准确率在0.85上下浮动,判错的可能性为0.25,那么13颗树判断错误的可能性为:
排列组合问题结果是比决策树小的,这也就说明随机森林判断错误的概率要比决策树小。
random_state
random_state固定时,随机森林得到一组固定的树,但每颗树是不一致的,这里的随机性是指:"随机挑选特征进行分支"。用袋装法集成时,基分类器应当是相互独立的,是不相同的。
#随机森林中的random_state控制的是生成森林的模式
rfc = RandomForestClassifier(n_estimators=20,random_state = 2)
rfc = rfc.fit(Xtrain, Ytrain)
#随机森林的重要属性之一:estimators,查看森林中树的状况
rfc.estimators_[2].random_state
#打印出森林中所有树的随机模式
for i in range(len(rfc.estimators_)):
print(rfc.estimators_[i].random_state)设置oob_score参数为True,使用袋外数据来做测试集
#无需划分训练集和测试集
rfc = RandomForestClassifier(n_estimators=25,oob_score=True)
rfc = rfc.fit(wine.data,wine.target)
#重要属性oob_score_
rfc.oob_score_重要属性和接口
#大家可以分别去尝试一下这些属性和接口
rfc = RandomForestClassifier(n_estimators=25)
rfc = rfc.fit(Xtrain, Ytrain)
rfc.score(Xtest,Ytest)
rfc.feature_importances_ #接口 特征重要性
rfc.apply(Xtest) #接口 返回预测结果在叶子节点的索引
rfc.predict(Xtest) #接口 返回预测值
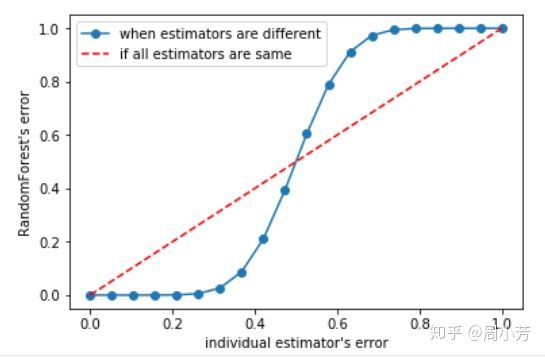
rfc.predict_proba(Xtest) #接口 返回预测概率决策树的预测错误率和随机森林预测错误率的关系(运行下面代码。可进行图形输出)
import numpy as np
x = np.linspace(0,1,20)
y = []
for epsilon in np.linspace(0,1,20):
E = np.array([comb(25,i)*(epsilon**i)*((1-epsilon)**(25-i))
for i in range(13,26)]).sum()
y.append(E)
#绘制图形
plt.plot(x,y,"o-",label="when estimators are different")
plt.plot(x,x,"--",color="red",label="if all estimators are same")
plt.xlabel("individual estimator's error")
plt.ylabel("RandomForest's error")
plt.legend()
plt.show()三、随机森林回归器 (填充缺失值案例-波士顿数据集)
首先说明的两点是:
- 所有的参数,属性和接口,基本与随机森林分类器一致,仅有的不同就是回归树与分类树不同,不纯度的指标,参数Criterion不一致
- 随机森林回归没有predict_proba这个借口,因为对于回归来说,并不存在一个样本要被分到某个类别的概率问题
在回归树中,我们追求的是MSE越小越好,MSE是:
- 分枝质量衡量指标
- 最常用的回归树回归质量的衡量指标
- 交叉验证时,或者用其他方式获取回归结果时,往往选择均方误差作为我们的评估(默认是R方)
然而,回归树的接口score返回的是R方,并不是MSE
填补缺失值(波士顿数据集)
sklearn.impute.SimpleImputer来轻松地将均值,中值,或者其他最常用数值填补到数据中
#导包
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import load_boston
from sklearn.impute import SimpleImputer # 填补缺失值,要求aklearn是20以上版本才行
from sklearn.model_selection import cross_val_score #交叉验证
from sklearn.ensemble import RandomForestRegressor #随机森林回归器
#查看sklearn版本
import sklearn
sklearn.__version__
#简单的进行随机森林回归,进行交叉验证
boston = load_boston()
regressor = RandomForestRegressor(n_estimators=100,random_state=0)
cross_val_score(regressor, boston.data, boston.target, cv=10,scoring = "neg_mean_squared_error")
#填补缺失值案例,原数据的特征和标签
dataset = load_boston()
dataset.data.shape
#总共506*13=6578个数据
X_full, y_full = dataset.data, dataset.target
n_samples = X_full.shape[0]
n_features = X_full.shape[1]
#查看每一列的缺失值个数
pd.DataFrame(dataset.data).isnull().sum()由于数据没有缺失值,我们现在需要创造一些缺失值出来
#首先确定我们希望放入的缺失数据的比例,在这里我们假设是50%,那总共就要有3289个数据缺失
rng = np.random.RandomState(0)
missing_rate = 0.5 #缺失值比率
n_missing_samples = int(np.floor(n_samples * n_features * missing_rate)) #缺失值个数
n_missing_samples #np.floor向下取整,返回.0格式的浮点数
missing_features = rng.randint(0,n_features,n_missing_samples) #随机选取列索引
missing_features
missing_samples = rng.randint(0,n_samples,n_missing_samples) #随机选取行索引
missing_samples
X_missing = X_full.copy() #复制一份,以免破坏原始数据集
y_missing = y_full.copy()
X_missing[missing_samples,missing_features] = np.nan #填充为NAN
X_missing = pd.DataFrame(X_missing) #DataFrame查看
#确认没有全部行都为缺失值
X_missing.head()
#使用均值进行填补
from sklearn.impute import SimpleImputer #导包
imp_mean = SimpleImputer(missing_values=np.nan, strategy='mean') #实例化
X_missing_mean = imp_mean.fit_transform(X_missing) # fit_transform
pd.DataFrame(X_missing_mean).head() #DataFrame查看均值填充后的数据
#使用0进行填补
imp_0 = SimpleImputer(missing_values=np.nan, strategy="constant",fill_value=0) #实例化
X_missing_0 = imp_0.fit_transform(X_missing) # fit_transform
pd.DataFrame(X_missing_0).head() #DataFrame查看0填充后的数据利用随机森林回归器对缺失值进行填充,思路(仅填充一列作为示范):
- 选出缺失值最多的一列,作为要预测的y
- 拼接剩余的X和旧的y作为新的x数据集
- 根据y是否是缺失值,将数据不缺失部分作为训练集,有缺失的部分作为测试集
- 训练模型,将测试集的预测结果填充到测试集y的缺失值中
X_missing_reg = X_missing.copy() #拷贝原数据集
#返回缺失值由多到少依次排序的索引
sortindex = np.argsort(X_missing_reg.isnull().sum(axis=0)).values
for i in sortindex:
# 构建我们的新特征矩阵和新标签
df = X_missing_reg #原始数据
fillc = df.iloc[:,i] # 取出新“y”
#将抽取了新“y”后的剩余X数据集和旧的y合并,组成新特征矩阵
df = pd.concat([df.iloc[:,df.columns != i],pd.DataFrame(y_full)],axis=1)
#在新特征矩阵中,对含有缺失值的列,进行0的填补
df_0 =SimpleImputer(missing_values=np.nan,
strategy='constant',fill_value=0).fit_transform(df)
#找出我们的训练集和测试集
Ytrain = fillc[fillc.notnull()]
Ytest = fillc[fillc.isnull()]
Xtrain = df_0[Ytrain.index,:]
Xtest = df_0[Ytest.index,:]
#用随机森林回归来填补缺失值
rfc = RandomForestRegressor(n_estimators=100)
rfc = rfc.fit(Xtrain, Ytrain)
Ypredict = rfc.predict(Xtest)
#将填补好的特征返回到我们的原始的特征矩阵中
X_missing_reg.loc[X_missing_reg.iloc[:,i].isnull(),i] = Ypredict对所有数据进行建模,观察效果
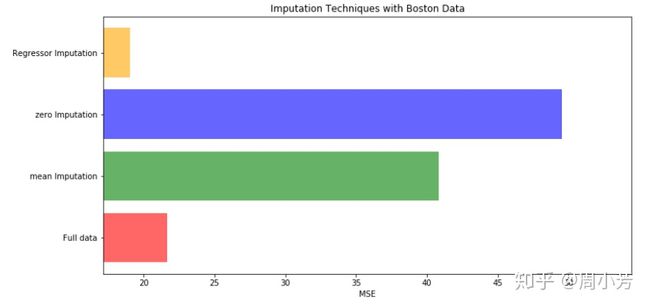
#对所有数据进行建模,取得MSE结果
X = [X_full,X_missing_mean,X_missing_0,X_missing_reg]
mse = []
std = []
for x in X:
estimator = RandomForestRegressor(random_state=0, n_estimators=100)
scores =cross_val_score(estimator,x,y_full,scoring='neg_mean_squared_error',cv=5).mean()
mse.append(scores * -1)
#画图
x_labels = ['Full data','mean Imputation','zero Imputation','Regressor Imputation']
colors = ['r', 'g', 'b', 'orange']
plt.figure(figsize=(12, 6))
ax = plt.subplot(111)
for i in np.arange(len(mse)):
ax.barh(i, mse[i],color=colors[i],alpha = 0.6, align='center')
ax.set_title('Imputation Techniques with Boston Data')
ax.set_xlim(left=np.min(mse) * 0.9,right=np.max(mse) * 1.1)
ax.set_yticks(np.arange(len(mse))) #设置y轴的刻度
ax.set_xlabel('MSE') #设置x的标签
ax.set_yticklabels(x_labels)
plt.show()回归器在捕捉的时候,消除了原数据的一些噪音,所以回归结果比原数据都要好!