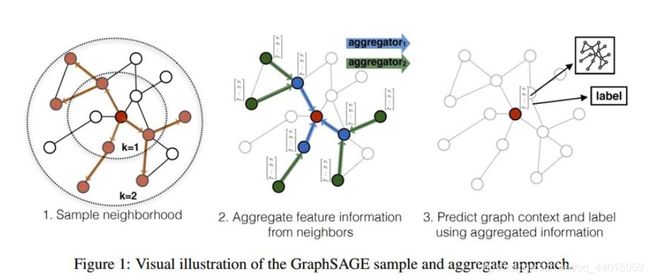
论文笔记: Inductive Representation Learning on Large Graphs
前言
PATCHY-SAN方法是将图结构的数据,通过一些列的取样和邻居节点的选择,将图结构的数据转化为序列结构的数据。将在欧氏空间表现良好的卷积方法作用于变形后的据图结构数据
此篇文章提出GraphSage方法旨在找出适用于图结构类型数据的卷积方法,也就是如何在图结构类型的数据上进行类似于卷积的操作。与结合图中全部节点进行权重更新的MPNN不同。区别于传统的全图卷积,GraphSage利用采样的部分节点的方式进行学习,根据采样的部分节点聚合一定数目的其邻居节点进行结点更新。
概括
在此文章之前前人的方法本质上是transductive,因为在学习过程中图中所有的顶点都参与进来,不能自然地泛化到未见过的顶点。从而提出了一个inductive的GraphSAGE算法。GraphSAGE同时利用节点特征信息和结构信息得到Graph Embedding的映射,相比之前的方法,之前都是保存了映射后的结果,而GraphSAGE保存了生成embedding的映射,可扩展性更强,对于节点分类和链接预测问题的表现也比较突出。也就是说GraphSAGE是为了学习一种节点表示方法,即如何通过从一个顶点的局部邻居采样并聚合顶点特征,而不是为每个顶点训练单独的embedding。
要点
-
传统transductive方法的局限性和GraphSAGE的优势:
最初的transductive方法虽然在某些任务上表现不错,但是在现实世界中固定的图较少同时要求快速地对未见过的结点进行嵌入。但是由于之前是将图中所有结点用于训练导致模型的泛化能力较差。但是对于GraphSAGE这种inductive的方法,是通过训练学习一种基于所选择节点邻居节点进行特征提取的模型。可以快速高效的预测未见过的结点。同时对于具有相似结构特点的模型可以快速高效的进行泛化。
GraphSAGE方法具有良好的泛化能力。例如,可以在源自模型生物的蛋白质-蛋白质相互作用图上训练嵌入生成器,然后使用经过训练的模型轻松生成节点嵌入,以收集在新生物上收集的数据。 -
transductive learning得到新节点的表示的难处:
要想得到新节点的表示,需要让新的graph或者subgraph去和已经优化好的node embedding去“对齐(align)”。然而每个节点的表示都是受到其他节点的影响,因此添加一个节点,意味着许许多多与之相关的节点的表示都应该调整。这会带来极大的计算开销,即使增加几个节点,也要完全重新训练所有的节点。 -
GraphSAGE基本思路:
既然新增的节点,一定会改变原有节点的表示,那么为什么一定要得到每个节点的一个固定的表示呢?何不直接学习一种节点的表示方法。去学习一个节点的信息是怎么通过其邻居节点的特征聚合而来的。 学习到了这样的“聚合函数”,而我们本身就已知各个节点的特征和邻居关系,我们就可以很方便地得到一个新节点的表示了。
GCN等transductive的方法,学到的是每个节点的一个唯一确定的embedding; 而GraphSAGE方法学到的node embedding,是根据node的邻居关系的变化而变化的,也就是说,即使是旧的node,如果建立了一些新的link,那么其对应的embedding也会变化,而且也很方便地学到。
同时该方法还可以利用所有图形中都存在的结构特征(例如,节点度)。因此,该算法也可以应用于没有节点特征的图。 -
GraphSAGE的训练结果:
GraphSAGE的核心:GraphSAGE不是试图学习一个图上所有node的embedding,而是学习一个为每个node产生embedding的映射。
算法训练了一组聚合函数学会从节点的本地邻域聚合特征信息。而不是为每个节点训练不同的嵌入向量。每个聚合函数从一个顶点的不同的hops或者说不同的搜索深度聚合信息。测试或是推断的时候,使用训练好的系统,通过学习到的聚合函数来对完全未见过的顶点生成embedding。
算法流程
1.Embedding generationalgorithm (生成节点embedding的前向传播算法)
GraphSAGE的前向传播算法如下,前向传播描述了如何使用聚合函数对节点的邻居信息进行聚合,从而生成节点embedding:
伪代码中存在一个问题就是第四行聚合后应该得到的是 k − 1 k-1 k−1层的邻居结点的特征表示。进而第五行也是 k − 1 k-1 k−1层的邻居结点的特征表示
 在每次迭代的过程中,顶点从它们的局部邻居聚合信息,并且随着这个过程的迭代,顶点会从越来越远的地方获得信息
在每次迭代的过程中,顶点从它们的局部邻居聚合信息,并且随着这个过程的迭代,顶点会从越来越远的地方获得信息
算法描述了在整个图上生成embedding的过程,其中
- G = ( V , E ) G=(V,E) G=(V,E)表示一个图
- K K K是网络的层数,也代表着每个顶点能够聚合的邻接点的层数,每增加一层,可以聚合更远的一层邻居的信息
- x v , ∀ v ∈ V x_v,∀v∈V xv,∀v∈V表示结点 v v v的特征向量作为输入(注意这里是全部的结点)
- h u k − 1 , ∀ u ∈ N ( v ) {h_u^{k−1} ,∀u∈N(v)} huk−1,∀u∈N(v)表示在 k − 1 k-1 k−1层中结点 v v v的邻居结点 u u u的embedding
- h N ( v ) k h_{N(v)}^k hN(v)k表示在第 k k k层,结点 v v v的所有邻居节点的特征表示
- h v k , ∀ v ∈ V h_v^k,∀v∈V hvk,∀v∈V表示在第 k k k层,结点 v v v的特征表示
- N ( v ) N(v) N(v)定义为从集合 u ∈ v : ( u , V ) ∈ E u ∈ v : ( u , V ) ∈ E u ∈ v : ( u , V ) ∈ E {u∈v:(u,V)∈Eu \in v: (u, \mathcal{V}) \in \mathcal{E}u∈v:(u,V)∈E} u∈v:(u,V)∈Eu∈v:(u,V)∈Eu∈v:(u,V)∈E中的固定size的均匀取出
即GraphSAGE中每一层的节点邻居都是是从上一层网络采样的,并不是所有邻居参与,并且采样的后的邻居的size是固定的
在解释完符号信息,具体看一下算法的流程是什么:
Neighborhood definition - 采样邻居顶点
出于对计算效率的考虑,对每个顶点采样一定数量的邻居顶点作为待聚合信息的顶点。设需要的邻居数量,即采样数量为 S S S,若顶点邻居数少于 S S S,则采用有放回的抽样方法,直到采样出 S S S个顶点。若顶点邻居数大于 S S S,则采用无放回的抽样。(即采用有放回的重采样/负采样方法达到 S S S)
当然,若不考虑计算效率,完全可以对每个顶点利用其所有的邻居顶点进行信息聚合,这样是信息无损的。
统一采样一个固定大小的邻域集,以保持每个batch的计算占用空间是固定的(即 graphSAGE并不是使用全部的相邻节点,而是做了固定size的采样)。
这样固定size的采样,每个节点和采样后的邻居的个数都相同,可以把每个节点和它们的邻居拼成一个batch送到GPU中进行批训练。
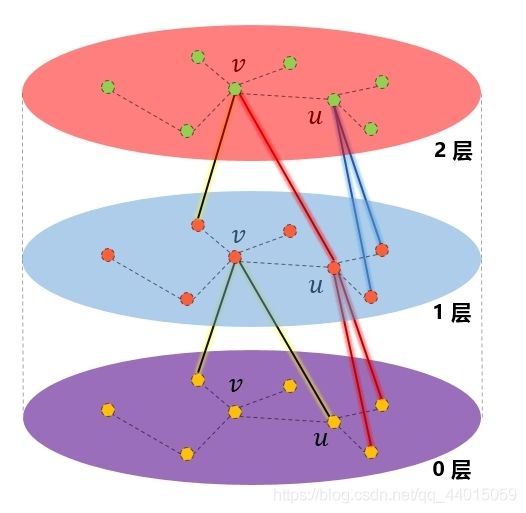
- 算法中 k k k从1开始循环,因此看第一层。 v v v是我们选取的顶点,在这里我们设置采样邻居节点的数量为2和采样邻居的阶数也为2.根据算法流程,我们可以获得第一层的 h N ( v ) 0 h_{N(v)}^0 hN(v)0(由第0层随机选取的顶点 v v v的两个邻居获得)。与此同时 v v v的邻居 u u u根据分布特征也聚合了第0层的两个邻居信息
- 因此我们可以分别获取第一层中 v v v和 u u u的特征信息(由第0层 v v v和 u u u的两个邻居特征信息和第0层的 v v v和 u u u的特征信息之和所得到)
实际上我们可以看出第 k k k层目标顶点的特征向量就是由第 k − 1 k-1 k−1层的目标顶点和邻居顶点的特征向拼接后经过某种操作生成的 - 到了第2层,可以看到节点 v v v通过“1层”的节点u和另一邻居得到了第1层 v v v的邻居节点信息,其中借助 u u u扩展到了“0层”的二阶邻居节点。因此,在聚合时,聚合K次,就可以扩展到K阶邻居。
第二层仍然满足我们总结的规律,只是在此时 k − 1 k-1 k−1层的邻居顶点包含了 k − 2 k-2 k−2层的邻居顶点,也就达到了扩展到 k k k阶邻居的目的 - 实验发现,K不必取很大的值,当K=2时,效果就很好了。至于邻居的个数,文中提到 S 1 ⋅ S 2 ≤ 500 S1⋅S2≤500 S1⋅S2≤500即两次扩展的邻居数之际小于500,大约每次只需要扩展20来个邻居时获得较高的性能。
- 论文里说固定长度的随机游走其实就是随机选择了固定数量的邻居
2聚合函数的选取
在图中顶点的邻居是无序的,所以希望构造出的聚合函数是对称的(即也就是对它输入的各种排列,函数的输出结果不变),同时具有较高的表达能力。 聚合函数的对称性(symmetry property)确保了神经网络模型可以被训练且可以应用于任意顺序的顶点邻居特征集合上。
Mean aggregator
mean aggregator将目标顶点和邻居顶点的第 k − 1 k−1 k−1层向量拼接起来,然后对向量的每个维度进行求均值的操作,将得到的结果做一次非线性变换产生目标顶点的第 k k k层表示向量。
文中用下面的式子替换算法1中的4行和5行得到GCN的inductive变形
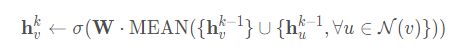 原始第4,5行是
原始第4,5行是
这里要注意的是伪代码中存在一个问题就是第四行聚合后应该得到的是 k − 1 k-1 k−1层的邻居结点的特征表示。进而第五行也是 k − 1 k-1 k−1层的邻居结点的特征表示
 修改后的基于均值的聚合器是convolutional的,这个卷积聚合器和文中的其他聚合器的重要不同在于它没有算法1中第5行的CONCAT操作可以看到替换后,是对 h v k − 1 h^{k−1}_v hvk−1和集合 { h u k − 1 , ∀ u ∈ N ( v ) } \left\{\mathbf{h}_{u}^{k-1}, \forall u \in \mathcal{N}(v)\right\} { huk−1,∀u∈N(v)}取并集,然后一起算均值,再乘上权重
修改后的基于均值的聚合器是convolutional的,这个卷积聚合器和文中的其他聚合器的重要不同在于它没有算法1中第5行的CONCAT操作可以看到替换后,是对 h v k − 1 h^{k−1}_v hvk−1和集合 { h u k − 1 , ∀ u ∈ N ( v ) } \left\{\mathbf{h}_{u}^{k-1}, \forall u \in \mathcal{N}(v)\right\} { huk−1,∀u∈N(v)}取并集,然后一起算均值,再乘上权重
LSTM aggregator
文中也测试了一个基于LSTM的复杂的聚合器[Long short-term memory]。和均值聚合器相比,LSTMs有更强的表达能力。但是,LSTMs不是symmetric的,也就是说不具有排列不变性(permutation invariant),因为它们以一个序列的方式处理输入。因此,需要先对邻居节点随机顺序,然后将邻居序列的embedding作为LSTM的输入。
Pooling aggregator
pooling聚合器,它既是对称的,又是可训练的。Pooling aggregator 先对目标顶点的邻居顶点的embedding向量进行一次非线性变换,之后进行一次pooling操作(max pooling or mean pooling),将得到结果与目标顶点的表示向量拼接,最后再经过一次非线性变换得到目标顶点的第k层表示向量。
一个element-wise max pooling操作应用在邻居集合上来聚合信息:
注意这里应该得到的也是 k − 1 k-1 k−1层的邻居结点的特征表示 h N ( v ) k − 1 h_{N(v)}^{k-1} hN(v)k−1
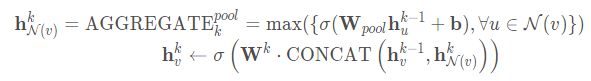
- max表示element-wise最大值操作,取每个特征的最大值
- σ σ σ是非线性激活函数
- 所有相邻节点的向量共享权重,先经过一个非线性全连接层,然后做max-pooling
- 按维度应用 max/mean pooling,可以捕获邻居集上在某一个维度的突出的/综合的表现。
3.Learning the parameters of GraphSAGE (有监督和无监督)参数学习
在定义好聚合函数之后,接下来就是对函数中的参数进行学习。文章分别介绍了无监督学习和监督学习两种方式。
基于图的无监督损失
无监督损失函数的设定来学习结点embedding可以供下游多个任务使用。监督学习形式根据任务的不同直接设置目标函数即可,如最常用的节点分类任务使用交叉熵损失函数
参数学习
通过前向传播得到节点 u u u的embedding z u z_u zu,然后梯度下降(实现使用Adam优化器) 进行反向传播优化参数 W k W^k Wk和聚合函数内的参数
新节点embedding的生成
这个 W k W^k Wk就是所谓的dynamic embedding的核心,因为保存下来了从节点原始的高维特征生成低维embedding的方式。现在,如果想得到一个点的embedding,只需要输入节点的特征向量,经过卷积(利用已经训练好的 W k W^k Wk 以及特定聚合函数聚合neighbor的属性信息),就产生了节点的embedding。
4实验
实验目的
- 比较GraphSAGE相比baseline算法的提升效果
- 比较GraphSAGE的不同聚合函数
数据集及任务
- Citation论文引用网络(节点分类)
- Reddit帖子论坛(节点分类)
- PPI蛋白质网络(graph分类)
baselines
- Random,随机分类器
- Raw features,手工特征(非图特征)
- deepwalk(图拓扑特征)
- DeepWalk + features,deepwalk + 手工特征
除此以外,还比较了GraphSAGE四个变种 ,并无监督生成embedding输入给LR和端到端有监督。因为,GraphSAGE的卷积变体是一种扩展形式,是Kipf et al. 半监督GCN的inductive版本,称这个变体为GraphSAGE-GCN。
分类器均采用LR
在所有这些实验中,预测在训练期间看不到的节点,在PPI数据集的情况下,实验在完全看不见的图上进行了测试。
实验设置
- K=2,聚合两跳内邻居特征
- S1=25,S2=10: 对一跳邻居抽样25个,二跳邻居抽样10个
- RELU 激活单元 Adam 优化器(除了DeepWalk,DeepWalk使用梯度下降效果更好)
- 文中所有的模型都是用TensorFlow实现
- 对每个节点进行步长为5的50次随机游走
- 负采样参考word2vec,按平滑degree进行,对每个节点采样20个
- 保证公平性:所有版本都采用相同的minibatch迭代器、损失函数、邻居采样器
- 实验测试了根据式1的损失函数训练的GraphSAGE的各种变体,还有在分类交叉熵损失上训练的可监督变体
- 对于Reddit和citation数据集,使用”online”的方式来训练DeepWalk
- 在多图情况下,不能使用DeepWalk,因为通过DeepWalk在不同不相交的图上运行后生成的embedding空间对它们彼此说可能是arbitrarily rotated的(见文中附录D)
4.1Inductive learning on evolving graphs: Citation and Reddit data 数据集介绍
前两个实验是在演化的信息图中对节点进行分类,这是一个与高吞吐量生产系统特别相关的任务,该系统经常遇到不可见的数据。
Citation data
第一个任务是在一个大的引文数据集中预测论文主题类别。文中使用来自汤姆森路透科学核心数据库(Thomson Reuters Web of Science Core Collection)的无向的引文图数据集(对应于2000-2005年六个生物相关领域的所有论文)。这个数据集的节点标签对应于六个不同的领域的标签。该数据集共包含302,424个节点,平均度数为9.15。文中使用2000-2004年的数据集对所有算法进行训练,并使用2005年的数据进行测试(30%用于验证)。对于特征,文中使用节点的度。此外,按照Arora等人的sentence embedding方法处理论文摘要(使用GenSim word2vec实现训练的300维单词向量)。
Reddit data
第二个任务预测不同的Reddit帖子(posts)属于哪个社区。Reddit是一个大型的在线论坛,用户可以在这里对不同主题社区的内容进行发布和评论。作者在Reddit上对2014年9月发布的帖子构建了一个图形数据集。本例中的节点标签是帖子所属的社区或“subreddit”。文中对50个大型社区进行了抽样,并构建了一个帖子-帖子的图,如果同一个用户评论了两个帖子,就将这两个帖子连接起来。该数据集共包含232,965个帖子,平均度为492。文中将前20天的用于训练,其余的用于测试(30%用于验证)。对于特征,文中使用现成的300维GloVe CommonCrawl词向量对于每一篇帖子,将下面的内容连接起来:
- 帖子标题的平均embedding
- 所有帖子评论的平均embedding
- 该帖子的得分
- 该帖子的评论数量
Generalizing across graphs: Protein-protein interactions
考虑跨图进行泛化的任务,这需要了解节点的角色,而不是社区结构。文中在各种蛋白质-蛋白质相互作用(PPI)图中对蛋白质角色进行分类,每个图对应一个不同的人体组织。并且使用从Molecular
Signatures Database中收集的位置基因集、motif基因集和免疫学signatures作为特征,gene ontology作为标签(共121个)。图中平均包含2373个节点,平均度为28.8。文中将所有算法在20个图上训练,然后在两个测试图上预测F1 socres(另外两个图用于验证)
4.2实验结果
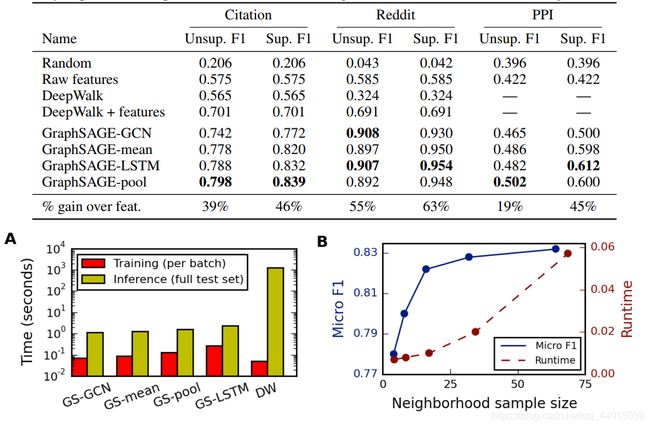
- 可以看到GraphSAGE的性能显著优于baseline方法
- 三个数据集上的实验结果表明,一般是LSTM或pooling效果比较好,有监督都比无监督好
- 无监督版本的GraphSAGE-pool对引文数据和Reddit数据的连接(concatenation)性能分别比DeepWalk embeddings和raw features的连接性能好13.8%和29.1%,而有监督版本的连接性能分别提高了19.7%和37.2%
- 尽管LSTM是为有序数据而不是无序集设计的,但是基于LSTM的聚合器显示了强大的性能
- 最后,可以看到无监督GraphSAGE的性能与完全监督的版本相比具有相当的竞争力,这表明文中的框架可以在不进行特定于任务的微调(task-specific fine-tuning)的情况下实现强大的性能
解读第二组图
- 计算时间:GraphSAGE中LSTM训练速度最慢,但相比DeepWalk,GraphSAGE在预测时间减少100-500倍(因为对于未知节点,DeepWalk要重新进行随机游走以及通过SGD学习embedding)
- 邻居采样数量:图B中邻居采样数量递增,F1也增大,但计算时间也变大。 为了平衡F1和计算时间,将S1设为25
- 聚合K跳内信息:在GraphSAGE, K=2 相比K=1有10-15%的提升;但将K设置超过2,效果上只有0-5%的提升,但是计算时间却变大了10-100倍
通过第二组图得到如下结论
- LSTM和pool的效果较好
- 为了更定量地了解这些趋势,实验中将设置六种不同的实验,即(3个数据集)×(非监督vs.监督))
- GraphSAGE-LSTM比GraphSAGE-pool慢得多(≈2×),这可能使基于pooling的聚合器在总体上略占优势
- LSTM方法和pooling方法之间没有显著差异
- 文中使用非参数Wilcoxon Signed-Rank检验来量化实验中不同聚合器之间的差异,在适用的情况下报告T-statistic和p-value。
5.总结
GraphSAGE的核心:GraphSAGE不是试图学习一个图上所有node的embedding,而是学习一个为每个node产生embedding的映射
改进方向:扩展GraphSAGE以合并有向图或者多模式图;探索非均匀邻居采样函数
为什么GCN是transductive,为什么要把所有节点放在一起训练?
不一定要把所有节点放在一起训练,一个个节点放进去训练也是可以的。无非是如果想得到所有节点的embedding,那么GCN可以把整个graph丢进去,直接得到embedding,还可以直接进行节点分类、边的预测等任务。
其实,通过GraphSAGE得到的节点的embedding,在增加了新的节点之后,旧的节点也需要更新,这个是无法避免的,因为,新增加点意味着环境变了,那之前的节点的表示自然也应该有所调整。只不过,对于老节点,可能新增一个节点对其影响微乎其微,所以可以暂且使用原来的embedding,但如果新增了很多,极大地改变的原有的graph结构,那么就只能全部更新一次了。从这个角度去想的话,似乎GraphSAGE也不是什么“神仙”方法,只不过生成新节点embedding的过程,实施起来相比于GCN更加灵活方便了。在学习到了各种的聚合函数之后,其实就不用去计算所有节点的embedding,而是需要去考察哪些节点,就现场去计算,这种方法的迁移能力也很强,在一个graph上学得了节点的聚合方法,到另一个新的类似的graph上就可以直接使用了。