前面我们讲过了单因素和多因素cox回归分析,那么怎么样将结果以森林图的形式来展示呢?
森林图简介
森林图(forest plot),从定义上讲,它一般是在平面直角坐标系中,以一条垂直于X轴的无效线(通常坐标X=1或0)为中心,用若干条平行于X轴的线段,来表示每个研究的效应量大小及其95%可信区间,并用一个棱形来表示多个研究合并的效应量及可信区间,它是Meta分析中最常用的结果综合表达形式,现在也广泛应用在biomarker此类研究中。
森林图的科研用途
提到森林图,很多人的第一反应就是Meta分析。实际上,除了Meta分析,森林图还有很多用处。森林图可以直观的反映出效应量(例如RR、OR、HR或者WMD)大小及其95% CI,这些效应量指标通常都是通过采用多因素回归分析所得,因此我们同样可以把森林图借鉴过来,用于展示单因素或者多因素回归分析的结果。总结来说,森林图的科研用途主要用于Meta和临床实验。
临床实验普通分析,常规森林图
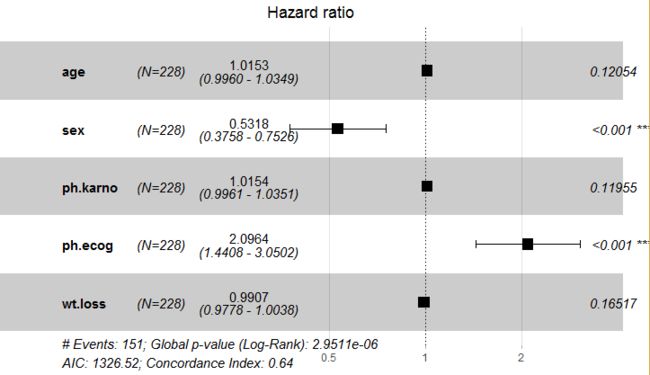
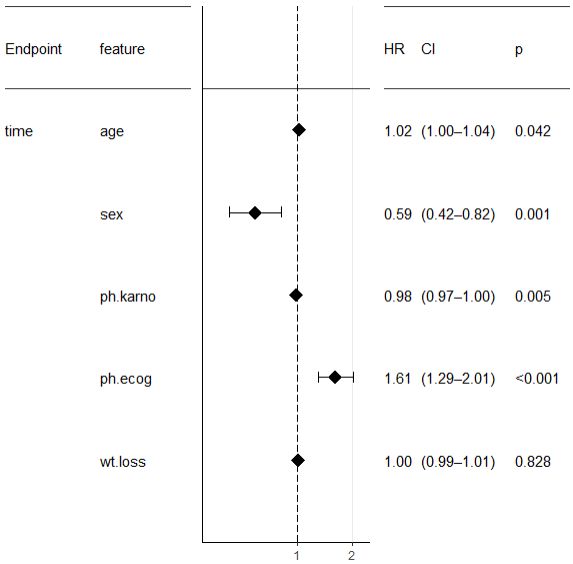
下图就是常规Cox回归结果的森林图展示,主要体现了变量、病人数量、P值和HR值。比如: ph.ecog变量位于无效线(即中间的那条竖线)右侧,说明ph.ecog有助于死亡。森林图在常规情况下事件结局是"生/死"这种两分类,但有时候事件结局是"有效/无效"、"治疗/未治疗"等等其他二分类情况,评估事件是好事还是坏事。比如生存(生:0;死:1),位于无效线左侧的变量,说明这些变量不利于事件发生,是保护因素;位于无效线右侧的变量,说明这些变量有助于事件发生,是危险因素;当与无效线相交时,说明这些变量与事件发生之间关系不强!在整体数据上,用来评估这些变量因素对事件结局的影响!
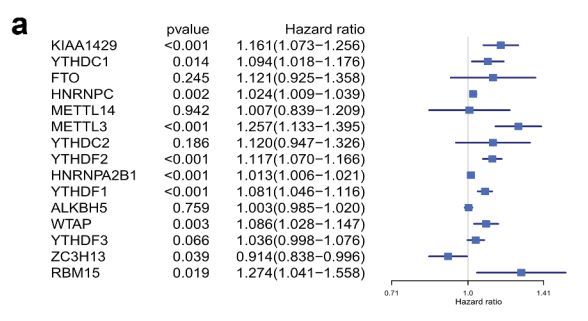
小编在下面这篇文章中
看到了如下的森林图,
今天小编就带大家一起来重现这张图,我们还是用单因素和多因素cox回归分析中提到的lung这套数据来举例。小编用三种不同的方法来实现这张图。
第一种,我们用最原始的plot函数,lines函数从底层来实现。后边两种方法,我们用现成的R包来实现。
#加载这两个R包
library("survival")
library("survminer")
#加载肺癌这套数据
data("lung")
###########################################
#批量单因素cox回归分析
############################################
#假设我们要对如下5个特征做单因素cox回归分析
covariates <- c("age", "sex", "ph.karno", "ph.ecog", "wt.loss")
#分别对每一个变量,构建生存分析的公式
univ_formulas <- sapply(covariates,
function(x) as.formula(paste('Surv(time, status)~', x)))
#对每一个特征做cox回归分析
univ_models <- lapply( univ_formulas, function(x){coxph(x, data = lung)})
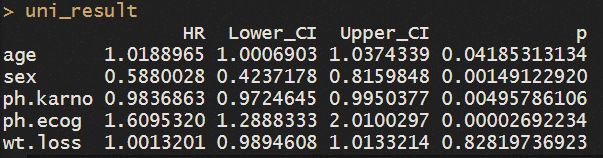
#提取HR,95%置信区间和p值
univ_results <- lapply(univ_models,
function(x){
x <- summary(x)
#获取p值
p.value<-signif(x$wald["pvalue"], digits=2)
#获取HR
HR <-signif(x$coef[2], digits=2);
#获取95%置信区间
HR.confint.lower <- signif(x$conf.int[,"lower .95"], 2)
HR.confint.upper <- signif(x$conf.int[,"upper .95"],2)
HR <- paste0(HR, " (",
HR.confint.lower, "-", HR.confint.upper, ")")
res<-c(p.value,HR)
names(res)<-c("p.value","HR (95% CI for HR)")
return(res)
})
#转换成数据框,并转置
res <- t(as.data.frame(univ_results, check.names = FALSE))
res <-as.data.frame(res,stringsAsFactors=F)
#############################################################
#对HR (95% CI for HR)做处理,得到HR和low .95和high .95
#当然也可以改计算univ_results这一步的代码,不要将HR和CI贴起来
############################################################
HR=gsub("[\\(\\)]","",res$`HR (95% CI for HR)`)
HR=gsub("-"," ",HR)
HR=as.data.frame(do.call(cbind,strsplit(HR," ")),stringsAsFactors=F)
names(HR)=rownames(res)
#################################
#开始绘图,直接保存到pdf文件中
#################################
pdf(file="univariate_forest.pdf",width=7)
#左边和右边边距稍微留多一点来写变量名称,pvalue和HR
par(mar=c(5,6,4,13))
#先用小方块画出HR
plot(as.numeric(HR[1,]),1:dim(HR)[2],
pch=15,cex=2,col="blue",bty='n',yaxt='n',ylab=NA,xlab="Hazard Ratio",
xlim=range(as.numeric(unlist(HR)))
)
#添加中线
abline(v=1,col="grey",lwd=2,lty=2)
for(i in 1:ncol(HR)){
x=as.numeric(HR[2:3,i])
#循环画出CI
lines(x,c(i,i),col="blue")
#添加变量名
text(0.2,i,rownames(res)[i],xpd=T,adj = c(0,0))
#添加p值
text(2.1,i,as.numeric(res[i,1]),xpd=T,adj = c(0,0))
#添加HR和CI
text(2.7,i,as.character(res[i,2]),xpd=T,adj = c(0,0))
}
#添加标题
text(2.1,ncol(HR)+0.5,"pvalue",xpd=T,adj = c(0,0))
text(2.7,ncol(HR)+0.5,"HR(CI)",xpd=T,adj = c(0,0))
dev.off()
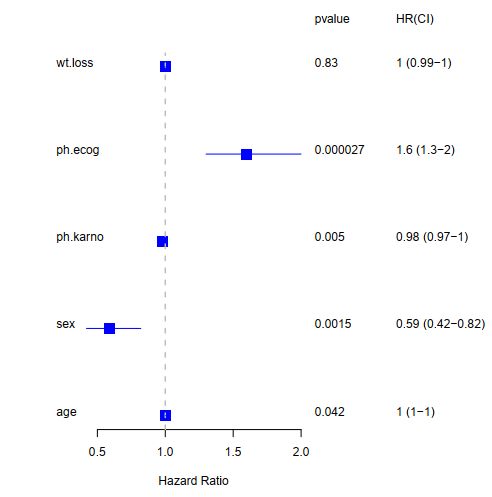
会得到下面这张图,是不是跟文章中的长的很像,这可是小编纯手工打造的。
多因素的森林图,如果你理解了上面单因素的绘图的思路和原理,应该也不难,大家可以自己练练手。
第二种方法,使用survivalAnalysis包来实现
这个包不仅可以画forest图,还可以计算cox回归的结果。
先来看单因素cox分析的结果和forest图
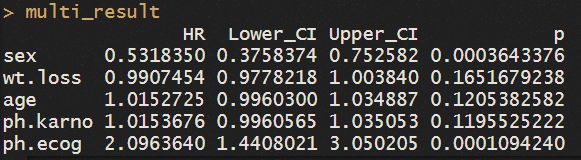
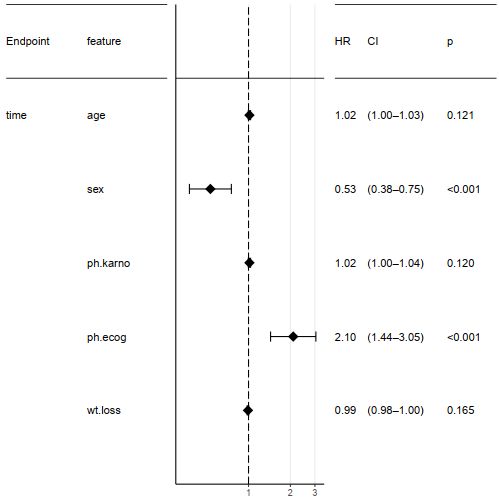
再来看看多因素cox分析的结果和forest图
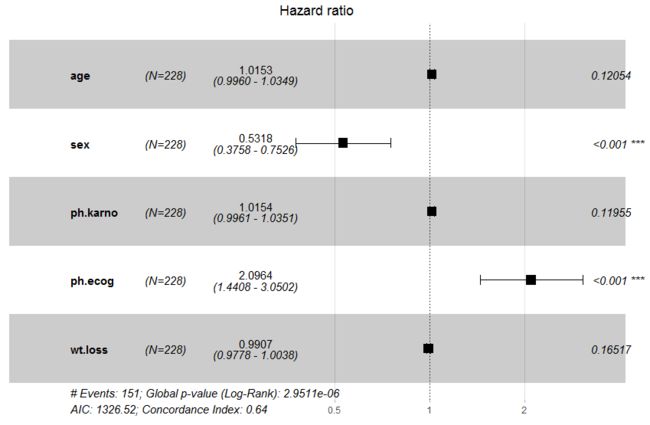
第三种方法,使用ggforest函数来实现
完整代码参考
R绘制森林图(forestplot),附代码