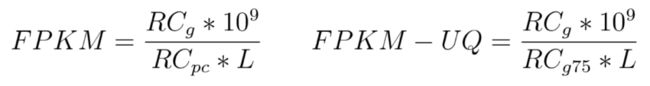箱线图怎么判断异常值_箱线图的生物学含义
我们阅读量破万的综述:RNA-seq这十年(3万字长文综述)
给粉丝朋友们带来了很多理解上的挑战,所以我们开辟专栏慢慢介绍其中的一些概念性的问题,上一期:
表达矩阵的归一化和标准化,去除极端值,异常值
描述数据,或者解读数据的时候,不能只关注其“集中性”和“离散性”指标(如均数、中位数、标准差、四分位数等),还得关注原始数据的分布形式。
箱线图是能同时反映数据统计量和整体分布,又很漂亮的展示图。
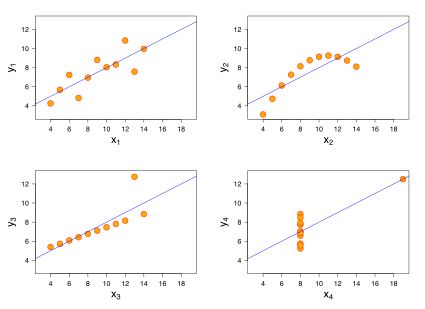
一、Anscome's Quartet
Anscombe的四重奏是四个数据集,这些数据集有着几乎相同的描述统计指标(均数,方差,相关性),但却有着区别明显的数据分布,如下图。
Anscome's Quartet
通过这些数据集的分布,使用 简单的汇总统计数据检查时,所有 四个集合都是 相同的,但在 绘制数据集分布时会有 很大差异。 “数值计算是准确的,可是图表太粗糙了”。不仅要关注数据的统计学指标,更要关注 异常值和其他对统计特性有影响的指标。
而数据分布是在数据分析中常常会被大多人所忽略的一步。 统计学模型都是基于统计分布提出的,如果仅凭一些描述统计学的指标来判断,会造成很大的误差,这时,了解 数据集的分布是很必要的。
二、描述统计与数据分布
datasauRus是Anscome's Quartet的一个进阶,包含有13个描述统计指标相同但分布差异极大的数据集。
通过R语言来探索一下datasauRus:
# 安装datasauRus的R扩展包
install.packages("datasauRus")
# 加载R包
library(datasauRus)
if(requireNamespace("dplyr")){
suppressPackageStartupMessages(library(dplyr))
datasaurus_dozen %>%
group_by(dataset) %>%
summarize(
mean_x = mean(x),
mean_y = mean(y),
std_dev_x = sd(x),
std_dev_y = sd(y),
corr_x_y = cor(x, y)
)
}
这13个数据集的描述统计指标完全一样。
# A tibble: 13 x 6
dataset mean_x mean_y std_dev_x std_dev_y corr_x_y
1 away 54.3 47.8 16.8 26.9 -0.0641
2 bullseye 54.3 47.8 16.8 26.9 -0.0686
3 circle 54.3 47.8 16.8 26.9 -0.0683
4 dino 54.3 47.8 16.8 26.9 -0.0645
5 dots 54.3 47.8 16.8 26.9 -0.0603
6 h_lines 54.3 47.8 16.8 26.9 -0.0617
7 high_lines 54.3 47.8 16.8 26.9 -0.0685
8 slant_down 54.3 47.8 16.8 26.9 -0.0690
9 slant_up 54.3 47.8 16.8 26.9 -0.0686
10 star 54.3 47.8 16.8 26.9 -0.0630
11 v_lines 54.3 47.8 16.8 26.9 -0.0694
12 wide_lines 54.3 47.8 16.8 26.9 -0.0666
13 x_shape 54.3 47.8 16.8 26.9 -0.0656
接下来使用ggplot2来查看这13个数据集的分布情况。
if(requireNamespace("ggplot2")){
library(ggplot2)
ggplot(datasaurus_dozen, aes(x=x, y=y, colour=dataset))+
geom_point()+
theme_void()+
theme(legend.position = "none")+
facet_wrap(~dataset, ncol=3)
}
这些分布和想象的差距巨大!!!
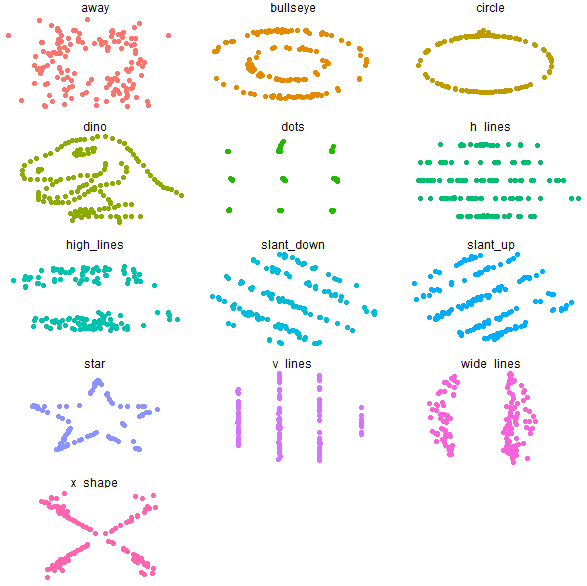 datasauRus的分布
datasauRus的分布
来个更震撼的动态图!!
数据分布动态变换图三、如何绘制箱线图
1.一些描述统计学概念
平均值,方差,最大值,最小值这些应该很熟悉,主要介绍一下中位数和四分位数的概念。
中位数:就是样本排序后,最中间的那个数了。
(将数据集从最大值一直排到最小值,从小到大也可以,那个最中间的数。如果样本数是奇数,那么中间的数只有一个就是中位数,如果样本数是偶数,那么取中间两个值的平均数就是他们的中位数)
四分位数:实际上就是把数据集分成四等份,要把一个苹果切成四块,需要几刀?答案是三。如果要把一个数据集分成四份,那么就需要3个指标来描述(描述性统计学):上四分位数,中位数,和下四分位数。
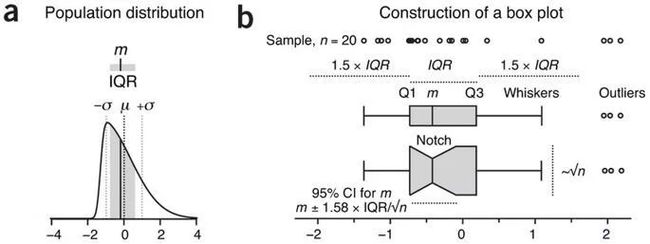
2.箱线图的组成
箱形图使用第25,50和75百分位数(也称为下四分位数(Q1),中位数(m或Q2)和上四分位数(Q3),以及四分位数范围(IQR = Q3-Q1,涵盖50%的中央数据)来反映样本的分布。四分位数不受异常值影响,并保留了中央数据和分布的信息。因此,对于不对称或不规则形状的种群分布以及具有极端异常值的样本,优于平均值和标准差。在这些不规则或异常分布下,平均值是偏离大部分数据的,标准差不适用来解释这类数据分布。
如下图,箱线图的核心是一个框,长度是IQR,宽度任意。框内的线表示中位数,不一定在中心。垂直或水平都可以,但水平时,样本的分布要按顺序。线延伸到最极端的数据点,有不超过不超过1.5×IQR的Tukey风格,也有一直延伸到最大值和最小值的Spear风格。
用四分位数绘制箱形图的是一个公认的惯例:永远不应使用箱子或线来显示平均值、标准差或标准误。中位数不一定在箱子中心,两边延伸的线也不一定是对称的。
1.5×IQR1.5乘数对应±2.7σ(其中σ是标准差),覆盖了99.3%的正态分布数据。
延伸线之外的异常值可以单独绘制。
箱形图构造需要至少n = 5(越多越好)的样本,尽管某些软件不检查这一点。对于n <5,建议显示所有数据点。
箱线图的组成
样本大小可以通过成比例的调整箱线图的宽度实现,如上图b中的第二个箱线图,箱子的凸凹程度表示样本量的多少。
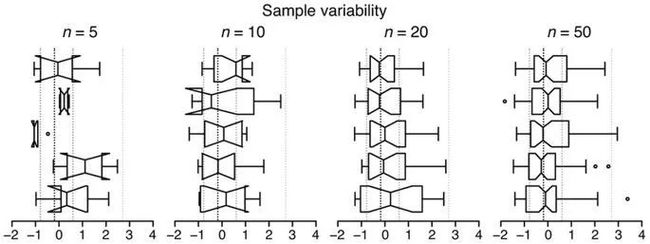
3.样本量对箱线图的影响
样本量越大,样本分布描述的准确性就越高。
不同样本量绘制箱线图
4.箱线图的边界的确定
箱线图的箱子边界的确切位置取决于软件。首先,没有一种普遍认可的方法来计算四分位数,可以通过取均值或线性插值计算。其次,一些软件如R使用铰链hinges而非四分位数来作为箱边界,下铰链和上铰链分别是数据下半部分和上半部分的中位值,这种箱线图与基于四分位数的箱线图略有不同。
箱线图的宽度,上下限的位置,凹口尺寸和异常值都需要调整,因此,在文章描述清箱线图的构造方式是非常重要的。
四、箱线图的优点与不足
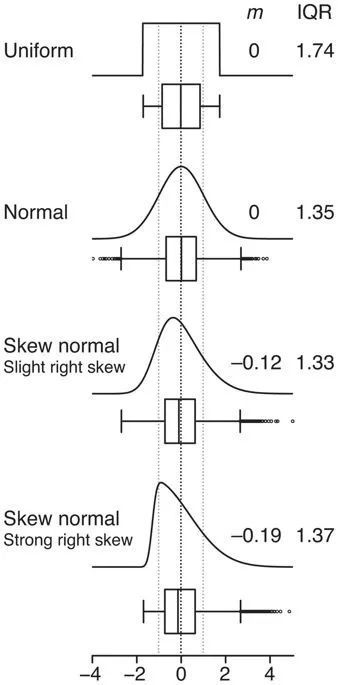
1.箱线图能直观展现样本的分布
从下图中可以看出箱线图的统计描述比均值和标准差更直观的展现了数据集的统计分布。
偏斜分布的箱线图
2.箱线图能展示更多的样本信息
箱线图提供一种更具沟通性的方式来展示样本数据的分布。
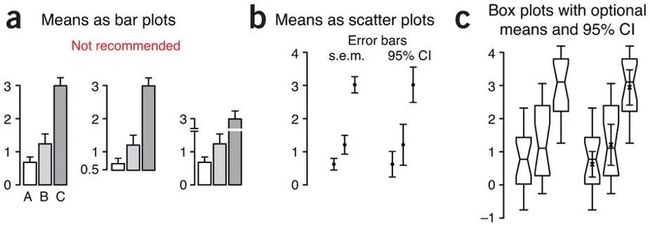
直方图、散点图和箱线图比较上图展示了三个样本量为20的正态分布数据,其中标准差都为1,AB均值为1,C均值为3。图a比较了选取不同基线切割y轴对直方图高度的影响;图b是当样本量大于3时,标准差和95%置信区间的散点图适合比较集中趋势的数据;图c的箱线图能同时结合均值和95%置信区间,在相同的空间上能展示更多与样本相关的信息。
3.类箱线图与直方图的比较
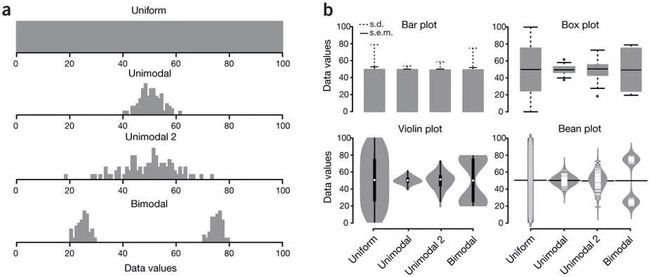
下图不同数据分布的柱状图/条形图t、箱线图、小提琴图和豆图。柱状图/条形图只能比较数据标准差或标准误的异同,箱线图可以反映数据分布的集中趋势,小提琴图和豆图是对数据分布的真正反映,尤其是Biomodal数据集。
箱形图的数据可视化比较
图a中,100个数据点的样本集,每个数据从上到下依次是均匀分布,具有两种不同方差的两个单峰分布,双峰分布。图b是直方图和箱线图、几种类箱线图可视化的比较,条形图通常仅展示了平均值和标准差,箱线图从下往上,依次展示了数据集的五个指标:最小值,小四分位数,中位数,上四分位数和最大值。小提琴图和豆图是箱线图的一种变形,展示了各个数据集的实际分布。
4.箱线图的生物学意义
在生物医学研究中,通常需要比较具有不同分布的多个数据集。条形图或直方图基于简单的统计测量--平均值和方差,来比较数据集。然而,反应数据总体指标的统计量(平均值和方差等)无法反映数据结构的分布(潜在差异等),可能得到与实际相反的结论。
箱线图利用摘要统计指标(中位数和四分位数)和主要数据(四分位数内的50%的数据)的分布。箱形图可以展示任何数据集的最小值,下四分位数,中位数,上四分位数和最大值,可以反映数据集的分布和差异。
以转录组中最常见到的表达矩阵为例,有的基因的表达丰度上千,有的基因几乎不表达,甚至接近于零,这些从数据本身也很容易看出。可是仅关注平均值和方差时,这些极值的差异就很容易被忽略掉,这也是在选择不同基因表达量计算时,需要关注的一点。
箱线图可以直观展示和比较,某个基因在不同处理下表达量的分布和差异情况,也可以间接评估前面归一化和标准化后的基因表达量。
话说TGCA提供FPKM-UQ的表达量,好多人都很迷惑。其实UQ就是上四分位数,其实就是把分母换成了从最小值到上分位数之间75%的样本。
为什么要这么做呢,用箱线图画一下counts矩阵试试啊,说不定就能找到答案了。
五、总结
正所谓:一图胜千言。
可视化的两个目标:加强对样本数据的理解;以可以看见的方式来比较样本。
箱线图是一种简单但功能强大的图形,可以同时满足这两个目标。
直方图绘图要求至少30个样本,而箱线图最小样本量仅为5。在“箱子”的两条线上提供了更多信息,方便于三个或者更多样本之间进行比较。
最后推荐一个发在nature methods上的boxplot的在线工具:http://shiny.chemgrid.org/boxplotr/,上面最后展示的箱线图,都是用这个工具画的哦!
六、参考资料
Anscome's Quartet https://en.wikipedia.org/wiki/Anscombe%27s_quartet
Visualizing samples with box plots https://www.nature.com/articles/nmeth.2813
BoxPlotR: a web tool for generation of box plots https://www.nature.com/articles/nmeth.2811
https://docs.gdc.cancer.gov/Data/Bioinformatics_Pipelines/Expression_mRNA_Pipeline/
https://www.biostars.org/p/243104/
Same Stats, Different Graphs: Generating Datasets with Varied Appearance and Identical Statistics through Simulated Annealing https://www.autodeskresearch.com/publications/samestats
R语言学习 - 箱线图(小提琴图、抖动图、区域散点图)https://www.cnblogs.com/freescience/p/7454874.html

全国巡讲约你
第1-11站北上广深杭,西安,郑州, 吉林,武汉,成都,港珠澳(全部结束)
一年一度的生信技能树单细胞线下培训班(已结束)
全国巡讲第13站-杭州(生信技能树爆款入门课)(下一站甘肃兰州,火热报名)