专题汇总
<一> 科学家发布生物进化与多组学综合分析软件
<二> eGPS开篇与使用示例一
<三> eGPS使用示例二
eGPS 下载地址:http://www.egps-software.org/
写作背景
画外音:为什么会有这一系列的教程?官网不是提供了使用手册吗?
答:确实有使用手册,不过使用手册写的都是一些API式的文档(例如Javadoc、Python的document、R的参数手册)。这些文档不大适合用户去实际操作,而适合对这个工具很了解之后直接去查看。一般用户去用一个软件往往是自己已经有数据了,只想知道这个软件怎么用,参数怎么设置,结果怎么解读,最好图和图注都做好了。所以我们还是会写这样的教程。
近期 eGPS 已经更新到1.0.3版本了,我们稳定在大概一两个月更新一版,每次添加新的功能与特性并修复错误,在此特别感谢反馈错误信息与提供意见的使用者。等我们不断地更新、更新再更新。我们可以不妨畅想一下,画个饼例如:“只下一个软件,完成一篇3-4分文章的数据分析”。
这一期我们要介绍的是,这个软件在群体遗传学上的运用,软件的开发者本身所在的实验室擅长的也是这个领域。进行各种群体遗传学分析的输入文件一般都是VCF文件。因为这些数据都是不同样本的基因组数据,因此也可以被称为基因组学分析。
总的来说,群体遗传学的分析包括如下的几大块:检测选择、估计群体历史、描述多样性、探究重组率、生成模拟数据等。我们现在已经实现了其中的一部分。
还有一些数据的查看与操作,这些以前可能需要写一些命令行的脚本才行,这些通过一个软件就能完成。
eGPS实现的基于VCF的相关分析
基因组的各个功能模块主要是针对VCF文件各种处理,包括:
- 快速查看VCF文件中的内容
- 过滤VCF文件中的variant记录
- 针对VCF文件计算每个滑动窗口的统计量
- 构建基于基因组的系统发育树
- 根据群体历史模拟数据
本教程基于 v1.0.3 http://www.egps-software.org/ 现在最新版是 v1.0.3
场景:假设研究的的物种要做一些群体遗传学的分析,公司做好了前置的一些工作,然后将VCF文件发给了你。当然,或者公司只是把原始数据发给了你,你可以通过eGPS Cloud等方式进行call SNP流程,生成VCF文件。
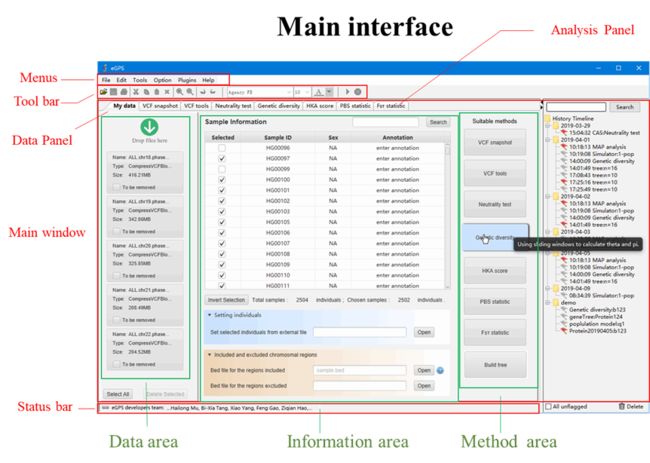
主界面介绍
首先将数据导入eGPS.
导入VCF文件之后,数据面板 的数据区域将会显示文件所包含的个体信息。用户可以直接点击表格中的勾选框以选择你感兴趣的个体。 反转按钮可以对现在勾选的情况,进行反选操作。
如果用户希望一次性批量地选择多个个体,可以将包含个体名称的文本文件通过点击Setting individuals 栏目下的Open按钮将文本文件输入进去。注意文件的后缀名需要为txt。
同时我们还支持输入Bed格式的文件,选择你要分析的目标区域。
现在eGPS的分析功能将在Suitable methods区域以若干个Button的形式存在,我们下面来逐一讲述。
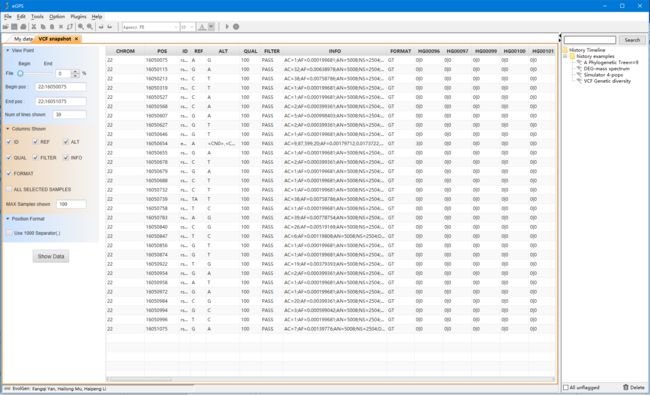
VCF快速查看
点击VCF Snapshot 即可进入快速查看模块,进入后如下图所示:
对于.gz结尾的VCF文件,如果是bgzip压缩的,我们支持快速跳到用户所要查看的区域。gzip压缩的话,可以通过VCF Tools模块生成bgzip压缩的文件。对于未压缩的纯文本文件,只能用左上角的滑动条大致查看文件内容。
更多的详细设置,请查看官网使用手册!
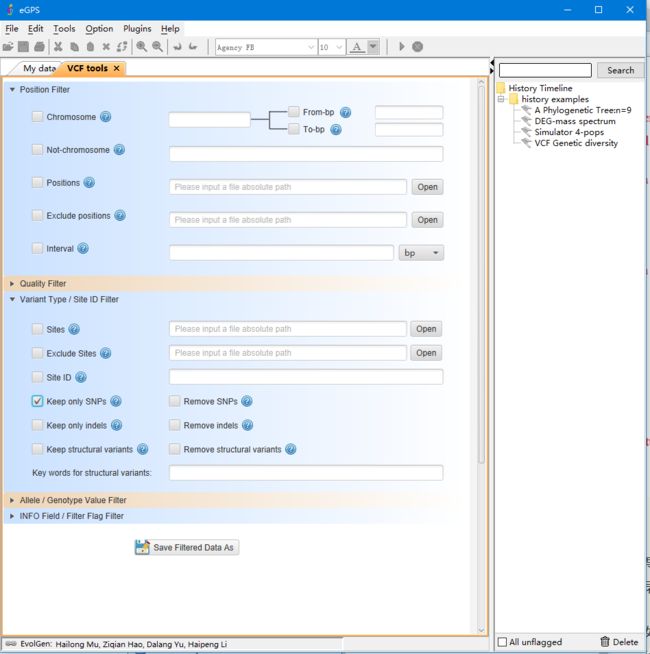
筛选VCF文件中的variant
Variant就是所有可能的突变类型的总称,很多时候我们需要对SNP做下游分析,这个时候,我们就可以用VCF Tools来筛选。
点击VCF Tools 即可进入快速查看模块,进入后如下图所示:
例如我们可以勾选 Keep only SNPs 选项,然后输出bgzip压缩的文件。
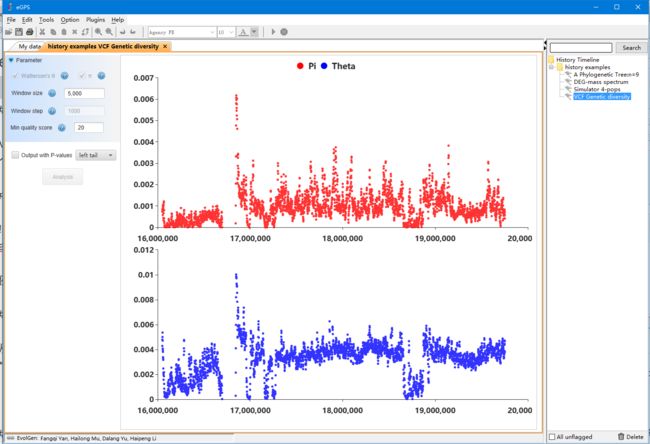
计算每个滑动窗口的统计量
从第三到倒数第二个Button都是根据滑动窗口计算的统计量的模块,我们以 Genetic Diversity 为例。
下面放的是一张从历史记录得到的计算板块与完成后效果图,eGPS可以把一些总要的分析结果储存在历史记录面板中,下次可以回溯结果。
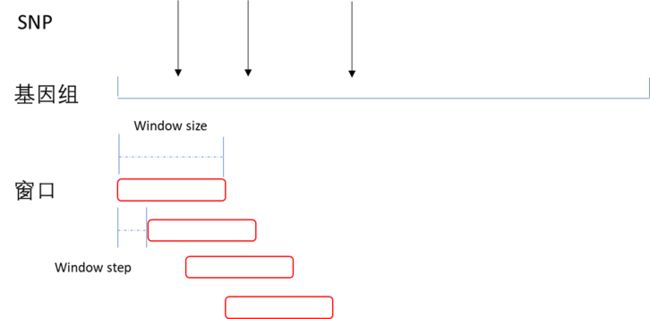
选择你要计算的参数,比如 watterson' theta。 然后设置两个重要参数 window size和 window step。
重要参数 window size和 window step,这两个参数是不一样的,要注意。
其它的几个模块类似,详细使用请见官网使用手册!
根据SNP建立基因组系统发育树
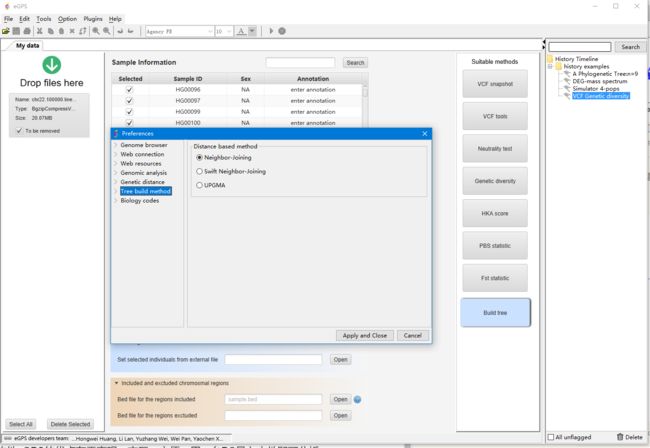
点击build tree将会开始建树,我们打开设置中心,先设置参数。
比如我们在Genetic distance中选bootstrap 1000,遗传距离为K2P,然后构树方法用NJ法,确认并建树,即可得到每个个体的基因组树。
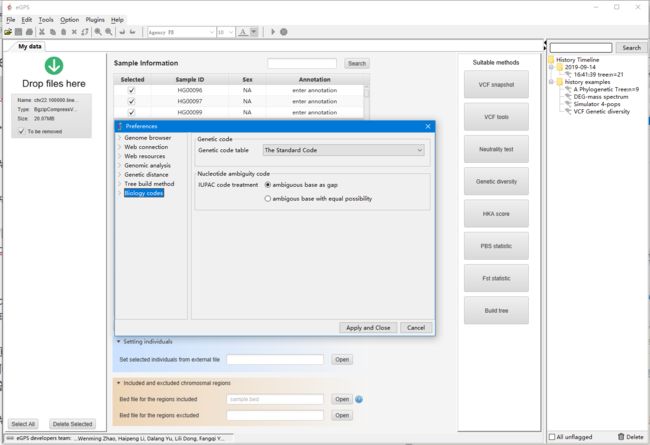
画外音:那我有个疑问,如果是根据VCF的SNP信息建树的话,那杂合子怎么办如何处理?
答:我们提供了两种方式,见下图
因为IUPAC字符中有字符可以表示杂合子,比较有意义的是你可以设置模糊碱基以等可能的概率计算遗传距离。例如R与A的距离就是1/2AA 与 1/2AG,最终是0.5个转换(transition)。
根据群体历史模拟数据
首先我们搭建群体历史模型:
然后我们可以直接就根据溯祖原理模拟数据:
总结:
软件eGPS的优点在于直观、交互、方便。用一台PC导入文件即可分析。
感谢
感谢现在加入群聊的各位人员提供给我们的开发建议:也希望各位能够加入eGPS的使用交流群。QQ group: 550899355
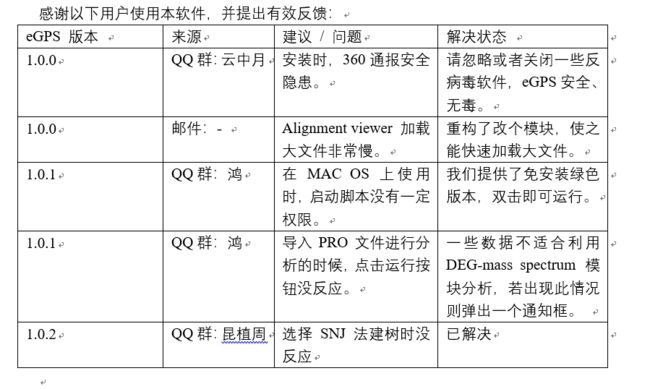
这里特别感谢如下的一些人提供的反馈:
倡议
希望大家多多使用eGPS,我们还会不断加功能,我们会一直开发,一直更新。欢迎你直接来提一些详细、靠谱的需求,例如,下面这个链接的文案:https://pan.baidu.com/s/1l3iFmcZEBEI8wLoXVNbBEQ