DNA 组装(Assembly)
DNA Assembly是分子克隆(Molecular cloning,即核酸片段的in vivo增殖)中不可或缺的步骤。它将目标核酸序列引入微生物体的载体(Vector)中,通常充当vector的是细菌的质粒
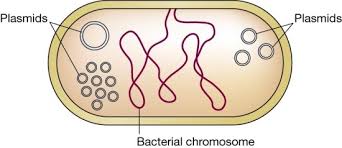
质粒(Plasmid)
- 质粒是除基因组之外的环状DNA,是微生物的交流工具
- 易于储存、提取和转化,是基因工程的载体
- 大小在几k到十几k,最大达200k
质粒的结构
质粒由三个部分组成:复制起始位点(Origin)、AB(抗生素)Resistance、多克隆位点(MCS)。其中 AB Resistance 用于表达抗生素抗性蛋白,用于筛选成功转入质粒的细菌。MCS用于组装目标基因:商业化质粒的MCS往往含有多种Restriction site且每个位点唯一
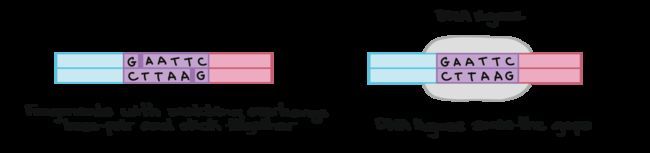
DNA的连接(ligation)
DNA连接酶(ligase)可将缺口(nick,即断裂的磷酸二酯键)修复连接。而裂口(gap,有碱基的缺失)则首先需要DNA Polymerase的活性,最后DNA ligase修补nick
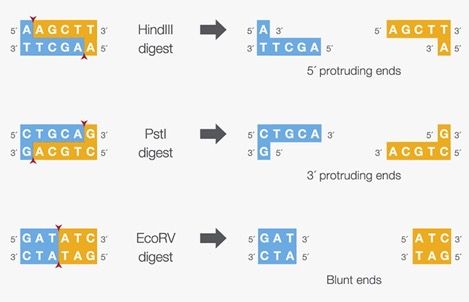
DNA的限制性酶切(Ristricted Digestion)
- Ristricted指酶切具有特定的识别位点(Restriction Site)
- 末端:粘末端(可互补配对),分为5'粘末端与3'粘末端
- 来源:细菌抵御来自噬菌体等的外源性DNA时会将其切断。 a)若切割某一固定位点,为限制性内切酶(ristricted endonuclease)(自身序列往往很少含有此类内切酶的酶切位点,或即使含有,也受到甲基化保护,以防止自身DNA受到切割) b)若切割位点与给定外源DNA序列有关,则为CRISPER
标准化组装
标准化组装试图解决MCS只能组装一个DNA片段的缺陷。其方法有两种
- 每次组装一个片段,可重复组装(以Biobrick Assembly为代表)
- 一次组装多个片段(以Golden Gate为代表)
SLIC组装
SLIC(Sequence and Ligation independent cloning)发明于2007年,是一种一次组装多个片段的组装方法。SLIC在目标片段上构造overlap,并使得overlap形成互补的粘末端
- 首先设计PCR引物,使得DNA片段拥有相邻片段的端部序列
- 利用T4 DNA Polymerase的3'端外切活性形成DNA片段的粘性末端。粘性部分的长度由反应时间粗略控制
- 退火,粘末端互补,利用DNA Polymerase 和 DNA Ligase修补gap
缺点:无法应用与较短片段的连接(3'端外切无法准确控制长度)。Polymerase补全gap时容易引入错误
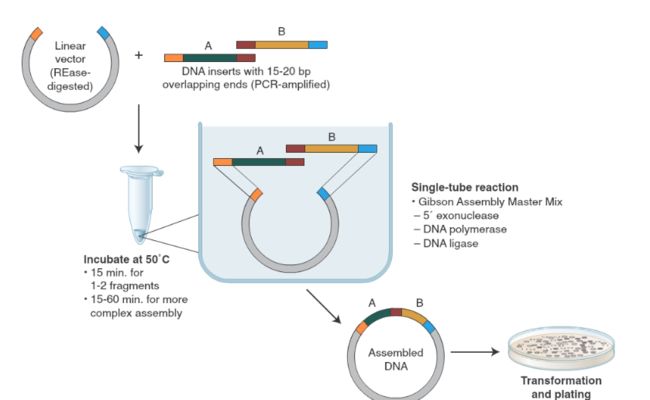
Gibson组装
Gibson组装的基本思想与SLIC无异,也是一种利用overlap的拼接策略。其改进之处在于将外切、聚合、连接集中于一个反应之中。
2010年,Venter实验室发表了人造生命Synthia的合成,其第一作者Gibson以自己的名字命名并发表了这种DNA组装方式。正是这种新颖的组装方式帮助人类造出了第一例完全携带人造DNA的生命体
典型protocal的反应溶液包括T5 exonuclease、Phusion polymerase与Taq ligase。5'外切酶首先将DNA片段的3'末端暴露,随后粘末端互补,phusion聚合酶的聚合作用快于exonuclease的外切活性,最终追及形成nick,由Taq ligase补全
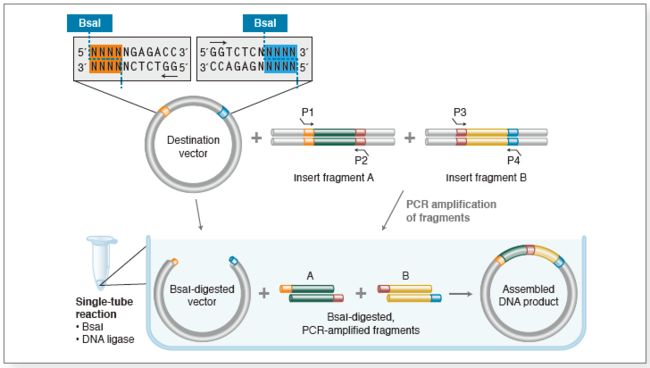
Golden Gate组装
Golden Gate组装是一种利用限制性内切酶进行一次多个片段组装的策略。它使用了一种与众不同的限制性内切酶——BsaI,其识别序列与酶切序列不在同一位置
限制性内切酶BsaI提供了这样两个特性:1.酶切后的粘末端可以人为设计 2.切口两侧仅有一侧拥有酶切位点。利用前一个特性,可以指定多个片段相互连接的先后顺序。利用后一个特性,可以保证在酶切活性的环境中,仅有正确连接的片段能保存完整,而错误连接的片段会再次被酶切
以两个片段的Golden Gate组装为例。Vector质粒提供载体,而Target质粒提供目标片段。将Vector的BsaI识别区域设计在内内侧,而将Target的BsaI设别区域设计在外侧(如图)。如此仅有正确连接(Vector + Target)的质粒不含有BsaI识别位点
将设计好的片段与BsaI、ligase混合在高温(37℃)与低温(16℃)之间迭代数次。高温有利酶切而低温有利连接,每次迭代会导致未连接或连接回原质粒的片段再次遭到酶切,从而以更大的概率保留正确组装的质粒。最后以55℃加热,有利于酶切且ligase失活,尽可能排除错误的组装
BioBrick组装
BioBrick是最早提出的DNA标准化组装策略,是iGEM竞赛中最常用的组装策略。2004年由MIT提出
同尾酶(isocaudomer)指识别序列不同而酶切后粘末端相同的序列,如XbaI与SpeI。其粘末端在退火后互补,但互补后的片段不再含有酶切位点
利用一组同尾酶和另外两个酶切位点(EcoRI、PstI),BioBrick实现了可重复的、顺序性的DNA拼接。例如,将A片段拼接在B片段之前,则首先将A质粒用E与S酶切,将B质粒用E与X酶切。分别PCR扩增目标序列,随后将二者混合,退火并连接即可。连接后的质粒仍然带有EX/SP前缀与后缀,因此仍是一个可以继续连接组件的BioBrick元件
BioBrick组装会在序列之间留下一段8bp的疤痕(scar),导致ORF的移动,影响了融合蛋白(fusion protein)的表达。2008年提出的BB-2策略修改了酶切位点,使得Scar变为6bp
另一种改进策略将BioBrick的同尾酶替换为BgI II与BamH I。这对同尾酶产生的scar可被翻译为短蛋白linker从而应用于fusion protein
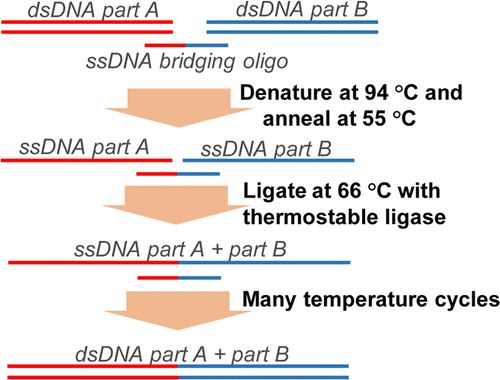
LCR(Ligase Cycling Reaction)组装
LCR的思路与PCR有些相似。LCR是一种比较暴力的,基于耐热ligase的DNA组装方法,发明于2016年。同PCR一样,LCR也要求首先一段单链“引物”,只不过在LCR中这段引物同时和两段DNA片段互补,成为单链桥接寡核苷酸(single strand bridging oligo)。高温下DNA变形后有一定几率恰好同时与此LCR引物结合,退火后耐热ligase修补缺口。经过多个循环,大量的DNA片段在bridging oligos的引导下被ligase连接
此方法可应用于在长片段、多片段的连接中正确率相比传统方法更高,但成功率一般