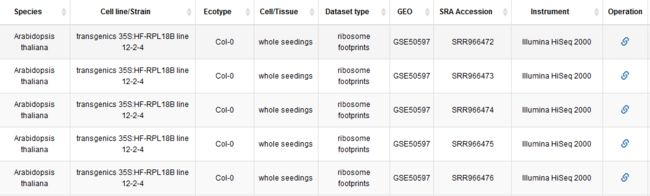
之前老师给了我一个储存RNA-seq和Ribo-seq数据的数据库translatome db让我在上面下数据,但是这个数据库本身并不存储测序数据,每一组数据需要点击后面的链接,得到相应的NCBI链接,再去根据链接自行下载:
这样一个一个操作太过于麻烦,上千组数据可以点到毕业,所以必须用爬虫爬取。记得当年出于学习的目的用实验室电脑在P站上爬过1T的人体写真,虽然这些图片至今我一眼都没看过,但我有一个朋友看了之后说还不错。
言归正传,首先想到的是python自带的requests库:
看得出来作者也是个逗比。
我的目的很明确,爬取网站上左右Ribo-seq数据的SRR编号,方便转化为下载链接。
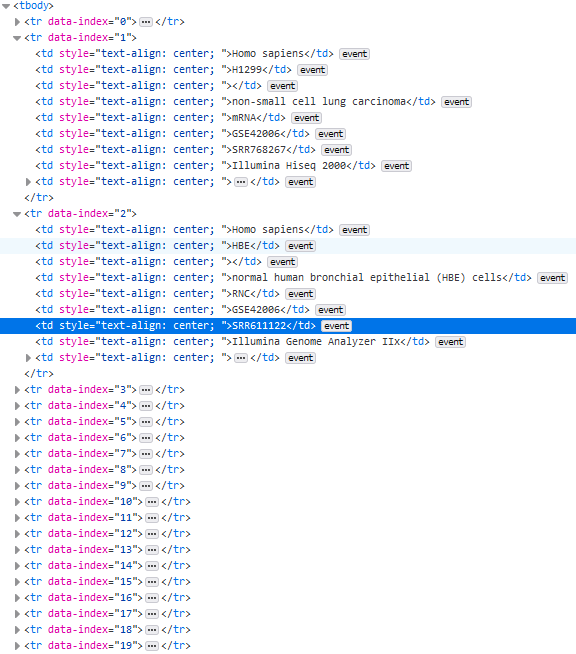
首先,进入网站包含SRR号码的页面,右键网页,检查元素,看看我们要的SSR号藏在哪里:
可以发现data-index代表不同的数据,而所有的信息就包含在不同的index里。看起来很简单。
- 第一步,加载包并设定网站url和请求头:
import requests
import json
import pandas as pd
url = 'http://www.translatomedb.net/SWSYWS.asmx/searchExperimentalData'
header={'User-Agent':'Mozilla/5.0 (Macintosh; Intel Mac OS X 10.15; rv:77.0) Gecko/20100101 Firefox/77.0',
'Host':'www.translatomedb.net',
'Content-Type': 'application/json; charset=UTF-8',
'Accept': 'application/json, text/javascript, */*; q=0.01',
'Accept-Encoding': 'gzip, deflate',
'Referer': 'http://www.translatomedb.net/searchDownload.html',
'Origin': 'http://www.translatomedb.net',
'Content-Type': 'application/json; charset=UTF-8',
'Accept-Language': 'zh-CN,zh;q=0.8,zh-TW;q=0.7,zh-HK;q=0.5,en-US;q=0.3,en;q=0.2',
'X-Requested-With': 'XMLHttpRequest',
'Connection': 'keep-alive',
'Cookie':'Hm_lvt_db1e66967eaace6d18db99b987c0c0ee=1591430772,1591875677; Hm_lpvt_db1e66967eaace6d18db99b987c0c0ee=1591878733'}
- 第二步,发送请求
response = requests.get("https://www.baidu.com", headers=header)
但是使用正则表达式在返回的数据里没有匹配到SRR编号,发现并没有这些信息。也就是说,我请求到的数据和检查元素看到的数据并不一样——requests.get()得到的数据不全。
后来发现,这个网站并非是静态的html网页,中间数据框的那一部分是渲染后的代码,并不是原始url对应的代码。那么,怎么获得中间的数据呢?
既然是动态的,那就动态分析,打开网络监控:
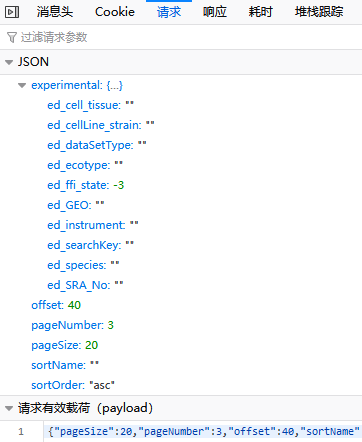
然后在网页上点击下一页,发现有一个post请求:
点开这个请求,发现在我点击的时候发送了如下数据:
那么就可以轻易的模拟出这个请求,网站一共291页,我们就可以发送291次请求:
# 用于储存ribo-seq数据的列表
ribo_dic = []
# 迭代发送请求,一共291页
for j in range(1,291):
# 模拟请求
pyload = {"pageSize":20,
"pageNumber":j,
"offset":5780,
"sortName":"",
"sortOrder":"asc",
"experimental":{"ed_species":"",
"ed_cellLine_strain":"",
"ed_ecotype":"",
"ed_cell_tissue":"",
"ed_dataSetType":"",
"ed_instrument":"",
"ed_GEO":"",
"ed_SRA_No":"",
"ed_searchKey":"",
"ed_ffi_state":-3}}
# 得到返回值
r = requests.post(url,data=json.dumps(pyload),headers=header)
# 返回值为json格式的,进行解析
result = r.json()
# 将解析结果转换为字典
result_dic = dict(result)
# 选择Ribo-seq数据
for i in result_dic['d']['rows']:
# 判断是否为ribo-seq
if i['ed_dataSetType'] == 'ribosome footprints' or i['ed_dataSetType'] == 'ribosome footprint' or i['ed_dataSetType'] == 'RPF':
ribo_dic.append(i)
# 保存所有信息
table = pd.DataFrame(ribo_dic)
# 丢弃不需要的信息
table = table.drop(['__type', 'ed_id', 'ed_sp_id'], axis=1)
table.to_csv('./result.csv')
- 转换为下载链接
EBL下载链接有三种,可以都输出到下载脚本里,总有一个是对的。
#!usr/bin/python3
import argparse
def readfile(file):
link_1 = []
link_2 = []
link_3 = []
command = 'wget -nc '
forward = 'ftp://ftp.sra.ebi.ac.uk/vol1/fastq/'
back = '.fastq.gz &'
list_1 = []
for i in file:
list_1.append(i.strip('\n'))
for i in list_1:
link_1.append(command + forward + str(i[0:6]) + '/' + str(i) + '/' + str(i) + back)
link_2.append(command + forward + str(i[0:6]) + '/0' + str(i[-2:]) + '/' + str(i) + '/' + str(i) + back)
link_3.append(command + forward + str(i[0:6]) + '/00' + str(i[-1:]) + '/' + str(i) + '/' + str(i) + back)
final_list = link_2 + link_1 + link_3
return final_list
def writefile(x):
f1 = open('result.sh', 'w+')
for i in x:
f1.write(i)
f1.write('\n')
f1.close()
if __name__ == '__main__':
parse = argparse.ArgumentParser('Generate url link of EBI from SRRid')
parse.add_argument('-f', dest='file', required=True, help='SRRid file')
arg = parse.parse_args()
file = open(arg.file)
f_list = readfile(file)
writefile(f_list)
这里的-f是每行包含一个SRR号的文件。直接批量生成下载脚本。