近年来,全基因组CRISPR文库筛选技术被广泛应用于肿瘤及病毒相关调控因子筛选。通过同时sgRNA文库同时敲除全基因组的不同基因,经流式或药物筛选目的细胞表型,并扩增sgRNA区域序列及NGS测序,由此获得显著富集或减少的sgRNA所对应的基因。这种通过细胞表型鉴定基因表型的方式可以帮助我们快速鉴定影响肿瘤生长或病毒复制等过程中的关键基因,找到一些未知的生物学机制。那么,针对CRIPSR文库筛选测序的结果该如何分析呢?本文将为大家介绍一款非常实用的数据分析工具,即MAGeCKFlute。让我们一起来学习吧!
前期准备:
硬件:
64位电脑,大于4GB内存,安装有Linux系统,例如CentOS 7。
软件:
MAGeCK v.0.5.6版本及以上(https://sourceforge.net/projects/mageck/)
MAGeCK-VISPR v.0.5.3版本及以上(https://bitbucket.org/liulab/mageck-vispr)
Conda v.4.5.4版本及以上(https://conda.io/docs/)
Python v.3.4.3版本及以上(https://www.python.org)
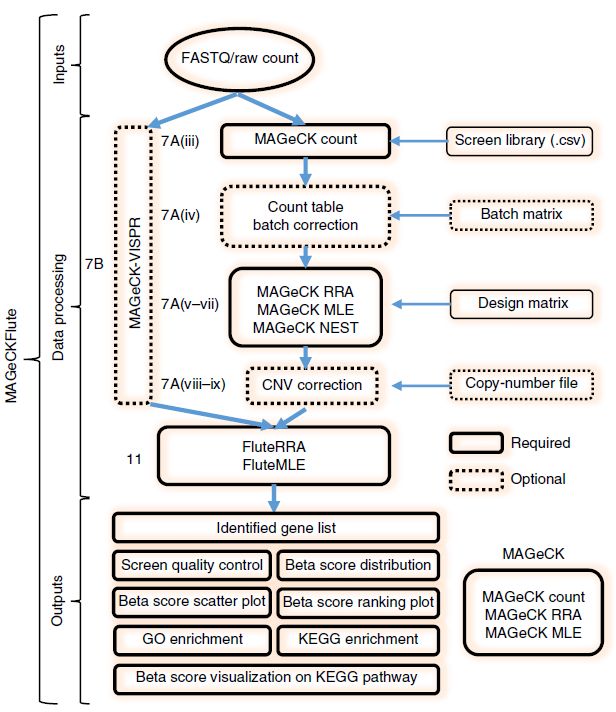
分析流程如图所示:
环境部署:
1. 下载及安装Miniconda(https://docs.conda.io/en/latest/miniconda.html)并存在至本地目录;
$bash path/to/file/Miniconda3-latest-Linux-x86_64.sh
2. 添加Bioconda channels,为MAGeCK创建独立的工作环境
$conda config --add channels conda-forge
$conda config --add channels bioconda
$conda create -n mageck-vispr mageck mageck-vispr python=3
$conda activate mageck-vispr
3. 安装MAGeCK NEST
从https://bitbucket.org/liulab/mageck_nest/src/master/网站下载源代码,文件名为‘mageck_nest-3.0.tar.gz’,解压缩并安装:
$cd path/to/file/
$tar -zxvf mageck_nest-3.0.tar.gz
$cd mageck_nest-3.0
$python3 setup.py install
数据分析:
1. 进入FastQ测序文件的存放目录,采用MAGeCK处理CRISPR screen测序数据:
$mageck count -l library.csv -n TreatedvsCtrl --sample-label Treated_R1,Treated_R2,Ctrl_R1,Ctrl_R2 --fastq Treated_combined_R1.fastq.gz Treated_combined_R2.fastq.gz Ctrl_combined_R1.fastq.gz Ctrl_combined_R2.fastq.gz
其中library.csv文件格式如下图所示:
2. 采用MAGeCK RRA寻找靶基因
$mageck test -k TreatedvsCtrl.count.txt -t Treated_R1,Treated_R2 -c Ctrl_R1,Ctrl_R2 -n TreatedvsCtrl_rra --remove-zero both --remove-zero-threshold 0
3. 下游分析
采用R安装MAGeCKFlute包
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("MAGeCKFlute")
Library(MAGeCKFlute)
FluteRRA(gene_summary = "path/to/file/TreatedvsCtrl_rra.gene_summary.txt", prefix="FluteRRA", organism="hsa")
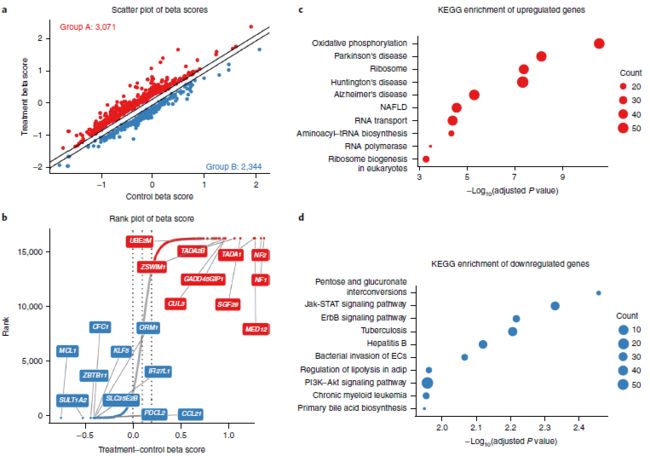
部分结果展示:
参考文献:
[1] Shalem O, Sanjana NE, Zhang F. High-throughput functional genomics using CRISPR-Cas9. Nat Rev Genet. 2015 May;16(5):299-311.
[2] Wang BB, Wang M, Zhang WB, et al. Integrative analysis of pooled CRISPR geneticscreens using MAGeCKFlute. Nat Protoc. 2019 March; 14:756-780.
怎么样,是不是很简单呢?快来试一试吧!