机器学习之主成分分析(PCA)
降维的目的
- 进行数据降维处理,我们可以容易地实现高维数据可视化——将其降为三维甚至二维。
- 由于可能存在许多冗余特征量,所以需要减少特征量的数量。(比如同时有英寸和米的数据,这样的数据是线性相关的,可以去掉其中之一)
- 机器学习算法的复杂度和数据的维数有着密切关系,甚至与维数呈指数级关联。因此我们必须对数据进行降维。
降维的方法
在机器学习领域,我们对原始数据进行特征提取,有时会得到比较高维的特征向量。在这些向量所处的高维空间中,包含很多的冗余和噪声。我们希望通过降维的方式来寻找数据内部的特性,从而提高特征的表达能力,降低训练复杂度。
主要有三种方法:1)主成分分析(PCA);2)因子分析(FA);3)独立成分分析(ICA)。其中 PCA是一种具有严格数学基础并且已被广泛采用的降维方法。它属于一种线性、无监督、全局的降维方法。
PCA的理论基础和思想
- PCA理论中,主要是用到线性代数中的特征值和特征向量。(详情可参考本博主的特征值和特征向量的几何意义的博文)
- PCA的目标是降低每一个特征量到投影直线的距离,也就是降低投影误差。
- PCA深入浅出的基本思想讲解还可参考
吴恩达老师的《机器学习课程》 - 用到的统计中的概念:均值、方差和协方差矩阵
给定一个含有m个样本的集合:X={X1,…,Xm}
均值:
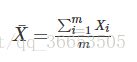
方差:
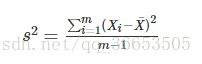
均值描述的是样本集合的平均值,方差描述的是样本集合中各个点到均值的距离的平均,也就是数据的离散程度。
如果数据的维数比较多的话,我们想知道某两个特征之间是否有关系,比如说统计学生的成绩,数学成绩和物理成绩
之间是否有关系,如果有是正相关呢还是负相关,协方差就是用来描述两个随机变量X,Y之间关系的统计量,协方差
协方差的结果为正的话,表明二者之间是正相关,为负的话表明二者之间是负相关。这里的X,Y两个一维的随机变量,
如果随机变量的个数很多呢,这时候就引入了协方差矩阵的概念:对于一个n维的数据,分别计算每两个维度之间的协方差,就构成了一个协方差矩阵:

可见这是一个对称矩阵,对角线上的是方差。如果数据是三维的,则协方差矩阵如下:

PCA算法实现过程推导
-
给定一个训练集 x 1 x^{1} x1, x 2 x^{2} x2,…, x m x^{m} xm。其中m表示样本数量, x i x^{i} xi是表示
i样本的n维特征向量(为列向量)
x ( i ) = ( x 1 ( i ) x 2 ( i ) . . . x n ( i ) ) x^{(i)}=\begin{pmatrix} x^{(i)}_{1} \\x^{(i)}_{2}\\.\\.\\.\\x^{(i)}_{n} \end{pmatrix} x(i)=⎝⎜⎜⎜⎜⎜⎜⎜⎛x1(i)x2(i)...xn(i)⎠⎟⎟⎟⎟⎟⎟⎟⎞ -
对数据进行预处理(特征缩放,均值为零)
μ j = 1 m ∑ i = 1 m x j ( i ) μ_{j}=\frac{1}{m}\sum_{i=1}^{m}x^{(i)}_{j} μj=m1i=1∑mxj(i)
用 x j ( i ) − μ j x^{(i)}_{j}-μ_{j} xj(i)−μj来代替原来的特征 x j ( i ) x^{(i)}_{j} xj(i) -
构造协方差矩阵,对于单个样本
i
c o v = ( x ( i ) ) ( x ( i ) ) T cov=(x^{(i)})(x^{(i)})^{T} cov=(x(i))(x(i))T
对所有样本的协方差矩阵,就是对所有样本求个平均值
C = 1 m ∑ i = 1 m ( x ( i ) ) ( x ( i ) ) T C=\frac{1}{m}\sum_{i=1}^{m}(x^{(i)})(x^{(i)})^{T} C=m1i=1∑m(x(i))(x(i))T设所有样本组成的矩阵为A
A = ( ∣ ∣ ∣ ∣ x ( 1 ) x ( 2 ) ⋯ x ( m ) ∣ ∣ ∣ ∣ ) A=\begin{pmatrix} | & | &| &|\\x^{(1)} &x^{(2)} &\cdots &x^{(m)}\\| &| &| &|\end{pmatrix} A=⎝⎛∣x(1)∣∣x(2)∣∣⋯∣∣x(m)∣⎠⎞
则
C = 1 m A A T C=\frac{1}{m}AA^{T} C=m1AAT -
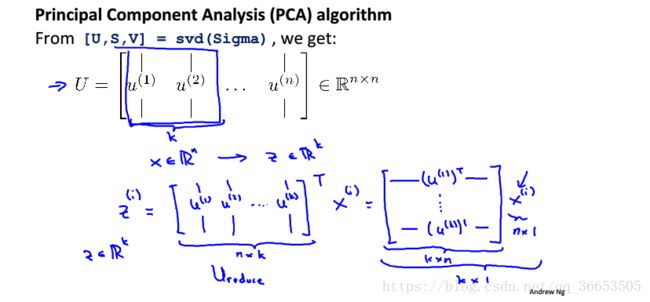
最后使用选取的特征向量(这些特征向量构成了低维的子空间),对特征进行降维,如下图(同样来自吴恩达老师课件):
最大方差理论推导PCA
在信号处理中认为信号具有较大的方差,噪声有较小的方差,信噪比就是信号与噪声的方差比,越大越好。如前面的图,样本在横轴上的投影方差较大,在纵轴上的投影方差较小,那么认为纵轴上的投影是由噪声引起的。所以我们认为最好的k维特征是将n维的样本点转化之后,每一维上的样本方差都很大。
比如下图右5个样本点,所有的点已经做过预处理,均值为0,特征方差归一。
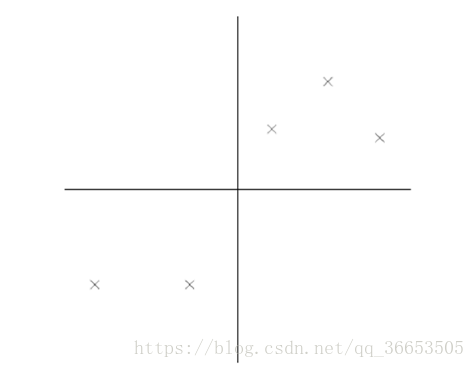
对与这样的一个二维平面上的点,我们现在想要把它投影到一个一维中去,也就是找到一条直线,将这些点全部投影到直线上去,达到降维的目的。下面是两条直线的比较
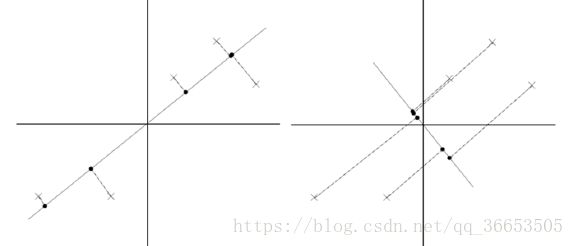
很明显,左边那条直线的投影效果更好,数据的方差更大,越有利于分类。

如图所示,红色的点表示的是样本点,蓝色的点表示的是样本点在向量u上的投影点,这里规定u是单位向量,则x在u上的投影可以表示为 x T u x^{T}u xTu,假设现在有很多个样本点x,我们希望存在这样的一个u使得这些样本点投影到u上之后方差最大。由于样本点的每一维特征的均值都是0,所以投影到u上之后的样本点的均值也是0。
计算方差:
D ( x ) = 1 m ∑ i = 1 m ( x ( i ) T u ) 2 = 1 m ∑ i = 1 m u T x ( i ) x ( i ) T u = u T ( 1 m ∑ i = 1 m x ( i ) x ( i ) T ) u D(x)=\frac{1}{m}\sum_{i=1}^{m}(x^{(i)^{T}}u)^{2} = \frac{1}{m}\sum_{i=1}^{m}u^{T}x^{(i)}x^{(i)^{T}}u=u^{T}(\frac{1}{m}\sum_{i=1}^{m}x^{(i)}x^{(i)^{T}})u D(x)=m1i=1∑m(x(i)Tu)2=m1i=1∑muTx(i)x(i)Tu=uT(m1i=1∑mx(i)x(i)T)u
其中 D ( x ) D(x) D(x)表示所有样本的投影方差。很显然,中间括号的那一部分就是所有样本的协方差矩阵,唯一不同的地方就是协方差计算的时候是除m-1,这里是除m。好了,现在的目标就是要最大化这个式子,用 ∑ \sum ∑表示协方差,用 λ \lambda λ表示优化目标,那么上述问题可以写成:
![]()
两边同时乘以u,u是单位向量,所以右边 u T u u^{T}u uTu相乘的结果是1,变成
![]()
也就是
![]()
所以,最大化方差,即最大化 λ \lambda λ,就是求解协方差矩阵的最大特征值,最佳投影方向就是最大投影值所对应的特征向量。次佳投影方向位于最佳投影方向的正交空间,是第二大特征值对应的特征向量,以此类推。
要选择多少个主成分,就意味着就选择多少个特征向量构成的子空间对数据进行降维。对于要选择的主成分(特征向量)数量,可用下式计算:
η = ∑ i = 1 k λ i 2 ∑ i = 1 n λ i 2 \eta = \sqrt{\frac{\sum_{i=1}^{k} \lambda_{i}^{2}}{\sum_{i=1}^{n}\lambda_{i}^{2}}} η=∑i=1nλi2∑i=1kλi2
其中k为选择的特征向量的数量,即为子空间的维数,n为每个样本的特征维数。
或者用最小平方误差理论推导PCA
这里省略最小平方误差理论的推导,一般理解最大方差理论就够啦。。。。
PCA算法实现(python)
以吴恩达老师的《机器学习》课程关于PCA的作业为例:
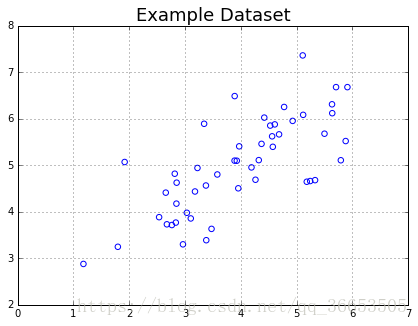
- 展示数据
datafile = 'data/ex7data1.mat'
mat = scipy.io.loadmat( datafile )
X = mat['X']
#Quick plot
plt.figure(figsize=(7,5))
plot = plt.scatter(X[:,0], X[:,1], s=30, facecolors='none', edgecolors='b')
plt.title("Example Dataset",fontsize=18)
plt.grid(True)
- 实现PCA
## 特征归一化
def featureNormalize(myX):
#Feature-normalize X, return it
means = np.mean(myX,axis=0)
myX_norm = myX - means
stds = np.std(myX_norm,axis=0)
myX_norm = myX_norm / stds
return means, stds, myX_norm
## 使用SVD分解求解特征值和特征向量
def getUSV(myX_norm):
# Compute the covariance matrix
cov_matrix = myX_norm.T.dot(myX_norm)/myX_norm.shape[0]
# Run single value decomposition to get the U principal component matrix
U, S, V = scipy.linalg.svd(cov_matrix, full_matrices = True, compute_uv = True)
return U, S, V
# Feature normalize
means, stds, X_norm = featureNormalize(X)
# Run SVD
U, S, V = getUSV(X_norm)
# "...output the top principal component (eigen- vector) found,
# and you should expect to see an output of about [-0.707 -0.707]"
print 'Top principal component is ',U[:,0]
#Quick plot, now including the principal component
plt.figure(figsize=(7,5))
plot = plt.scatter(X[:,0], X[:,1], s=30, facecolors='none', edgecolors='b')
plt.title("Example Dataset: PCA Eigenvectors Shown",fontsize=18)
plt.xlabel('x1',fontsize=18)
plt.ylabel('x2',fontsize=18)
plt.grid(True)
#To draw the principal component, you draw them starting
#at the mean of the data
plt.plot([means[0], means[0] + 1.5*S[0]*U[0,0]],
[means[1], means[1] + 1.5*S[0]*U[0,1]],
color='red',linewidth=3,
label='First Principal Component')
plt.plot([means[0], means[0] + 1.5*S[1]*U[1,0]],
[means[1], means[1] + 1.5*S[1]*U[1,1]],
color='fuchsia',linewidth=3,
label='Second Principal Component')
leg = plt.legend(loc=4)
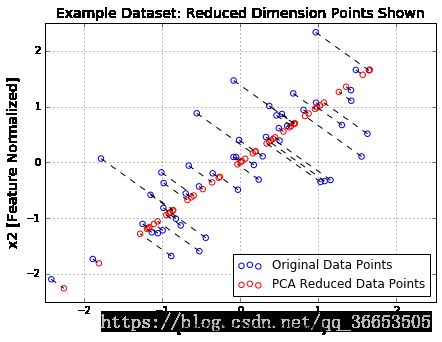
- 降维
def projectData(myX, myU, K):
"""
Function that computes the reduced data representation when
projecting only on to the top "K" eigenvectors
"""
#Reduced U is the first "K" columns in U
Ureduced = myU[:,:K]
z = myX.dot(Ureduced)
return z
# "...project the first example onto the first dimension
# "and you should see a value of about 1.481"
z = projectData(X_norm,U,1)
print 'Projection of the first example is %0.3f.'%float(z[0])
def recoverData(myZ, myU, K):
Ureduced = myU[:,:K]
Xapprox = myZ.dot(Ureduced.T)
return Xapprox
#Quick plot, now drawing projected points to the original points
plt.figure(figsize=(7,5))
plot = plt.scatter(X_norm[:,0], X_norm[:,1], s=30, facecolors='none',
edgecolors='b',label='Original Data Points')
plot = plt.scatter(X_rec[:,0], X_rec[:,1], s=30, facecolors='none',
edgecolors='r',label='PCA Reduced Data Points')
plt.title("Example Dataset: Reduced Dimension Points Shown",fontsize=14)
plt.xlabel('x1 [Feature Normalized]',fontsize=14)
plt.ylabel('x2 [Feature Normalized]',fontsize=14)
plt.grid(True)
for x in xrange(X_norm.shape[0]):
plt.plot([X_norm[x,0],X_rec[x,0]],[X_norm[x,1],X_rec[x,1]],'k--')
leg = plt.legend(loc=4)
#Force square axes to make projections look better
dummy = plt.xlim((-2.5,2.5))
dummy = plt.ylim((-2.5,2.5))
后记
降维是通过低维的向量更好的表示特征。。与神经网络中的稀疏编码表示输入向量在思想上有相似之处。
参考
- https://www.cnblogs.com/lzllovesyl/p/5235137.html
- https://blog.csdn.net/zhongkelee/article/details/44064401(最大方差理论详细推导)
- https://blog.csdn.net/YZXnuaa/article/details/80746287
- https://blog.csdn.net/aiiyouwei/article/details/80173658
- https://blog.csdn.net/huang1024rui/article/details/46662195
- https://www.zhouyongyi.com/andrew-ng-machine-learning-notes-10/
- https://blog.csdn.net/zouxy09/article/details/8775518