前言
有关GCN的原理可以参考:GCN图卷积神经网络原理
一开始是打算手写一下GCN,毕竟原理也不是很难,但想了想还是直接调包吧。在使用各种深度学习框架时我们首先需要知道的是框架内的数据集结构,因此这篇文章主要讲讲PyG中的数据结构。
1. PyG数据集
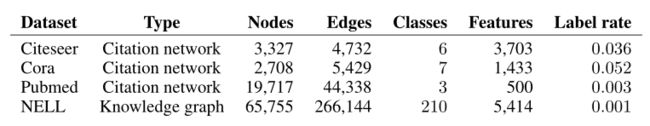
原始论文中使用的数据集:
本篇文章使用Citeseer网络。Citeseer网络是一个引文网络,节点为论文,一共3327篇论文。论文一共分为六类:Agents、AI(人工智能)、DB(数据库)、IR(信息检索)、ML(机器语言)和HCI。如果两篇论文间存在引用关系,那么它们之间就存在链接关系。
使用PyG加载数据集:
data = Planetoid(root='/data/CiteSeer', name='CiteSeer') print(len(data))
输出:
1
CiteSeer中只有一个网络,然后我们输出一下这个网络:
data = data[0] print(data) print(data.is_directed())
输出:
Data(x=[3327, 3703], edge_index=[2, 9104], y=[3327], train_mask=[3327], val_mask=[3327], test_mask=[3327]) False
x=[3327, 3703]。表示一共有3327个节点,然后节点的特征维度为3703,这里实际上是去除停用词和在文档中出现频率小于10次的词,整理得到3703个唯一词。
edge_index=[2, 9104],表示一共9104条edge。数据一共两行,每一行都表示节点编号。
输出一下data.y:
tensor([3, 1, 5, ..., 3, 1, 5])tensor([3, 1, 5, ..., 3, 1, 5])
data.y表示节点的标签编号,比如3表示该篇论文属于第3类。
输出data.train_mask:
tensor([ True, True, True, ..., False, False, False])
data.train_mask的长度和y的长度一致,如果某个位置为True就表示该样本为训练样本。val_mask和test_mask类似,分别表示验证集和训练集。
比如我们输出:
print(data.y[data.test_mask])
结果为:
tensor([4, 5, 4, 4, 4, 1, 4, 2, 3, 3, 3, 3, 2, 3, 3, 4, 2, 0, 1, 2, 0, 3, 3, 4,
2, 4, 0, 4, 3, 3, 3, 5, 4, 5, 4, 5, 1, 1, 3, 3, 3, 3, 3, 1, 2, 3, 3, 3,
1, 2, 2, 3, 3, 1, 5, 5, 5, 3, 2, 3, 3, 3, 3, 3, 3, 3, 5, 1, 3, 1, 1, 4,
1, 3, 3, 1, 3, 3, 2, 4, 3, 3, 3, 1, 2, 2, 2, 3, 5, 2, 1, 3, 2, 2, 2, 4,
3, 3, 4, 0, 3, 1, 2, 2, 2, 2, 3, 2, 2, 2, 1, 1, 5, 2, 2, 1, 2, 4, 3, 1,
1, 3, 2, 3, 4, 3, 3, 4, 4, 3, 2, 2, 1, 3, 4, 4, 4, 4, 4, 4, 5, 0, 3, 1,
1, 3, 1, 3, 1, 3, 4, 4, 3, 2, 3, 5, 3, 3, 3, 4, 2, 2, 2, 5, 3, 1, 0, 3,
2, 5, 2, 3, 2, 4, 2, 2, 2, 0, 5, 1, 3, 4, 4, 4, 1, 1, 5, 1, 2, 0, 1, 0,
2, 2, 3, 3, 3, 3, 5, 4, 4, 3, 1, 1, 2, 1, 2, 2, 2, 2, 5, 0, 1, 2, 2, 4,
0, 4, 1, 1, 2, 3, 1, 1, 2, 3, 3, 5, 2, 5, 5, 3, 1, 0, 5, 5, 5, 5, 3, 3,
3, 0, 4, 5, 3, 4, 5, 4, 5, 2, 0, 5, 5, 5, 1, 1, 3, 1, 2, 2, 2, 3, 2, 4,
5, 3, 3, 1, 3, 1, 2, 2, 1, 3, 1, 3, 1, 2, 1, 2, 1, 2, 2, 2, 2, 5, 4, 4,
5, 0, 3, 4, 5, 4, 4, 4, 4, 4, 0, 0, 1, 4, 1, 1, 5, 0, 2, 2, 3, 3, 2, 2,
0, 0, 3, 2, 4, 1, 1, 0, 0, 1, 2, 2, 2, 2, 2, 0, 4, 0, 1, 4, 1, 1, 2, 2,
3, 3, 1, 3, 2, 4, 4, 0, 0, 3, 4, 4, 2, 2, 2, 5, 5, 2, 5, 5, 5, 5, 4, 0,
2, 2, 0, 2, 4, 5, 4, 0, 3, 3, 5, 3, 3, 4, 2, 1, 5, 5, 0, 1, 3, 3, 3, 5,
3, 3, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 4, 2, 2, 0, 2, 2, 2, 2, 4, 3, 3,
5, 5, 4, 5, 2, 4, 4, 4, 5, 5, 4, 2, 2, 3, 3, 4, 4, 3, 1, 3, 2, 0, 5, 5,
5, 3, 4, 1, 4, 0, 5, 5, 0, 3, 0, 2, 3, 5, 3, 4, 2, 2, 3, 5, 1, 5, 3, 4,
5, 5, 2, 2, 4, 3, 3, 3, 3, 2, 2, 2, 2, 2, 3, 0, 0, 5, 1, 2, 3, 3, 1, 3,
2, 4, 3, 1, 3, 3, 3, 3, 3, 1, 0, 5, 4, 4, 1, 1, 3, 4, 4, 4, 4, 5, 4, 2,
2, 2, 2, 2, 2, 2, 3, 2, 2, 2, 1, 4, 0, 1, 4, 4, 4, 1, 2, 1, 5, 5, 2, 4,
4, 2, 2, 3, 1, 1, 0, 0, 2, 1, 0, 1, 5, 1, 2, 2, 3, 2, 0, 0, 3, 3, 3, 2,
2, 2, 1, 1, 1, 3, 3, 3, 5, 3, 5, 2, 3, 2, 3, 1, 5, 2, 2, 3, 3, 3, 1, 1,
1, 3, 3, 3, 3, 4, 4, 1, 4, 4, 1, 3, 3, 1, 0, 3, 5, 4, 4, 2, 4, 1, 0, 3,
1, 4, 1, 4, 4, 0, 5, 3, 2, 2, 2, 5, 5, 0, 4, 4, 1, 2, 2, 3, 3, 3, 5, 5,
5, 1, 5, 1, 4, 3, 1, 5, 5, 4, 4, 2, 3, 1, 0, 0, 5, 3, 1, 2, 1, 4, 1, 4,
1, 2, 2, 5, 1, 2, 1, 4, 5, 5, 1, 4, 5, 5, 1, 1, 5, 5, 3, 1, 0, 0, 1, 0,
0, 2, 0, 4, 3, 4, 3, 3, 1, 2, 3, 5, 3, 5, 5, 5, 5, 5, 3, 4, 4, 5, 4, 2,
2, 5, 1, 4, 4, 4, 3, 1, 5, 3, 1, 3, 4, 2, 2, 4, 2, 1, 5, 2, 2, 5, 5, 3,
3, 4, 1, 1, 2, 5, 3, 4, 4, 4, 5, 5, 1, 5, 5, 1, 5, 5, 1, 1, 1, 4, 2, 3,
5, 4, 1, 1, 4, 5, 2, 3, 1, 2, 1, 4, 1, 4, 1, 1, 1, 0, 0, 1, 5, 0, 2, 1,
1, 5, 1, 1, 3, 2, 3, 3, 1, 1, 2, 3, 2, 3, 5, 5, 5, 5, 5, 5, 5, 5, 5, 3,
3, 5, 2, 2, 3, 4, 4, 4, 4, 0, 3, 0, 3, 4, 1, 1, 3, 3, 0, 4, 5, 0, 0, 0,
2, 1, 3, 4, 5, 2, 1, 1, 3, 3, 4, 4, 4, 2, 2, 1, 5, 4, 0, 5, 5, 4, 3, 4,
5, 0, 3, 0, 3, 4, 4, 3, 3, 3, 3, 3, 3, 3, 5, 2, 0, 0, 1, 0, 0, 0, 3, 1,
5, 3, 2, 3, 5, 3, 3, 3, 1, 5, 5, 5, 5, 1, 2, 1, 4, 5, 4, 3, 3, 5, 5, 1,
4, 2, 5, 4, 1, 4, 4, 4, 4, 5, 5, 4, 3, 4, 3, 5, 3, 3, 1, 1, 0, 4, 4, 3,
1, 1, 1, 1, 3, 3, 3, 4, 3, 1, 4, 1, 1, 3, 5, 5, 5, 4, 4, 1, 3, 1, 4, 3,
3, 3, 1, 2, 2, 5, 3, 2, 5, 1, 3, 3, 5, 5, 4, 0, 3, 5, 5, 5, 1, 2, 2, 4,
1, 4, 5, 5, 5, 4, 5, 2, 1, 5, 4, 4, 0, 3, 5, 4, 1, 3, 3, 5, 4, 2, 1, 0,
1, 3, 2, 4, 3, 2, 4, 4, 1, 1, 0, 3, 3, 3, 1, 5])
可以发现,我们输出的是测试集的内容。
那么很显然,如果我们最终得到了预测值,我们就可以通过以下代码来计算分类的正确数:
correct = int(pred[data.test_mask].eq(data.y[data.test_mask]).sum().item())
模型输出的pred实际上包含了所有节点的预测值,而我们只需要取测试集中的内容,即:
pred[data.test_mask]
然后再与data.y[data.test_mask]进行比较,最后计算二者对应位置相等的个数即可。
2. 构造数据集
如果我们需要的数据集在PyG中没有,我们就需要自己手动构造数据集。
例如对于一个无向图,我们知道了其节点特征矩阵x:
x = torch.tensor([[-1, 1], [0, 1], [1, 3]], dtype=torch.float)
一共3个节点,每个节点具有两个特征。
然后我们知道了节点间的邻接关系:
edge_index = torch.tensor([[0, 1, 1, 2],
[1, 0, 2, 1]], dtype=torch.long)
一共4条边,第一条边为0->1,第2条边为1->0。
然后我们就可以构造数据集:
data = Data(x=x, edge_index=edge_index)
有关GCN的实现放在下一篇文章!
以上就是PyG搭建GCN需要准备的数据格式的详细内容,更多关于PyG搭建GCN数据格式的资料请关注脚本之家其它相关文章!