k-means、DBSCAN聚类算法代码
k-means聚类算法
优点
容易实现。
适用于高维。
缺点
对离群点敏感,对噪声点和孤立点很敏感。
不适用于非凸的簇或大小差别很大的簇。
需要自定义k,不同的初始点可能得到的结果完全不同。
可能收敛到局部最小值,在大规模数据集上收敛较慢。
适用数据类型
数值型数据。
伪代码
创建k个点作为起始质心(经常是随机选择)
当任意一个点的簇分配结果发生改变时
对数据集中的每个数据点
对每个质心
计算质心与数据点之间的距离
将数据点分配到距其最近的簇
对每一个簇,计算簇中所有点的均值并将均值作为质心
k-means代码
from numpy import *
def loadDataSet(fileName): # 将文本文件导入到列表
dataMat = []
fr = open(fileName)
for line in fr.readlines():
curLine = line.strip().split('\t')
fltLine = list(map(float, curLine)) # 需要在此处将map结果转化为列表
dataMat.append(fltLine)
return dataMat
def distEclud(vecA, vecB): # 计算两个向量的欧式距离
return sqrt(sum(power(vecA - vecB, 2)))
def randCent(dataSet, k): # 为给定的数据集构建一个包含k个质心的集合
n = shape(dataSet)[1]
centroids = mat(zeros((k, n)))
for j in range(n): # 随机质心必须要在整个数据集的边界之内
minJ = min(dataSet[:, j]) # 可以通过找到数据集每一维德最小和最大值来完成
rangeJ = float(max(dataSet[:, j]) - minJ)
centroids[:, j] = minJ + rangeJ * random.rand(k, 1)
return centroids
def kMeans(dataSet, k, distMeas=distEclud, createCent=randCent):
m = shape(dataSet)[0] # 样本数
clusterAssment = mat(zeros((m, 2))) # 创建一个矩阵,存储每个点的簇分配结果(一列记录簇索引值,另一列存储误差)
centroids = createCent(dataSet, k) # 初始化k个质心
clusterChanged = True
while clusterChanged:
clusterChanged = False
for i in range(m): # 遍历每一个样本
minDist = inf
minIndex = -1
for j in range(k): # 对当前样本找到离其最近的质心
distJI = distMeas(centroids[j, :], dataSet[i, :])
if distJI < minDist:
minDist = distJI
minIndex = j
if clusterAssment[i, 0] != minIndex: # 簇分配仍在进行
clusterChanged = True
clusterAssment[i, :] = minIndex, minDist ** 2
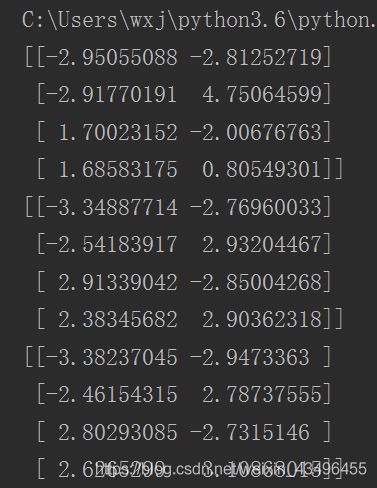
print(centroids)
for cent in range(k): # 更新质心的位置
ptsInClust = dataSet[nonzero(clusterAssment[:, 0].A==cent)[0]] # 每个簇中样本的值
centroids[cent, :] = mean(ptsInClust, axis=0) # 求每个簇的均值
return centroids, clusterAssment
dataMat = mat(loadDataSet('testSet.txt')) # 将文本文件导入到列表,再将列表转换为矩阵
myCentroids, clusterAssing = kMeans(dataMat, 4)
结果:经过3次迭代,算法就已经收敛。
聚类度量指标SSE
SSE(Sum of Squared Error,误差平方和)。SSE越小表示数据点越接近它们的质心,聚类效果也就越好。因为对误差取了平方,因此更加重视那些远离中心的点。
k-均值很容易会收敛到局部最小值,而二分k-均值算法能在相似的思想上达到更好的聚类效果。
二分k-均值算法
为克服K-均值算法收敛于局部最小值的问题,有人提出了二分k-均值的算法。该算法首先将所有点作为一个簇,然后将该簇一分为二。之后选择其中一个簇继续进行划分,选择哪一个簇取决于对其划分是否可以最大程度降低SSE的值。上述基于SSE的划分过程不断重复,知道得到用户指定的簇数目为止。
二分k-均值算法伪代码
将所有点看成一个簇
当簇数目小于k时
对于每一个簇
计算总误差
在给定的簇上面进行k-均值聚类(k=2)
计算将该簇一分为二之后的总误差
选择是的误差最小的那个簇进行划分操作
代码参考链接:https://www.cnblogs.com/multhree/p/11279140.html
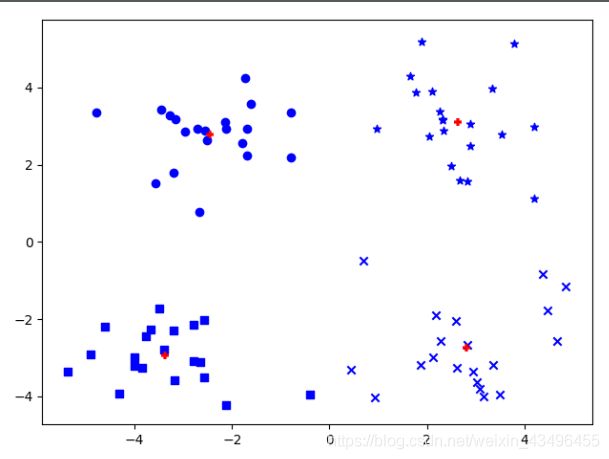
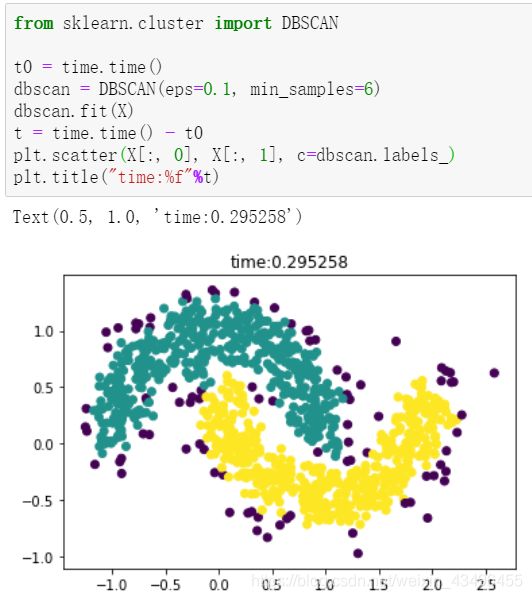
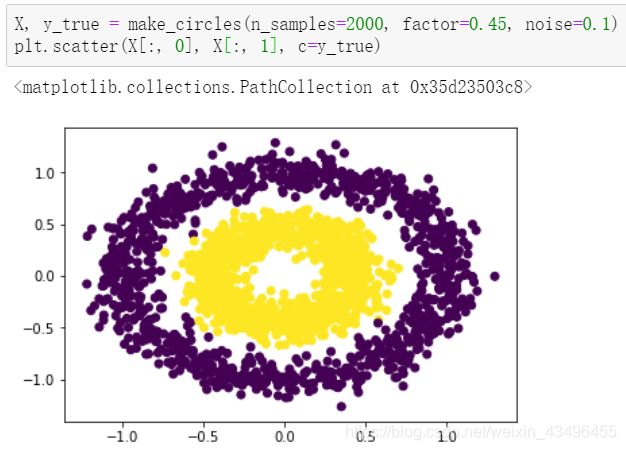
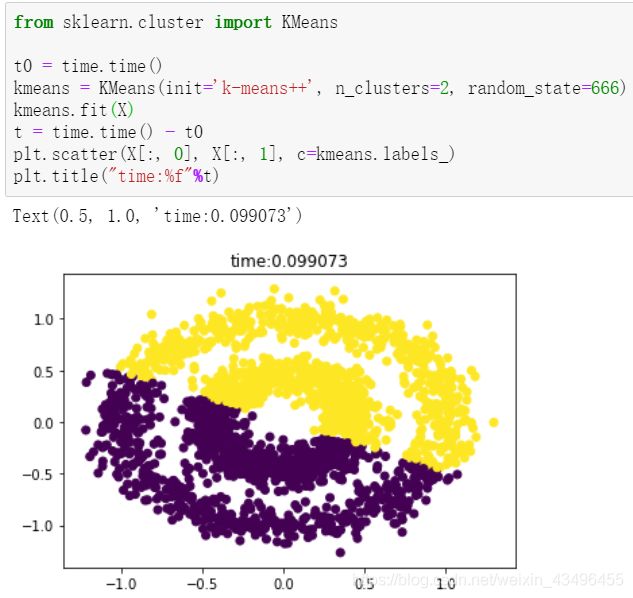
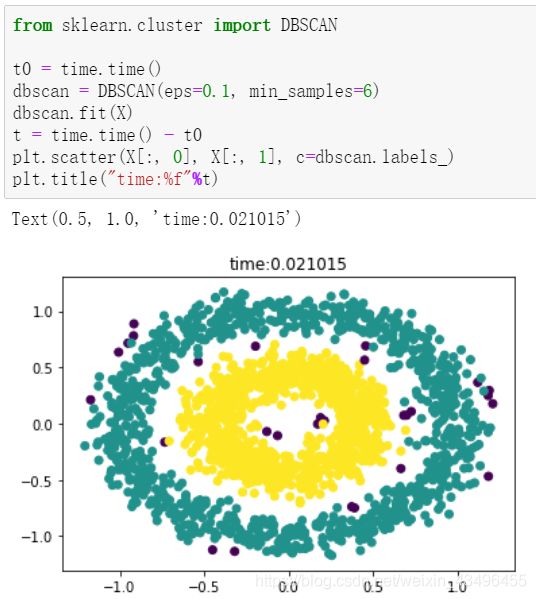
k-means与DBSCAN效果对比
k-means
DBSCAN
k-means
DBSCAN
K-Means++、Mini Bacth K-Means
因为K-Means算法对初次选取的类别中心敏感,不同的随机种子点得到的聚类结果会有不同。K-Means++算法便是在K-Means的基础上解决了这个问题,并不是一开始就把所有初始点找到,而是一个个地找初始点。这样达到的效果就是:初始的聚类中心之间的相互距离会比较远。
Mini Bacth K-Means算法:使用分批处理(Mini Bacth)的方法对数据点之间的距离进行计算。当样本点较多时,直接使用K-Means算法进行聚类的话,效率会比较慢,每一轮都有遍历计算所有的样本点到各个聚类中心的距离。
Mini Bacth K-Means的好处在于:不必使用所有的样本点进行距离计算、而是每次使用一个batch的样本点来进行K-means算法,进行计算到各个聚类中心的距离,从而得到新的聚类中心。batch_size的大小需自定义。
由于计算样本量的减少,相应的运行时间也会减少,但会带来准确度的降低。
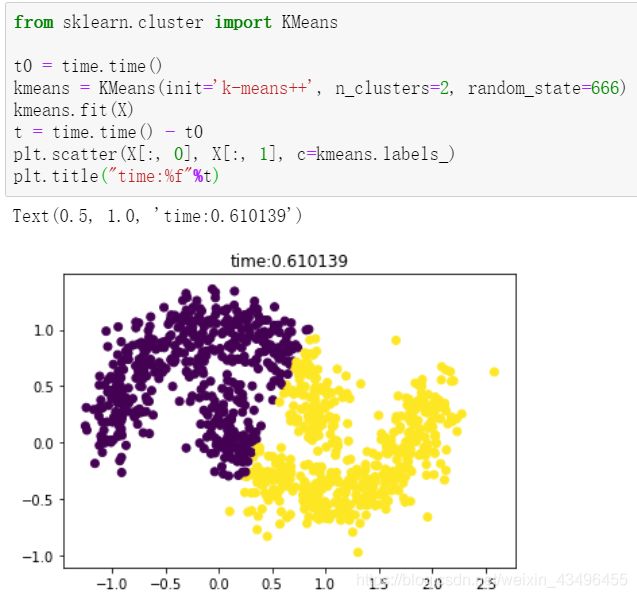
算法比较
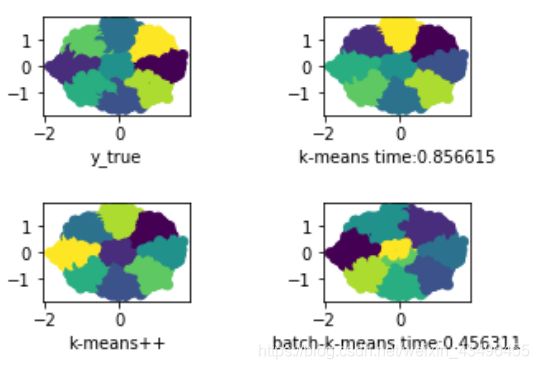
from sklearn.cluster import KMeans, MiniBatchKMeans
centers = [[1, 0], [-1, 0], [0, -1], [0, 1], [0, 0], [-0.7, -0.7], [-0.7, 0.7], [0.7, -0.7], [0.7, 0.7]]
X, y_true = make_blobs(n_samples=100000, centers=centers, cluster_std=0.18, random_state=0)
fig, axes = plt.subplots(2, 2)
axes[0, 0].scatter(X[:, 0], X[:, 1], c=y_true)
axes[0, 0].set_xlabel('y_true')
t0 = time.time()
y_pred1 = KMeans(init='random', n_clusters=9, random_state=666, n_init=1).fit_predict(X)
t_k_means = time.time() - t0
axes[0, 1].scatter(X[:, 0], X[:, 1], c=y_pred1)
axes[0, 1].set_xlabel('k-means time:%f'%t_k_means)
y_pred2 = KMeans(init='k-means++', n_clusters=9, random_state=666, n_init=1).fit_predict(X)
axes[1, 0].scatter(X[:, 0], X[:, 1], c=y_pred2)
axes[1, 0].set_xlabel('k-means++')
t0 = time.time()
y_pred3 = MiniBatchKMeans(init='random', n_clusters=9, batch_size=900, n_init=1, max_no_improvement=10).fit_predict(X)
t_batch_means = time.time() - t0
axes[1, 1].scatter(X[:, 0], X[:, 1], c=y_pred3)
axes[1, 1].set_xlabel('batch-k-means time:%f'%t_batch_means)
plt.subplots_adjust(left=0.2, bottom=0.2, right=0.8, top=0.8, hspace=0.9, wspace=0.9)
plt.show()
层次聚类与DBSCAN
代码参考:https://blog.csdn.net/sinat_18665801/article/details/90637272