(周志华《机器学习》西瓜书 小白Python学习笔记(八) ———— 第六章 支持向量机SVM LIBSVM实现以及结果解读(课后题6.2)
周志华《机器学习》西瓜书 小白Python学习笔记(八) ———— 第六章 支持向量机LIBSVM实现及结果解读(课后题6.2)
- LIBSVM介绍
- 安装LIBSVM(MAC系统)
- 数据格式和实验数据
- SVM实现
-
- 结果解读
LIBSVM介绍
LIBSVM在python版本提供了两个模块,svmutil.py为高层次版本,svm.py为低层次版本。在低层次版本svm.py中,没有对python内置库ctypes类型进行封装,而svmutil.py则提供了简单易用的函数可以直接使用;具体的使用可以参考下载的LIBSVM包中python文件夹中的readme.txt,其中有详尽的说明。这是几个常用的函数,具体用法可以参照readme.txt:
| 函数 | 功能 |
|---|---|
| svm_train() | 训练SVM模型 |
| svm_predict() | 预测测试数据的结果 |
| svm_read_problem() | 读取数据 |
| svm_load_model() | 加载SVM模型 |
| svm_save_model() | 保存SVM模型 |
| evaluations() | 检验预测结果 |
安装LIBSVM(MAC系统)
- 首先在网站http://www.csie.ntu.edu.tw/~cjlin/libsvm/oldfiles/下载LIBSVM包:libsvm.zip;
- 将LIBSVM包解压,并在终端中进入此文件夹(
cd path),之后输入make,回车执行; - 再进入文件夹中的
python文件夹(cd python),再输入make并执行; - 将
libsvm文件夹中生成的libsvm.so.2复制到python的lib文件夹下的site-packages文件夹中,并在site-packages文件夹中新建一个名叫libsvm的文件夹,并将下载的libsvm包中python文件夹里的三个*.py文件复制到新libsvm文件夹里; - 并在新
libsvm文件夹里新建一个__init.py文件,否则无法实现导入; - 之后在使用LIBSVM的时候直接:
import sys
sys.path.append('/Users/jiang/PycharmProjects/svmsvm/virtual_env/lib/python3.6/site-packages/libsvm')
from svm import *
from svmutil import *
数据格式和实验数据
LIBSVM的数据格式为:
第一列为标签;
第二列为索引(1)和第一个特征值;
第三列为索引(2)和第二个特征值;
…依此类推…
本次所用的西瓜数据集,转化为LIBSVM包需要的数据格式,如下,保存为data.txt:
1 1:.697 2:.46
1 1:.774 2:.376
1 1:.634 2:.264
1 1:.608 2:.318
1 1:.556 2:.215
1 1:.402999999999999 2:.237
1 1:.481 2:.149
1 1:.437 2:.211
0 1:.665999999999999 2:.091
0 1:.243 2:.267
0 1:.245 2:.057
0 1:.342999999999999 2:.099
0 1:.639 2:.161
0 1:.657 2:.198
0 1:.36 2:.37
0 1:.593 2:.042
0 1:.719 2:.103
SVM实现
数据读取
def processData(filename):
y, x = svm_read_problem(filename)
return y, x
训练SVM,注释中有一些参数的说明
def xiguasvm(x, y):
"""
svm_train(y, x [, 'options']) -> model | ACC | MSE
svm_train(prob, [, 'options']) -> model | ACC | MSE
svm_train(prob, param) -> model | ACC| MSE
Train an SVM model from data (y, x) or an svm_problem prob using
'options' or an svm_parameter param.
If '-v' is specified in 'options' (i.e., cross validation)
either accuracy (ACC) or mean-squared error (MSE) is returned.
'options':
-s svm_type : set type of SVM (default 0)
0 -- C-SVC
1 -- nu-SVC
2 -- one-class SVM
3 -- epsilon-SVR
4 -- nu-SVR
-t kernel_type : set type of kernel function (default 2)
0 -- linear: u'*v
1 -- polynomial: (gamma*u'*v + coef0)^degree
2 -- radial basis function: exp(-gamma*|u-v|^2)
3 -- sigmoid: tanh(gamma*u'*v + coef0)
4 -- precomputed kernel (kernel values in training_set_file)
-d degree : set degree in kernel function (default 3)
-g gamma : set gamma in kernel function (default 1/num_features)
-r coef0 : set coef0 in kernel function (default 0)
-c cost : set the parameter C of C-SVC, epsilon-SVR, and nu-SVR (default 1)
-n nu : set the parameter nu of nu-SVC, one-class SVM, and nu-SVR (default 0.5)
-p epsilon : set the epsilon in loss function of epsilon-SVR (default 0.1)
-m cachesize : set cache memory size in MB (default 100)
-e epsilon : set tolerance of termination criterion (default 0.001)
-h shrinking : whether to use the shrinking heuristics, 0 or 1 (default 1)
-b probability_estimates : whether to train a SVC or SVR model for probability estimates, 0 or 1 (default 0)
-wi weight : set the parameter C of class i to weight*C, for C-SVC (default 1)
-v n: n-fold cross validation mode
-q : quiet mode (no outputs)
"""
model1 = svm_train(y, x,'-t 0') # 线性
model2 = svm_train(y, x,'-t 2') # 高斯
svm_save_model('Linear.model', model1)
svm_save_model('Gauss.model', model2)
主函数:
if __name__ == '__main__':
y, x = processData(data.txt)
xiguasvm(x, y)
完整程序:
import sys
sys.path.append('/Users/haoranjiang/PycharmProjects/svmsvm/virtual_env/lib/python3.6/site-packages/libsvm')
from svm import *
from svmutil import *
def processData(filename):
y, x = svm_read_problem(filename)
return y, x
def xiguasvm(x, y):
"""
svm_train(y, x [, 'options']) -> model | ACC | MSE
svm_train(prob, [, 'options']) -> model | ACC | MSE
svm_train(prob, param) -> model | ACC| MSE
Train an SVM model from data (y, x) or an svm_problem prob using
'options' or an svm_parameter param.
If '-v' is specified in 'options' (i.e., cross validation)
either accuracy (ACC) or mean-squared error (MSE) is returned.
'options':
-s svm_type : set type of SVM (default 0)
0 -- C-SVC
1 -- nu-SVC
2 -- one-class SVM
3 -- epsilon-SVR
4 -- nu-SVR
-t kernel_type : set type of kernel function (default 2)
0 -- linear: u'*v
1 -- polynomial: (gamma*u'*v + coef0)^degree
2 -- radial basis function: exp(-gamma*|u-v|^2)
3 -- sigmoid: tanh(gamma*u'*v + coef0)
4 -- precomputed kernel (kernel values in training_set_file)
-d degree : set degree in kernel function (default 3)
-g gamma : set gamma in kernel function (default 1/num_features)
-r coef0 : set coef0 in kernel function (default 0)
-c cost : set the parameter C of C-SVC, epsilon-SVR, and nu-SVR (default 1)
-n nu : set the parameter nu of nu-SVC, one-class SVM, and nu-SVR (default 0.5)
-p epsilon : set the epsilon in loss function of epsilon-SVR (default 0.1)
-m cachesize : set cache memory size in MB (default 100)
-e epsilon : set tolerance of termination criterion (default 0.001)
-h shrinking : whether to use the shrinking heuristics, 0 or 1 (default 1)
-b probability_estimates : whether to train a SVC or SVR model for probability estimates, 0 or 1 (default 0)
-wi weight : set the parameter C of class i to weight*C, for C-SVC (default 1)
-v n: n-fold cross validation mode
-q : quiet mode (no outputs)
"""
model1 = svm_train(y, x, '-t 0') # 线性核
model2 = svm_train(y, x, '-t 2') # 高斯核
svm_save_model('Linear.model', model1)
svm_save_model('Gauss.model', model2)
if __name__ == '__main__':
y, x = processData(data.txt)
xiguasvm(x, y)
实验结果:

并生成了两个模型的model文件,打开后分别为:
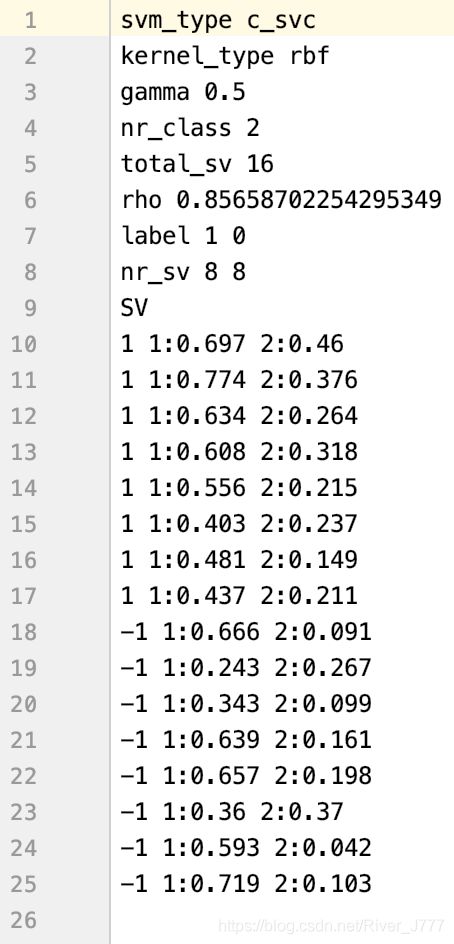
线性核函数:

结果解读
| #iter | 迭代次数 |
| nu | 与前面的操作参数 -n nu 相同 |
| obj | SVM 文件转换为的二次规划求解得到的最小值 |
| rho | 为判决函数的常数项 b |
| nSV | 为支持向量个数 |
| nBSV | 边界上的支持向量个数 |
| Total nSV | 为支持向量总个数 |
训练后的模型保存为文件 *.model ,用记事本打开其内容如下:
| svm_type | 训练所采用的 svm 类型 |
| kernel_type | 训练采用的核函数类型 |
| gamma | 设置核函数中的 g ,默认值为 1 / k 1/ k 1/k |
| nr_class | 分类时的类别数,此处为两分类问题 |
| total_sv | 总共的支持向量个数 |
| rho | 决策函数中的常数项 b |
| label | 类别标签 |
| nr_sv | 各类别标签对应的支持向量个数 |
| SV | 以下为支持向量 |