利用weka进行数据挖掘——基于Apriori算法的关联规则挖掘实例
文章目录
-
- 1. weka安装
- 2. 先分析一个Apriori算法的关联规则挖掘实例
- 3. 利用weka进行数据挖掘
-
- 3.1 将数据转为ARFF格式
- 3.2 利用weka进行分析
- 4. 参考文章
首先,如果不熟悉weka的使用的话,可以从我的git仓库里面拉取一下weka的相关教程,仓库里面还有包含此次实例的所有资源
1. weka安装
我们可以在weka的官网上下载weka软件:weka官网
如果下载速度慢的话也可以直接从我的git仓库里面拉取这个软件,软件是win64位的weka-3-8-6
然后找到对应版本:
点击就可以开始下载了,一路安装就好了,因为weka是基于Java开发,所以如果你的电脑没有Java环境的话,可能在安装的过程中会提示你安装Java,选择安装即可。这里我们需要记住一下我们安装的路径,因为我们后面还需要进入到安装目录中来
2. 先分析一个Apriori算法的关联规则挖掘实例
先打开这个文档,这个文档里面有一个基于Apriori算法的关联规则挖掘实例
可以先看一遍,看能不能用笔自己算出来,计算的步骤可以看下笔者的上一篇文章:数据挖掘十大算法之Apriori算法
只有理解了Apriori算法,才能够自己编写代码进行实践,weka只是工具,帮助我们快速分析的工具,在分析之前我们需要先知道分析的步骤
这里我们需要对下表中数据进行数据挖掘,寻找这些疾病之间的关系:
| 病人记录号 | 疾病名称 |
|---|---|
| 1 | 心力衰竭、其他疾病 |
| 2 | 心力衰竭 |
| 3 | 心力衰竭、尿毒症 |
| 4 | 心力衰竭、肾功能衰竭、糖尿病、尿毒症 |
| 5 | 心力衰竭、肾功能衰竭、糖尿病、尿毒症 |
| 6 | 糖尿病 |
| 7 | 糖尿病、心力衰竭、其他疾病 |
| 8 | 糖尿病、尿毒症 |
| 9 | 糖尿病 |
| 10 | 糖尿病、肾功能衰竭、尿毒症 |
| 11 | 糖尿病 |
| 12 | 心力衰竭、肾功能衰竭、糖尿病、尿毒症 |
| 13 | 心力衰竭、肾功能衰竭、糖尿病、尿毒症 |
| 14 | 肾功能衰竭 |
| 15 | 肾功能衰竭、其他疾病 |
| 16 | 肾功能衰竭、糖尿病 |
| 17 | 肾功能衰竭、尿毒症 |
| 18 | 肾功能衰竭 |
| 19 | 尿毒症、糖尿病、肾功能衰竭 |
| 20 | 尿毒症、肾功能衰竭 |
| 21 | 尿毒症 |
| 22 | 心力衰竭、肾功能衰竭、糖尿病、尿毒症 |
| 23 | 心力衰竭、肾功能衰竭、糖尿病、尿毒症 |
| 24 | 心力衰竭、肾功能衰竭、糖尿病、尿毒症 |
| 25 | 心力衰竭、肾功能衰竭、糖尿病、尿毒症 |
我们先回顾一下进行关联分析的步骤:
Step1:令K = 1 ,计算单个项目的支持度,并筛选出频繁1项集(大于最小支持度 )
Step2:(从K=2开始)根据K-1项的频繁项目集生成候选K项目集,并进行
预剪枝Step3:由候选K项目集生成频繁K项集(筛选出满足最小支持度的k项集)
重复步骤2和3,直到无法筛选出满足最小支持度的集合。(第一阶段结束)
Step4:将获得的最终的频繁K项集,依次取出。同时计算该次取出的这个K项集 的所有真子集,然后以排列组合的方式形成关联规则,并计算规则的置信度以及提升度,将符合要求的关联规则生成提出。(算法结束)
我们可以从上表中知道这些病在事物中出现的次数为:
| 心力衰竭 | 尿毒症 | 肾功能衰竭 | 糖尿病 | 其他疾病 |
|---|---|---|---|---|
| 12 | 15 | 16 | 16 | 3 |
我们先生成候选1项集,并计算其支持度,支持度(sup(x)) = 某个项集X在事物集中出现的次数 / 事物集中记录的总个数
| 项集 | 支持度 |
|---|---|
| 心力衰竭 | 12/25 = 0.48 |
| 尿毒症 | 15/25 = 0.6 |
| 肾功能衰竭 | 16 / 25 = 0.64 |
| 糖尿病 | 16 / 25 = 0.64 |
| 其他疾病 | 3 / 25 = 0.12 |
这里我们规定
最小支持度(Support) = 40%,并筛选出频繁一项集(将不满足min_support的项集去除掉)
| 项集 | 支持度 |
|---|---|
| 心力衰竭 | 12/25 = 0.48 |
| 尿毒症 | 15/25 = 0.6 |
| 肾功能衰竭 | 16 / 25 = 0.64 |
| 糖尿病 | 16 / 25 = 0.64 |
现在我们有筛选出的频繁一项集经过排列组合生成候选二项集,共有C42 = 6个候选二项集
同样我们可以算出每个项集的支持度(过程与上面一样,这里不再赘述):
| 项集 | 支持度 |
|---|---|
| 心力衰竭、糖尿病 | 36% |
| 心力衰竭、尿毒症 | 36% |
| 心力衰竭、肾功能衰竭 | 32% |
| 糖尿病、尿毒症 | 44% |
| 糖尿病、肾功能衰竭 | 44% |
| 尿毒症、肾功能衰竭 | 48% |
筛选出频繁二项集
| 项集 | 支持度 |
|---|---|
| 糖尿病、尿毒症 | 44% |
| 糖尿病、肾功能衰竭 | 44% |
| 尿毒症、肾功能衰竭 | 48% |
当然可以先进行
预剪枝,但是这里预剪枝排除不了候选项集。同理我们可以求出频繁三项集
| 项集 | 支持度 |
|---|---|
| 糖尿病、尿毒症、肾功能衰竭 | 40% |
最后一步我们需要求出不同项集之间的支持度(Support)和置信度(Confidence):
(支持度)Support=(AUB)count/n 即A和B同时出现的次数之和与事务数n之比。
(置信度)Confidence=(AUB)count/Acount 即A和B同时出现的次数之和与A出现次数之比。
我们可以计算出:
- 糖尿病→尿毒症 Confidence=11/16=68.7%
- 糖尿病→肾功能衰竭 Confidence=11/16=68.7%
- 糖尿病→(尿毒症∩肾功能衰竭) Confidence=10/16=62.5%
- (尿毒症∩糖尿病)→肾功能衰竭 Confidence=10/11=90%
为方便起见,将糖尿病标记为T,尿毒症标记为N,肾功能衰竭标记为S。总结出部分关联规则。
| 关联规则 | 支持度 | 置信度 |
|---|---|---|
| T→N | 44% | 68.7% |
| T→S | 44% | 68.7% |
| T→(N∩S) | 40% | 62.5% |
| (T∩N)→S | 40% | 90.00% |
根据以上关联规则可得出一下结论:
(1)糖尿病、尿毒症、肾功能衰竭三种疾病之间有一定的关联关系。
(2)对于同时患有糖尿病和尿毒症的44%的疾病人群而言,有68.7%的糖尿病患者会并发尿毒症。
(3)对于同时患有糖尿病和肾功能衰竭的44%的疾病人群而言,有68.7%的糖尿病患者会并发肾功能衰竭。
(4)有40%的患者同时患有糖尿病、尿毒症、肾功能衰竭,其中有62.5%的糖尿病患者会并发尿毒症和肾功能衰竭,有90%的糖尿病和尿毒症患者会并发肾功能衰竭。
3. 利用weka进行数据挖掘
如果数据量很大,例如数据库里有几十万条数据,那我们手算肯定是不行的,所以我们就需要借助weka这款软件来帮助我的进行数据分析和挖掘。weka的详细用法在上面已经给了下载的链接,可以看一下,接下来我们开始实战
首先我们要知道:WEKA存储数据的格式是ARFF(Attribute-Relation File Format)文件,这是一种ASCII文本文件
我们可以在weka安装目录的data目录下找到一些默认的ARFF格式的文件
使用WEKA作数据挖掘,面临的第一个问题往往是我们的数据不是ARFF格式的。
幸好,WEKA还提供了对CSV文件的支持,而这种格式是被很多其他软件所支持的。此外,WEKA还提供了通过JDBC访问数据库的功能。
3.1 将数据转为ARFF格式
首先我先以Excel为例,说明如何获得CSV文件。然后我们将知道CSV文件如何转化成ARFF文件,毕竟后者才是WEKA支持得最好的文件格式。面对一个ARFF文件,我们仍有一些预处理要做,才能进行挖掘任务。
Excel的XLS文件可以让多个二维表格放到不同的工作表(Sheet)中,我们只能把每个工作表存成不同的CSV文件。打开一个XLS文件并切换到需要转换的工作表,另存为CSV类型,点“确定”、“是”忽略提示即可完成操作。
ARFF是表示属性关系文件格式的首字母缩略词。它是使用标题的CSV文件格式的扩展,提供有关列中数据类型的元数据。
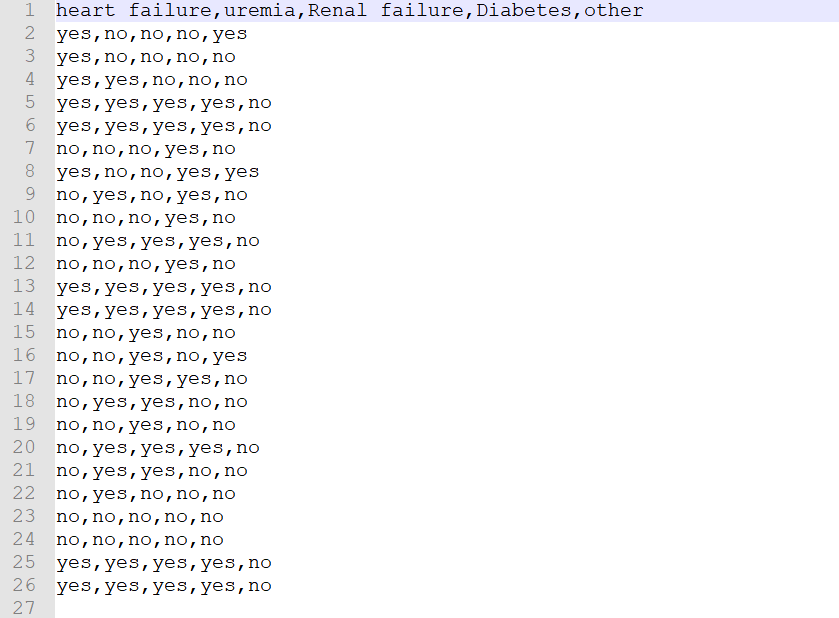
我们以上面我们手算过的关联案例来演示,首先我们需要把数据整理到Excel表中,后期我们可以通过Java编写程序来自动化完成这些步骤。这里我们将患病标记为yes,不患病标记为no,得到整理好的Excel表,此表也可以从我提供的git仓库中拉取到
我们再填Excel表的时候要注意的是:
- 不要包含中文
- 不要用数字做标识,要用yes/no做标识
当然还有很多值得注意的点,请看参考文章的第一篇
填好的Excel长这样,这里我用行数代替患者编号了,因为编号没有什么意义
我们将此表另存为,保存格式选择
CSV格式,不用管提示,一路确认就好
打开我们的CSV格式的文件,长这样
现在我们需要进一步转换格式:
.csv -> .arff
这里有两种方式,将CSV转换为ARFF最迅捷的办法是使用WEKA所带的命令行工具。
- 运行WEKA的主程序,出现GUI后可以点击下方按钮进入相应的模块。我们点击进入“Simple CLI”模块提供的命令行功能。在新窗口的最下方(上方是不能写字的)输入框写上
java weka.core.converters.CSVLoader 文件路径加上文件名.csv > 文件路径加上文件名.arff
- 在WEKA 3.5中提供了一个“Arff Viewer”模块,我们可以用它打开一个CSV文件将进行浏览,然后另存为ARFF文件。
进入“Exploer”模块,从上方的按钮中打开CSV文件然后另存为ARFF文件亦可
我这里采取命令行的形式,程序员吗,肯定还是喜欢用命令行一点
要转换的文件要带上地址,输出的文件也要带上,例如我的命令是:
java weka.core.converters.CSVLoader C:\Users\hw\Desktop\demo1001.csv > C:\Users\hw\Desktop\demo1001.arff
查看一下我们转换好的reff格式的文件,长这样:
3.2 利用weka进行分析
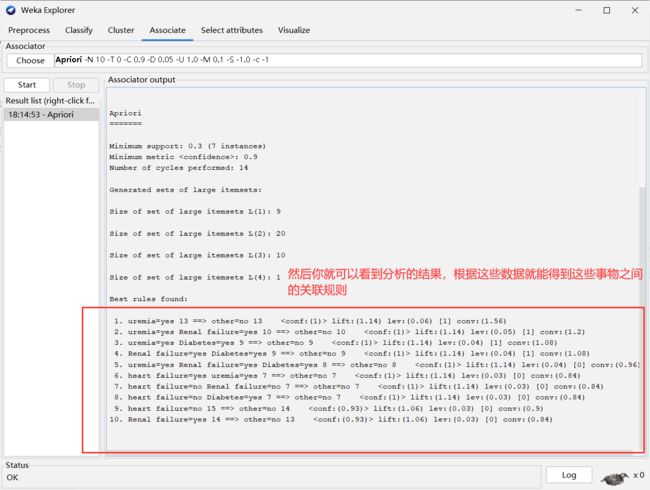
接下来我们就利用weka对这些数据进行分析,来查找这些数据之间的关系
那些面板的具体含义看一下我仓库里面WEKA中文详细教程ppt,写的很清楚
这里直接上手
当然weka的功能非常强大,我们这里仅仅这是做一个演示,来加深我们对Aprior算法的理解
上面提供的git仓库里面也有weka的API和一个demo,读者可以编写代码来对数据库的数据进行挖掘
4. 参考文章
- weka的基本使用
- 如何在Weka中加载CSV机器学习数据
- 【数据挖掘 data mining】 Weka在数据挖掘中的运用(双语字幕)
- 各大大学的数据挖掘、机器学习教学课件