【建模算法】基于遗传算法求解TSP问题(matlab求解)
【建模算法】基于遗传算法求解TSP问题(matlab求解)
TSP (traveling salesman problem,旅行商问题)是典型的NP完全问题,即其最坏情况下的时间复杂度随着问题规模的增大按指数方式增长,到目前为止还未找到一个多项式时间的有效算法。
一、问题描述
本案例以31个城市为例,假定31个城市的位置坐标如表1所列。寻找出一条最短的遍历31个城市的路径。
| 城市编号 | X坐标 | Y坐标 | 城市编号 | X坐标 | Y坐标 |
|---|---|---|---|---|---|
| 1 | 1.304 | 2.312 | 17 | 3.918 | 2.179 |
| 2 | 3.639 | 1.315 | 18 | 4.061 | 2.37 |
| 3 | 4.177 | 2.244 | 19 | 3.78 | 2.212 |
| 4 | 3.712 | 1.399 | 20 | 3.676 | 2.578 |
| 5 | 3.488 | 1.535 | 21 | 4.029 | 2.838 |
| 6 | 3.326 | 1.556 | 22 | 4.263 | 2.931 |
| 7 | 3.238 | 1.229 | 23 | 3.429 | 1.908 |
| 8 | 4.196 | 1.044 | 24 | 3.507 | 2.376 |
| 9 | 4.312 | 0.79 | 25 | 3.394 | 2.643 |
| 10 | 4.386 | 0.57 | 26 | 3.439 | 3.201 |
| 11 | 3.007 | 1.97 | 27 | 2.935 | 3.24 |
| 12 | 2.562 | 1.756 | 28 | 3.14 | 3.55 |
| 13 | 2.788 | 1.491 | 29 | 2.545 | 2.357 |
| 14 | 2.381 | 1.676 | 30 | 2.778 | 2.826 |
| 15 | 1.332 | 0.695 | 31 | 2.37 | 2.975 |
| 16 | 3.715 | 1.678 |
二、解决思路及步骤
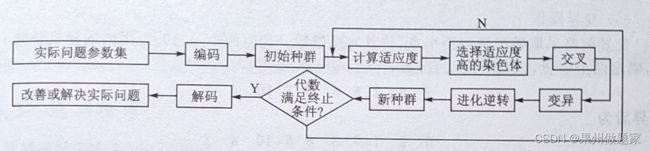
1、算法流程图
2、遗传算法实现及部分代码
(1)编码
采用整数排列编码方法。对于 n 个城市的 TSP 问题,染色体分为 n 段,其中每一段为对应城市的编号,如对 10 个城市的 TSP 问题 {1,2,3,4,5,6,7,8,9,10},则 | 1 | 10 | 2 | 4 | 5 | 6 | 8 | 7 | 9 | 3 就是一个合法的染色体。
(2)种群初始化
在完成染色体编码以后,必须产生一个初始种群作为起始解,所以首先需要决定初始化种群的数目。初始化种群的数目一般根据经验得到,一般情况下种群的数量视城市规模的大小而确定,其取值在 50~500 之间浮动。种群初始化函数 InitPop 代码为:
function Chrom = InitPop(NIND, N)
%% 初始化种群
% 输入:
% NIND:种群大小
% N:个体染色体长度(这里为城市的个数)
% 输出:
% 初始种群
Chrom = zeros(NIND, N); %用于存储种群
for i = 1 : NIND
Chrom(i, :) = randperm(N); %随机生成初始种群
end
(3)适应度函数

求种群个体的适应度函数 Fitness 的代码如下:
function FitnV = Fitness(len)
%% 适应度函数
% 输入:
% len: 个体的长度(TSP的距离)
% 输出:
% FitnV:个体的适应度值
FitnV = 1 ./ len;
(4)选择操作
选择操作即从旧群体中以一定概率选择个体到新群体中,个体被选中的概率跟适应度值有关,个体适应度值越大,被选中的概率越大。
选择操作函数 Select 的代码为:
function SelCh = Select(Chrom, FitnV, GGAP)
%% 选择操作
% 输入:
% Chrom:种群
% FitnV:适应度值
% GGAP:选择概率
% 输出:
% SelCh:被选择的个体
NIND = size(Chrom, 1);
NSel = max(floor(NIND * GGAP + .5), 2);
ChrIx = Sus(FitnV, NSel);
SelCh = Chrom(ChrIx, :);
其中,函数 Sus 的代码为:
function NewChrIx = Sus(FitnV, Nsel)
%% Select(选择)调用
% 输入:
% FitnV:个体的适应度值
% Nsel:被选择个体的数目
% 输出:
% NewChrIx:被选择个体的索引号
[Nind, ~] = size(FitnV);
cumfit = cumsum(FitnV); %cumsum:向量累计和
trials = cumfit(Nind) / Nsel * (rand + (0: Nsel - 1)');
Mf = cumfit(:, ones(1, Nsel));
Mt = trials(:, ones(1, Nind))';
[NewChrIx, ~] = find(Mt < Mf & [zeros(1, Nsel); Mf(1: Nind - 1, :)] <= Mt);
[~, shuf] = sort(rand(Nsel, 1));
NewChrIx = NewChrIx(shuf);
(5)交叉操作
采用部分映射杂交,确定交叉操作的父代,将父代样本两两分组,每组重复以下过程(假定城市数为10):
1.产生 [1,10] 区间内的随机整数 r1 和 r2 ,确定两个位置,对两位置的中间数据进行交叉
2.交叉后,同一个个体中有重复的城市编号,不重复的数字保留,有冲突的数字采用部分映射的方法消除冲突,即利用中间段的对应关系进行映射。
交叉操作函数 Recombin 的代码为:
function SelCh = Recombin(SelCh, Pc)
%% 交叉操作
% 输入:
% SelCh:被选择的个体
% Pc:交叉概率
% 输出:
% SelCh:交叉后的个体
NSel = size(SelCh, 1);
for i = 1 : 2 : NSel - mod(NSel, 2)
if Pc >= rand %交叉概率Pc
[SelCh(i, :), SelCh(i+1, :)] = intercross(SelCh(i, :), SelCh(i+1, :));
end
end
其中,函数 intercross 的代码为:
function [a, b] = intercross(a, b)
%% Recombin(交叉)调用
% 输入:
% a和b为两个待交叉的个体
% 输出:
% a和b为交叉后得到的两个个体
L = length(a);
r1 = randsrc(1, 1, [1: L]); % 随机生成1到L的整数
r2 = randsrc(1, 1, [1: L]);
if r1 ~= r2
a0 = a; b0 = b;
s = min([r1, r2]);
e = max([r1, r2]);
for i = s: e
a1 = a; b1 = b;
a(i) = b0(i); % 交换r1到r2之间的数
b(i) = a0(i);
x = find(a == a(i)); % 查找交换后相同值的位置
y = find(b == b(i));
i1 = x(x ~= i);
i2 = y(y ~= i);
if ~ isempty(i1) % x值与i值不相等
a(i1) = a1(i);
end
if ~isempty(i2)
b(i2) = b1(i);
end
end
end
(6)变异操作
变异策略采取随机选取两个点,将其对换位置。产生两个 [1,10] 范围内的随机整数 r1 和 r2 ,确定两个位置,将其对换位置。
变异操作函数 Mutate 的代码如下:
function SelCh = Mutate(SelCh, Pm)
%% 变异操作
% 输入:
% SelCh:被选择的个体
% Pm:变异概率
% 输出:
% SelCh:变异后的个体
[NSel, L] = size(SelCh);
for i = 1: NSel
if Pm >= rand
R = randperm(L); % 生成1到L没有重复元素的随机整数行向量
SelCh(i, R(1: 2)) = SelCh(i, R(2: -1: 1));
end
end
(7)进化逆转操作
为改善遗传算法的局部搜索能力,在选择、交叉、变异之后引进连续多次的进化逆转操作。这里的 “进化” 是指逆转算子的单方向性,即只有经逆转后,适应度值有提高的才接受下来,否则逆转无效。产生两个 [1,10] 区间内的随机整数 r1 和 r2,确定两个位置,将其对换位置。
对每个个体进行交叉变异,然后代入适应度函数进行评估,x 选择出适应值大的个体进行下一代的交叉和变异以及进化逆转操作。循环操作:判断是否满足设定的最大遗传代数 MAXGEN,不满足则跳入适应度值的计算;否则,结束遗传操作。
进行逆转函数 Reverse 的代码为:
function SelCh = Reverse(SelCh, D)
%% 进化逆转函数
% 输入:
% SelCh:被选择的个体
% D:各城市的距离矩阵
% 输出:
% SelCh:进化逆转后的个体
[row, col] = size(SelCh);
ObjV = PathLength(D, SelCh); % 计算路线长度
SelCh1 = SelCh;
for i = 1: row
r1 = randsrc(1, 1, [1: col]);
r2 = randsrc(1, 1, [1: col]);
mininverse = min([r1, r2]);
maxinverse = max([r1, r2]);
SelCh1(i, mininverse: maxinverse) = SelCh1(i, maxinverse: -1: mininverse);
end
ObjV1 = PathLength(D, SelCh1); % 计算路线长度
index = ObjV1 < ObjV;
SelCh(index, :) = SelCh1(index, :);
(8)画路线轨迹图
画出所给路线的轨迹图函数DrawPath的代码:
function DrawPath(Chrom, X)
%% 画路线图函数
% 输入:
% Chrom:待画路线
% X:各城市的坐标位置
R = [Chrom(1, :) Chrom(1, 1)]; % 一个随机解(个体)
figure;
hold on
plot(X(:, 1), X(:, 2), 'o', 'color', [0.5, 0.5, 0.5])
plot(X(Chrom(1, 1), 1), X(Chrom(1, 1), 2), 'rv', 'MarkerSize', 20)
for i = 1: size(X, 1)
text(X(i, 1) + 0.05, X(i, 2) + 0.05, num2str(i), 'color', [1, 0, 0]);
end
A = X(R, :);
row = size(A, 1);
for i = 2: row
[arrowx, arrowy] = dsxy2figxy(gca, A(i - 1: i, 1), A(i - 1: i, 2)); % 坐标转换
annotation('textarrow', arrowx, arrowy, 'HeadWidth', 8, 'color', [0, 0, 1]);
end
hold off
xlabel('横坐标')
ylabel('纵坐标')
title('轨迹图')
box on
3、遗传算法主函数
clear,clc;
close all
data=importdata('p_xy.xlsx'); %读取数据excel表
X=data.data.Sheet1(:,2:3); %各城市坐标
NIND = 300; %种群大小
MAXGEN = 1000;
Pc = 0.9; %交叉概率
Pm = 0.05; % 变异概率
GGAP = 0.9; % 代沟
D = Distance(X); % 生成距离矩阵
N = size(D, 1); % (31 * 31)
%% 初始化种群
Chrom = InitPop(NIND, N);
%% 在二维图上画出所有坐标点
% figure
% plot(X(:, 1), X(:, 2), 'o');
%% 画出随机解的线路图
DrawPath(Chrom(1, :), X);
%% 输出随机解的线路和总距离
disp('初始种群中的一个随机值:')
OutputPath(Chrom(1, :));
Rlength = PathLength(D, Chrom(1, :));
disp(['总距离:', num2str(Rlength)]);
disp('~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~');
%% 优化
gen = 0;
figure;
hold on; box on
xlim([0, MAXGEN])
title('优化过程')
xlabel('代数')
ylabel('最优值')
ObjV = PathLength(D, Chrom); %计算路线长度
preObjV = min(ObjV);
while gen < MAXGEN
%% 计算适应度
ObjV = PathLength(D, Chrom); %计算路线长度
line([gen - 1, gen], [preObjV, min(ObjV)]);
preObjV = min(ObjV);
FitnV = Fitness(ObjV);
%% 选择
SelCh = Select(Chrom, FitnV, GGAP);
%% 交叉操作
SelCh = Recombin(SelCh, Pc);
%% 变异
SelCh = Mutate(SelCh, Pm);
%% 逆转操作
SelCh = Reverse(SelCh, D);
%% 重插入子代的新种群
Chrom = Reins(Chrom, SelCh, ObjV);
%% 更新迭代次数
gen = gen + 1;
end
%% 画出最优解的路线图
ObjV = PathLength(D, Chrom); %计算路线长度
[minObjV, minInd] = min(ObjV);
DrawPath(Chrom(minInd(1), :), X)
%% 输出最优解的路线和总距离
disp('最优解:')
p = OutputPath(Chrom(minInd(1), :));
disp(['总距离:', num2str(ObjV(minInd(1)))]);
disp('-------------------------------------------------------------')
主函数里的部分函数列表如下:
function DrawPath:输出路线函数
function PathLength:计算各个体的路径长度
function Distance:计算两两城市之间的距离
function OutputPath:输出路线函数
function Reins:重插入子代的新种群
function dsxy2figxy:坐标转换
三、结果分析与改进
1、结果分析
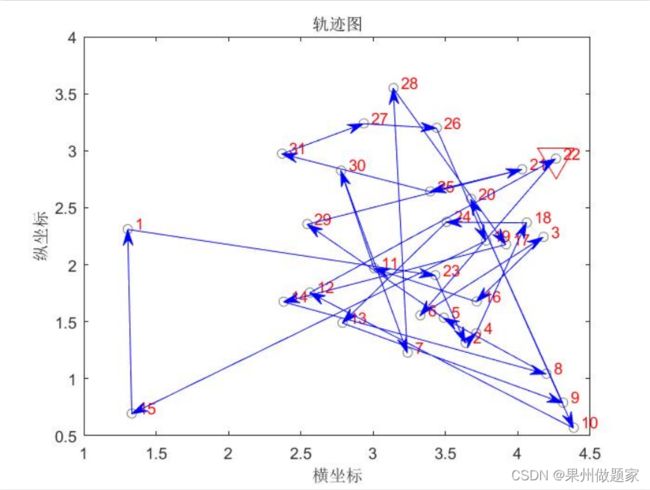
优化前的一个随机路线轨迹图
初始种群中的一个随机值:
22->6->3->16->11->30->7->28->17->14->8->5->29->21->25->31->27->26->19->15->1->23->2->4->18->24->13->9->20->10->12->22
总距离:41.9051
优化后的路线图:
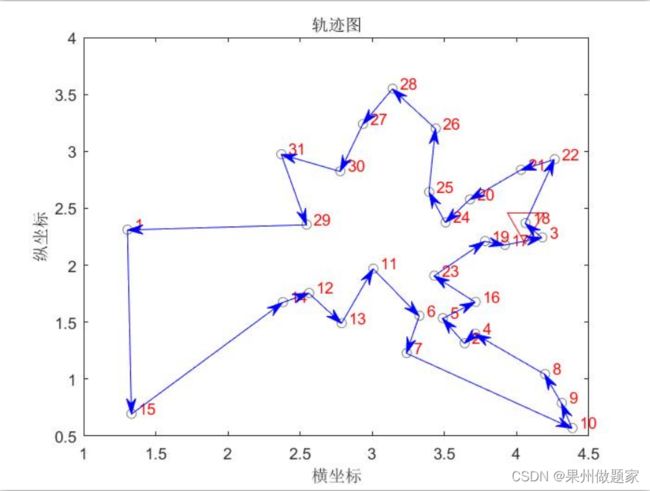
最优解:
18->22->21->20->24->25->26->28->27->30->31->29->1->15->14->12->13->11->6->7->10->9->8->4->2->5->16->23->19->17->3->18
总距离:15.4729
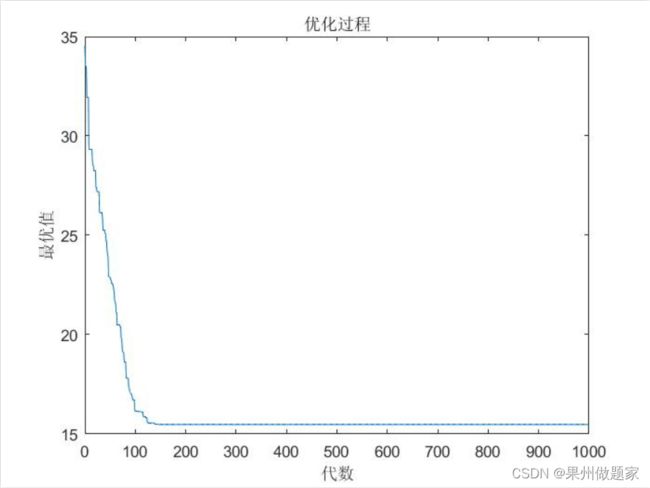
由进化图可以看出,优化前后路径长度得到很大改进,120代以后路径长度已经保持不变了,可以认为已经是最优解了,总距离由优化前的 41.9051 变为 15.4729,减为原来的36.92%。
2、遗传算法的改进
(1)使用精英策略
子代种群中的最优个体永远不会比父代最优的个体差,这样使得父代的好的个体不至于由于交叉或者变异操作而丢失。
(2)使用进化逆转操作
在本文的编码中,每一个染色体即对应一个 TSP 环游,如果染色体码串的顺序发生变化,则环游路径也随之改变。因此,TSP 问题解的关键地方就是码串的顺序。对照文中的交叉算子,可以发现,纵使两个亲代完全相同,通过交叉,仍然会产生不同于亲代的子代,且子代的码串排列顺序与亲代有较大的差异。交叉算子的这种变异效果所起的作用有两个方面,一方面它能起到维持群体内一定的多样性的作用,避免陷入局部最优解;但是另一方面,它却不利于子代继承亲代的较多信息,特别是当进化过程进入到后期,群体空间中充斥着大量的高适应度个体,交叉操作对亲代的较优基因破坏很大,使子代难以继承到亲代的优良基因,从而使交叉算子的搜索能力大大降低。
(3)算法的局限性
当问题规模n比较小时,得到的一般都是最优解;当规模比较大时,一般只能得到近似解。这时可以通过增大种群大小和增加最大遗传代数使得优化值更接近最优解。
完整源代码数据
链接:https://download.csdn.net/download/baidu/85223180