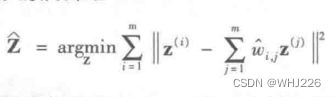猿创征文|机器学习实战(9)——降维
目录
1 主成分
2 低维度投影
3 方差解释率
4 选择正确数量的维度
5 PCA压缩
6 增量PCA
7 核主成分分析
8 选择核函数和调整超参数
9 局部线性嵌入
10 其他降维技巧
数据降维会丢失一些信息(好比压缩图像带来的效果一样),所以,它虽然能够加速训练,但是也会轻微降低系统性能。
我们简要说一下降维的两种主要方法:投影和流形学习。投影:高维空间的所有训练实例实际上受一个低得多的低维子空间所影响,将训练实例投影到该子空间就是投影。但许多情况下,子空间可能是弯曲的或转动的,就引入了流形学习。流形假设(流形假说)认为大多数现实世界的高维度数据集存在一个低维度的流形来重新表示。我们可以把瑞士卷看做一个二维流形的例子。更概括的说,d维流形就是n维空间的一部分(d 主成分分析(PCA)是迄今最流行的降维算法。它能识别出最接近数据的超平面,然后将数据投影该平面。同时该超平面也保留了数据最大差异性。 常规模块的导入以及图像可视化的设置: 主成分分析(PCA)可以在训练集中识别出哪条轴(一维超平面)对差异性的贡献度最高。即下图中由实线表示的轴。同时它也找出了第二条轴,它对剩余差异性的贡献度最高,与第一条轴垂直。 定义第i条轴的单位向量就叫作第i个主成分。上图中,第一个主成分是c1,第二个主成分是c2。前两个主成分是平面里正交的箭头所示,第三个主成分则垂直于平面。 那么我们该怎么找到训练集的主成分呢?我们有一种标准矩阵分解技术,叫作奇异值分解(SVD)。它可以将训练集矩阵X分解成三个矩阵的点积 下面的代码使用NumPy的svd()函数来获取训练集中所有主成分,并提取前两个: 一旦确定了所有主成分,就可以将数据集投影到由前d个主成分定义的超平面上,从而将数据集的维度降低到d维,这个超平面的选择,能确保投影保留尽可能多的差异性。 要将训练集投影到超平面上,简单地计算训练集矩阵X和矩阵 下面的代码将训练集投影到由前两个主成分定义的平面上: Scikit-Learn的PCA类使用SVD分解来实现主成分,下面代码应用PCA将数据集的维度降到二维(它会自动处理数据集中) : 我们可以通过变量components_来访问主成分: 运行结果如下: 第一个主成分等于 pca.components_.T[:,0] 。 运行结果如下: 方差解释率可以通过变量 explained_variance_ratio_ 获得。它表示每个主成分轴对整个数据集的方差贡献度。我们看一下(3D数据集)前两个主成分的方差解释率: 运行结果如下: 结果表示,数据集方差的84%由第一条轴贡献,14%由第二条轴。 除了选择要降至的维度数量,通常更好的办法是将靠前的主成分方差解释率依次相加,直到得到足够大比例的方差,此时的维度数量就是很好的选择。 下面代码(基于MNIST数据集)计算了PCA但是没有降维,而是计算若要保留训练集方差的95%所需要的最低维度数量: 运行结果如下: 然后,我们可以设置 n_components = d ,再次运行PCA。不过还有一个更好的方法:不需要指定保留主成分的数量,我们可以直接将 n_compents 设置为 0.0 到 1.0 之间的浮点数,表示希望保留的方差比: 运行结果如下: 我们再来看一下此时的方差解释率: 运行结果如下: 上述对MNIST数据集应用主成分分析后,保留的特征只有154个(原784个)。我们也可以将缩小的数据集解压缩会784维数据集,但是它只是非常接近原始数据,并不完全相同。原始数据和重建数据(压缩后再解压缩)之间的均方距离,被称为重建误差。我们可以使用 inverse_transform() 方法实现解压缩。 下面简单实现: 运行结果如下: 结果中,左图表示原始数据,右图表示重建数据。可以看出图像质量有轻微损伤外,数字基本完好无损。说明保留其方差的95%,保留了数据的绝大部分差异性。 主成分分析的问题在于它需要整个训练集都进入内存,才能运行SVD算法。增量主成分分析:我们可以将训练集分成一个个小批量,一次给IPCA算法喂一个。 下面的代码将MNIST数据集分成100个小批量(使用NumPy的array_split()函数),将它们提供给Scikit-Learn的 IncrementalPCA ,将数据集降到154维。注意,我们还必须为每个小批量调用 partial_fit() 方法,而不是之前整个训练集的fit()方法: 运行结果如下: 核技巧是一种数学技巧,隐性地将实例映射到非常高维的空间(特征空间),从而使支持向量机能够进行非线性分类和回归。同时核技巧也可以应用PCA,使复杂的非线性投影降维成为可能。这就是所谓的核主成分分析。 下面的代码使用 Scikit-Learn 的 KernelPCA ,执行带有RBF核函数的kPCA: 运行结果如下: 上图显示了使用不同核函数降到二维的瑞士卷,包括线性函数(PCA类)、RBF核函数,以及sigmoid核函数(Logistic)。 由于kPCA是一种无监督的学习算法,因此没有明显的性能指标来帮你选择最佳的核函数和超参数值。而降维通常是监督式学习任务的准备步骤,所以可以使用网格搜索,来找到使任务性能最佳的核和超参数。 例如,下面的代码创建了一个两步流水线,首先使用kPCA将维度将至二维,然后应用逻辑回归进行分类。接下来使用 GridSearchCV 为 kPCA 找到最佳的核和gamma 值,从而在流水线最后获得最准确的分类: 运行结果如下: 还有一种完全不受监督方法,就是选择重建误差最低的核和超参数。但是这个重建不像线性PCA重建那样容易。原因如下,如下图,左上是瑞士卷的原始3D数据集,和应用RBF核的kPCA得到的二维数据集(右上)。因为核技巧,所以这在数学上等同于:通过特征映射函数 如何重建?方法之一是训练一个监督式回归模型,以投影后的实例作为训练集,并以原始实例作为目标。如果我们设置 fit_inverse_transform=True ,Scikit-Learn会自动执行该操作,如下: 然后我们就可以计算重建原像误差: 运行结果如下: 然后,我们可以使用交叉验证的网格搜索寻找使这个原像重建误差最小的核和超参数。 局部线性嵌入(LLE)是另一种非常强大的非线性降维技术。不像之前的算法依赖于投影,它是一种流形学习技术。简单来说,LLE首先测量每个算法如何与其最近的邻居线性相关,然后为训练集寻找一个能最大程度保留这些局部关系的低维表示。这也使得它特别擅长展开弯曲的流形,特别是没有太多噪声时。 下面的代码使用 Scikit-Learn 的 LocallyLinearEmbedding 类来展开瑞士卷。得到的二维数据集如下图所示。瑞士卷展开后,实例之间的距离局部保存得很好,不过从整体来看,距离保存得不太理想:展开的瑞士卷左侧被挤压,右侧被拉长。但是,对于流形建模来说,LLE还是做得相当不错。 运行结果如下: LLE工作原理: 首先,对于每个训练实例 ,算法会识别出离它最近的 k个邻居(上面的代码中k=10),然后尝试将 重建为这些邻居的线性函数。具体来说,就是要找到权重 这一步完成后,权重矩阵 下面代码是上述算法的简单实现: 运行结果如下: 学习笔记——《机器学习实战:基于Scikit-Learn和TensorFlow》# Common imports
import numpy as np
import os
# to make this notebook's output stable across runs
np.random.seed(42)
# To plot pretty figures
%matplotlib inline
import matplotlib as mpl
import matplotlib.pyplot as plt
mpl.rc('axes', labelsize=14)
mpl.rc('xtick', labelsize=12)
mpl.rc('ytick', labelsize=12)1 主成分
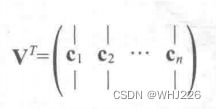
![]() ,其中
,其中 ![]() 正包含我们想要的所有主成分,公式如下:
正包含我们想要的所有主成分,公式如下:#3D数据集
np.random.seed(4)
m = 60
w1, w2 = 0.1, 0.3
noise = 0.1
angles = np.random.rand(m) * 3 * np.pi / 2 - 0.5
X = np.empty((m, 3))
X[:, 0] = np.cos(angles) + np.sin(angles)/2 + noise * np.random.randn(m) / 2
X[:, 1] = np.sin(angles) * 0.7 + noise * np.random.randn(m) / 2
X[:, 2] = X[:, 0] * w1 + X[:, 1] * w2 + noise * np.random.randn(m)
#获取主成分
X_centered = X - X.mean(axis=0)
U, s, Vt = np.linalg.svd(X_centered)
c1 = Vt.T[:, 0]
c2 = Vt.T[:, 1]2 低维度投影
![]() 的点积即可,
的点积即可,![]() 是包含前d个主成分的矩阵(即由矩阵
是包含前d个主成分的矩阵(即由矩阵 ![]() 的前d列组成的矩阵),如下式:
的前d列组成的矩阵),如下式:![]()
W2 = Vt.T[:, :2]
X2D = X_centered.dot(W2)from sklearn.decomposition import PCA
pca = PCA(n_components = 2)
X2D = pca.fit_transform(X)pca.components_array([[-0.93636116, -0.29854881, -0.18465208],
[ 0.34027485, -0.90119108, -0.2684542 ]])pca.components_.T[:,0]array([-0.93636116, -0.29854881, -0.18465208])3 方差解释率
pca.explained_variance_ratio_array([0.84248607, 0.14631839])4 选择正确数量的维度
from sklearn.datasets import fetch_openml
from sklearn.model_selection import train_test_split
mnist = fetch_openml('mnist_784', version=1, cache=True, as_frame=False)
X = mnist["data"]
y = mnist["target"]
X_train, X_test, y_train, y_test = train_test_split(X, y)pca = PCA()
pca.fit(X)
cumsum = np.cumsum(pca.explained_variance_ratio_)
d = np.argmax(cumsum >= 0.95) + 1
d154pca = PCA(n_components = 0.95)
X_reduced = pca.fit_transform(X)
pca.n_components_154np.sum(pca.explained_variance_ratio_)0.95034997020786035 PCA压缩
pca = PCA(n_components = 154)
X_reduced = pca.fit_transform(X_train)
X_recovered = pca.inverse_transform(X_reduced)
def plot_digits(instances, images_per_row=5, **options):
size = 28
images_per_row = min(len(instances), images_per_row)
images = [instance.reshape(size,size) for instance in instances]
n_rows = (len(instances) - 1) // images_per_row + 1
row_images = []
n_empty = n_rows * images_per_row - len(instances)
images.append(np.zeros((size, size * n_empty)))
for row in range(n_rows):
rimages = images[row * images_per_row : (row + 1) * images_per_row]
row_images.append(np.concatenate(rimages, axis=1))
image = np.concatenate(row_images, axis=0)
plt.imshow(image, cmap = mpl.cm.binary, **options)
plt.axis("off")
plt.figure(figsize=(7, 4))
plt.subplot(121)
plot_digits(X_train[::2100])
plt.title("Original", fontsize=16)
plt.subplot(122)
plot_digits(X_recovered[::2100])
plt.title("Compressed", fontsize=16)6 增量PCA
from sklearn.decomposition import IncrementalPCA
n_batches = 100
inc_pca = IncrementalPCA(n_components=154)
for X_batch in np.array_split(X_train, n_batches):
print(".", end="") # not shown in the book
inc_pca.partial_fit(X_batch)
X_reduced = inc_pca.transform(X_train)
X_recovered_inc_pca = inc_pca.inverse_transform(X_reduced)
plt.figure(figsize=(7, 4))
plt.subplot(121)
plot_digits(X_train[::2100])
plt.subplot(122)
plot_digits(X_recovered_inc_pca[::2100])
plt.tight_layout()7 核主成分分析
from sklearn.datasets import make_swiss_roll
X, t = make_swiss_roll(n_samples=1000, noise=0.2, random_state=42)
from sklearn.decomposition import KernelPCA
rbf_pca = KernelPCA(n_components = 2, kernel="rbf", gamma=0.04)
X_reduced = rbf_pca.fit_transform(X)
from sklearn.decomposition import KernelPCA
lin_pca = KernelPCA(n_components = 2, kernel="linear", fit_inverse_transform=True)
rbf_pca = KernelPCA(n_components = 2, kernel="rbf", gamma=0.0433, fit_inverse_transform=True)
sig_pca = KernelPCA(n_components = 2, kernel="sigmoid", gamma=0.001, coef0=1, fit_inverse_transform=True)
y = t > 6.9
plt.figure(figsize=(11, 4))
for subplot, pca, title in ((131, lin_pca, "Linear kernel"), (132, rbf_pca, "RBF kernel, $\gamma=0.04$"), (133, sig_pca, "Sigmoid kernel, $\gamma=10^{-3}, r=1$")):
X_reduced = pca.fit_transform(X)
if subplot == 132:
X_reduced_rbf = X_reduced
plt.subplot(subplot)
#plt.plot(X_reduced[y, 0], X_reduced[y, 1], "gs")
#plt.plot(X_reduced[~y, 0], X_reduced[~y, 1], "y^")
plt.title(title, fontsize=14)
plt.scatter(X_reduced[:, 0], X_reduced[:, 1], c=t, cmap=plt.cm.hot)
plt.xlabel("$z_1$", fontsize=18)
if subplot == 131:
plt.ylabel("$z_2$", fontsize=18, rotation=0)
plt.grid(True)
plt.show()![]() 使用不同核函数的kPCA将瑞士卷降到2D
使用不同核函数的kPCA将瑞士卷降到2D
8 选择核函数和调整超参数
from sklearn.model_selection import GridSearchCV
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import Pipeline
clf = Pipeline([
("kpca", KernelPCA(n_components=2)),
("log_reg", LogisticRegression(solver="liblinear"))
])
param_grid = [{
"kpca__gamma": np.linspace(0.03, 0.05, 10),
"kpca__kernel": ["rbf", "sigmoid"]
}]
grid_search = GridSearchCV(clf, param_grid, cv=3)
grid_search.fit(X, y)
#最佳的核和超参数
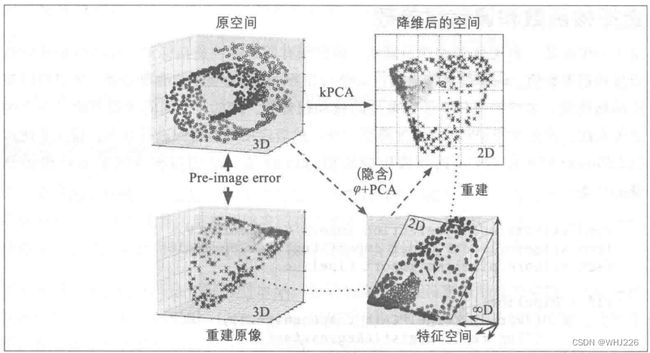
print(grid_search.best_params_){'kpca__gamma': 0.043333333333333335, 'kpca__kernel': 'rbf'}![]() ,将训练集映射到无限维度的特征空间(右下),然后用线性PCA将转换后的训练集投影到2D平面。注意,如果我们对一个已经降维的实例进行PCA逆转换,重建的点将存在于特征空间,而不是原始空间中(例如,图中x表示的那个点)。而这里特征空间是无限维度的,所以我们无法计算出重建点,因此也无法计算出真实的重建误差。不过,我们可以在原始空间中找到一个点,使其映射接近于重建点,这被称为重建原像。一旦有了这个原像,我们就可以测量它到原始实例的平方距离。最后,选择使这个重建原像误差最小化的核和超参数。
,将训练集映射到无限维度的特征空间(右下),然后用线性PCA将转换后的训练集投影到2D平面。注意,如果我们对一个已经降维的实例进行PCA逆转换,重建的点将存在于特征空间,而不是原始空间中(例如,图中x表示的那个点)。而这里特征空间是无限维度的,所以我们无法计算出重建点,因此也无法计算出真实的重建误差。不过,我们可以在原始空间中找到一个点,使其映射接近于重建点,这被称为重建原像。一旦有了这个原像,我们就可以测量它到原始实例的平方距离。最后,选择使这个重建原像误差最小化的核和超参数。rbf_pca = KernelPCA(n_components = 2, kernel="rbf", gamma=0.0433,
fit_inverse_transform=True)
X_reduced = rbf_pca.fit_transform(X)
X_preimage = rbf_pca.inverse_transform(X_reduced)from sklearn.metrics import mean_squared_error
mean_squared_error(X, X_preimage)4.236482168742404e-279 局部线性嵌入
X, t = make_swiss_roll(n_samples=1000, noise=0.2, random_state=41)
from sklearn.manifold import LocallyLinearEmbedding
lle = LocallyLinearEmbedding(n_components=2, n_neighbors=10, random_state=42)
X_reduced = lle.fit_transform(X)
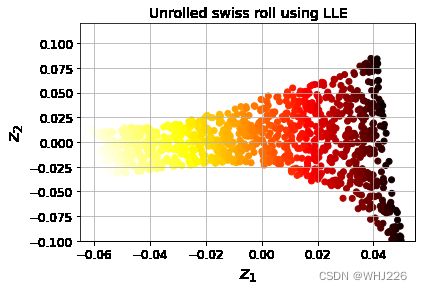
plt.title("Unrolled swiss roll using LLE", fontsize=14)
plt.scatter(X_reduced[:, 0], X_reduced[:, 1], c=t, cmap=plt.cm.hot)
plt.xlabel("$z_1$", fontsize=18)
plt.ylabel("$z_2$", fontsize=18)
plt.axis([-0.065, 0.055, -0.1, 0.12])
plt.grid(True)
plt.show()![]() 使实例和 之间的距离平方最小,如果实例 不是实例 的k个最近的邻居之一,
使实例和 之间的距离平方最小,如果实例 不是实例 的k个最近的邻居之一,![]() 。因此,LLE的第一步就是如下公式所示的约束优化问题,其中W是包含所有权重
。因此,LLE的第一步就是如下公式所示的约束优化问题,其中W是包含所有权重 ![]() 的权重矩阵,第二个约束则是简单地对每个训练实例 的权重进行归一。
的权重矩阵,第二个约束则是简单地对每个训练实例 的权重进行归一。![]() LLE第一步:对局部关系线性建模
LLE第一步:对局部关系线性建模
![]() (包含权重
(包含权重 ![]() )对训练实例之间的局部线性关系进行编码。现在,第二步就是要将训练实例映射到一个d维空间(d
)对训练实例之间的局部线性关系进行编码。现在,第二步就是要将训练实例映射到一个d维空间(d![]() 到
到 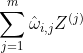 之间的平方距离尽可能小。如下无约束优化问题。它看起来与第一步雷士,但不是保持固定距离寻找最佳权重,而是保持固定权重,并在低维空间中找到每个实例映像的最佳位置。注意,Z是包含所有
之间的平方距离尽可能小。如下无约束优化问题。它看起来与第一步雷士,但不是保持固定距离寻找最佳权重,而是保持固定权重,并在低维空间中找到每个实例映像的最佳位置。注意,Z是包含所有![]() 的矩阵 。
的矩阵 。10 其他降维技巧
from sklearn.manifold import MDS
mds = MDS(n_components=2, random_state=42)
X_reduced_mds = mds.fit_transform(X)
from sklearn.manifold import Isomap
isomap = Isomap(n_components=2)
X_reduced_isomap = isomap.fit_transform(X)
from sklearn.manifold import TSNE
tsne = TSNE(n_components=2, random_state=42)
X_reduced_tsne = tsne.fit_transform(X)
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
lda = LinearDiscriminantAnalysis(n_components=2)
X_mnist = mnist["data"]
y_mnist = mnist["target"]
lda.fit(X_mnist, y_mnist)
X_reduced_lda = lda.transform(X_mnist)
titles = ["MDS", "Isomap", "t-SNE"]
plt.figure(figsize=(11,4))
for subplot, title, X_reduced in zip((131, 132, 133), titles,
(X_reduced_mds, X_reduced_isomap, X_reduced_tsne)):
plt.subplot(subplot)
plt.title(title, fontsize=14)
plt.scatter(X_reduced[:, 0], X_reduced[:, 1], c=t, cmap=plt.cm.hot)
plt.xlabel("$z_1$", fontsize=18)
if subplot == 131:
plt.ylabel("$z_2$", fontsize=18, rotation=0)
plt.grid(True)
plt.show()