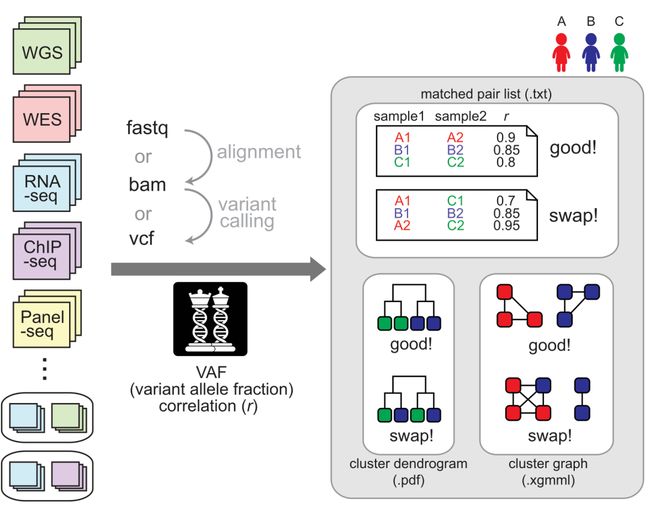
- 程序化交易系统中如何精准获取MACD、KDJ、BOLL等基础指标的值?
股票程序化交易接口
量化交易股票API接口Python股票量化交易程序化交易系统macd指标kdj指标boll指标股票量化接口股票API接口
Python股票接口实现查询账户,提交订单,自动交易(1)Python股票程序交易接口查账,提交订单,自动交易(2)股票量化,Python炒股,CSDN交流社区>>>基础指标在程序化交易系统中的重要性基础指标对交易决策的指导意义MACD、KDJ、BOLL等基础指标在程序化交易系统中扮演着重要角色。MACD可以帮助判断市场的趋势和买卖信号,通过分析其快线和慢线的交叉情况,能为投资者提供入场和出场的参
- Python爬虫实战:全方位爬取知乎学习板块问答数据
Python爬虫项目
2025年爬虫实战项目python爬虫学习开发语言scrapy游戏
1.项目背景与爬取目标知乎是中国最大的知识问答社区,聚集了大量高质量的学习资源和经验分享。爬取知乎“学习”板块的问答数据,可以为学习资料整理、舆情分析、推荐系统开发等提供数据支持。本项目目标:爬取“学习”话题下的热门问答列表抓取每个问答的标题、作者、回答内容、点赞数、评论数等详细信息实现动态加载内容的抓取,包含图片和富文本避免被反爬机制限制,保证数据采集稳定结合数据分析,为后续应用打基础2.知乎“
- Python实战:自动在知乎回答点赞并采集内容的高阶爬虫教程
Python爬虫项目
2025年爬虫实战项目python爬虫开发语言okhttp学习
✨写在前面:为什么做知乎自动化操作?知乎作为中国领先的知识问答平台,拥有大量结构化内容。对于研究舆情分析、情绪识别、用户画像,甚至产品舆情反馈采集的用户来说,如何自动获取知乎内容并进行交互行为(如点赞、回答),是一个非常实用的能力。本文将手把手带你用Python完成以下目标:✅自动登录知乎✅自动搜索某个关键词下的热门问题✅自动点赞高质量回答✅自动采集回答内容(文本、点赞数、评论数等)✅自动保存为本
- Python爬虫实战:爬取知乎问答与用户信息
Python爬虫项目
python爬虫php数据分析开发语言开源
简介随着网络信息量的爆炸,如何有效获取有价值的内容,成为了数据分析、机器学习等领域的基础之一。爬虫作为数据采集的基本工具之一,常常被用来获取互联网上的公开数据。在这篇博客中,我们将结合最新的Python爬虫技术,详细讲解如何爬取知乎问答与用户信息。本文将会介绍:Python爬虫的基础知识知乎问答网页结构分析使用Python进行知乎数据爬取爬取知乎问答内容与用户信息如何处理和存储爬取的数据使用最新的
- 【机器学习&深度学习】反向传播机制
目录一、一句话定义二、类比理解三、为什重要?四、用生活例子解释:神经网络=烹饪机器人4.1第一步:尝一口(前向传播)4.2第二步:倒着推原因(反向传播)五、换成人工智能流程说一遍六、图示类比:找山顶(最优参数)七、总结一句人话八、PyTorch代码示例:亲眼看到每一层的梯度九、梯度=损失函数对参数的偏导数十、类比总结反向传播(Backpropagation)是神经网络中训练过程的核心机制,它就像“
- 人脸识别算法赋能园区无人超市安防升级
智驱力人工智能
算法人工智能边缘计算人脸识别智慧园区智慧工地智慧煤矿
人脸识别算法赋能园区无人超市安防升级正文在园区无人超市的运营管理中,传统安防手段依赖人工巡检或基础监控设备,存在响应滞后、误报率高、环境适应性差等问题。本文从技术背景、实现路径、功能优势及应用场景四个维度,阐述如何通过人脸识别检测、人员入侵算法及疲劳检测算法的协同应用,构建高效、精准的智能安防体系。一、技术背景:视觉分析算法的核心支撑人脸识别算法基于深度学习的卷积神经网络(CNN)模型,通过提取面
- .NET C# async/定时任务的异步线程池调度方案最大线程数 = 处理器核心数 × 250
专注VB编程开发20年
.netc#开发语言
关于.NET中Threading.Timer的线程机制,结合线程池特性和异步协作原理分析如下:一、线程复用机制共享进程级线程池Threading.Timer的回调任务不会每次新建线程,而是提交到.NET进程全局线程池统一调度,该线程池与async/await任务共享同一资源池。线程池维护可复用工作线程队列,避免频繁创建/销毁开销任务优先由空闲线程执行,无空闲线程则进入全局队列等待线程池扩
- 企业级知识库私有化部署:腾讯混元+云容器服务TKE实战
大熊计算机
#腾讯云语言模型
1.背景需求分析在金融、医疗等数据敏感行业,企业需要构建完全自主可控的知识库系统。本文以某证券机构智能投研系统为原型,演示如何基于腾讯混元大模型与TKE容器服务实现:千亿级参数模型的私有化部署金融领域垂直场景微调高并发低延迟推理服务全链路安全合规方案1.1典型技术挑战#性能基准测试数据(单位:QPS)|场景|裸机部署|容器化部署|优化后||--------------------|--------
- SnowConvert:自动化数据迁移的技术解析与最佳实践
weixin_30777913
迁移学习数据库运维
SnowConvert是Snowflake生态系统的关键迁移工具,专为将传统数据仓库(如Oracle、Teradata、SQLServer等)的代码资产高效、准确地转换为Snowflake原生语法而设计。以下基于官方文档对其技术原理、工作流程及最佳实践进行深入分析:一、SnowConvert核心技术解析精准的语法映射引擎语言支持:深度解析源系统特有语法(OraclePL/SQL,TeradataB
- Linux ps 指令
halugin
Linux指令linux运维
Linuxps指令ps(ProcessStatus)是Linux系统中用于查看进程状态的核心命令行工具。它提供系统当前运行进程的快照,显示进程ID、CPU和内存使用情况、运行状态等信息。作为系统管理员或开发人员,ps是监控系统资源、排查性能问题和管理系统进程的必备工具。其灵活的选项和输出格式使其适用于从简单查询到复杂分析的各种场景。什么是ps指令?概述ps是一个经典的Linux/Unix命令,用于
- Linux netstat 指令
halugin
Linux指令linux运维
Linuxnetstat指令netstat(NetworkStatistics)是Linux系统中用于查看网络状态、连接、路由表和接口统计信息的经典命令行工具。它为系统管理员和开发人员提供了强大的网络诊断功能,帮助分析网络连接、监控流量以及排查网络问题。尽管在现代Linux系统中,netstat正在被更新的工具(如ss)部分取代,但其简单性和广泛适用性使其仍然是许多场景下的首选工具。什么是nets
- Linux ss 指令
halugin
Linux指令linux运维
Linuxss指令ss(SocketStatistics)是Linux系统中用于显示网络套接字(socket)信息的现代命令行工具,是netstat的继任者,性能更高、输出更简洁。它提供详细的网络连接、监听端口和协议统计信息,广泛用于网络监控、故障排查和性能分析。相比传统的netstat,ss直接从内核获取数据显示更快,功能更强大,适合现代Linux系统。什么是ss指令?ss是Linux系统中的一
- VB.NET,C#字典对象来保存用户数据,支持大小写
专注VB编程开发20年
java开发语言
用这个保存的,登录时大小写不一样会不会无法识别根据你提供的SaveUsersToJson方法,我注意到你使用了JSON序列化来保存用户数据,但没有显式指定字典的比较器。这意味着在反序列化时,默认会使用区分大小写的比较器,导致大小写不同的用户名无法正确匹配。问题分析当你保存用户数据时:PrivateSubSaveUsersToJson(usersAsDictionary(OfString,UserI
- 疲劳检测与行为分析:工厂智能化实践
智驱力人工智能
安全智慧城市行为识别人员属性识别疲劳检测抽烟检测徘徊检测
视觉分析算法赋能工厂疲劳与安全管理一、背景与需求在制造业中,疲劳作业是导致安全事故和效率下降的核心因素之一。传统人工巡检存在覆盖面不足、响应滞后等问题,而基于视觉分析的智能监控系统通过多算法协同,可实现全天候、高精度的疲劳检测与行为管理。本文围绕疲劳检测算法、人员计数算法、抽烟检测算法及徘徊检测算法,探讨其在工厂场景中的技术实现与应用价值。二、技术实现疲劳检测算法原理:基于PERCLOS(眼睑闭合
- GPT-4o重磅升级!只需一条指令,教你秒出SCI级专业科研图!
智写AI
AI学术写作指南信息可视化人工智能
经过数月爆肝,七哥终于完成专业的学术AI使用教程,估计也有个80万字的详细操作指南。分为多个细分的专业写作场景,跟着一步一步操作,借助ChatGPT做学术、干科研、写论文、课题申报都变得超简单。欢迎加我交流(yida985),祝你一臂之力。七哥之前写过关于用AI生成流程图的教程,不过需要借助其他软件才能搞定完美的流程图。近期GPT-4o全新推出了“生图功能”,这个生图的过程就更加方便轻松了,全能G
- Python 数据挖掘实战: 关联规则与聚类分析,解锁数据价值的钥匙
清水白石008
pythonPython题库python数据挖掘动画
Python数据挖掘实战:关联规则与聚类分析,解锁数据价值的钥匙引言在数字化浪潮席卷全球的今天,数据已成为企业和组织最重要的战略资产。海量数据蕴藏着巨大的价值,等待我们去挖掘和发现。数据挖掘(DataMining),作为从海量数据中提取有价值知识和模式的关键技术,正日益受到各行各业的重视。它如同探矿者的火眼金睛,能够穿透数据的迷雾,发现隐藏在背后的规律和趋势,为商业决策、科学研究和社会发展提供强有
- 潜入思维的海洋:SoftCoT++如何让语言模型更聪明
步子哥
智能涌现语言模型人工智能自然语言处理
在人工智能的浩瀚星空下,大型语言模型(LLMs)如同一颗颗璀璨的恒星,照亮了从文本生成到复杂推理的广阔领域。然而,这些模型在推理任务中往往像是在迷雾中航行——尽管它们能抵达目的地,却常常因为固定的思维路径而错过更优的航线。2025年5月,一篇题为《SoftCoT++:Test-TimeScalingwithSoftChain-of-ThoughtReasoning》的论文如同一盏明灯,照亮了如何让
- 数学分析(十八)-隐函数定理及其应用1-隐函数4:隐函数极值问题
u013250861
数学分析数学分析
f′(x)=−Fx(x,y)Fy(x,y)(5)f^{\prime}(x)=-\cfrac{F_{x}(x,y)}{F_{y}(x,y)}\quad\quad(5)f′(x)=−Fy(x,y)Fx(x,y)(5)y′′=−1Fy(Fxx+2Fxyy′+Fyyy′2)=2FxFyFxy−Fy2Fxx−Fx2FyyFy3,(
- 为什么90%企业的AI数据分析都失败了?奥威BI给出破局方案
qq_43696218
人工智能数据分析数据挖掘
一、引言:AI数据分析在数字化转型中的核心地位在当今企业全面数字化转型的背景下,AI数据分析已成为解锁业务增长潜力的关键钥匙。然而,市场上众多AI数据分析产品常陷入“伪需求场景”,看似前沿却难以真正落地。本文将深入探讨奥威BI如何通过其AI数据分析能力,突破伪需求,实现数据价值的最大化。二、AI数据分析:伪需求场景的挑战伪需求场景的定义与表现AI数据分析领域的伪需求场景,指的是那些表面创新实则难
- BI+AI实战:我们如何用3秒完成车企供应链推演
qq_43696218
人工智能
一、BI+AI引领财务分析新纪元在财务数据分析领域,奥威BI+AI正以革命性的姿态颠覆传统。当金蝶、用友等工具仍深陷报表泥潭时,奥威BI+AI通过深度融合商业智能(BI)与人工智能(AI),实现了从滞后报表到实时洞察的飞跃。这不仅极大地提升了财务分析的效率,更为企业的战略决策提供了前所未有的精准支持。二、BI+AI的核心技术优势实时动态分析o奥威BI+AI摒弃了静态数据集,依托原始科目余额表实
- CBAP50技术手册】#47 Use Cases & Scenarios(用例与场景):BA(业务分析师)让需求“活起来”的剧本写作术
郭菁菁
BA业务分析需求分析
把需求演绎成系统与用户的真实互动剧本。在一次项目需求评审会上,开发组沉默不语,业务方焦躁不安。写在文档里的需求,似乎谁都“看懂了”,但又好像“谁都没真正理解”。直到我用一组UseCases&Scenarios把冷冰冰的需求变成了一场场“用户剧本”,大家才终于“看见”了系统该如何运作,沟通顿时顺畅了。UseCases和Scenarios,就像是BA的“剧作笔”——把抽象需求,演绎成生动细节。什么是U
- 技术调研:时序数据库(一)
myskybeyond
时序数据库时序数据库数据库
选择时序数据库时,选择当下主流的解决方案。目前主流的开源解决方案有InfluxDB、TDengine和TimescaleDB。下文从多个维度对比分析,最终根据需求做出选型决策。1.核心架构与设计理念数据库架构特点核心优势InfluxDB-专为时序数据设计的分布式数据库-基于时间线(TimeSeries)模型-开源版(OSS)与商业版(Cloud/Enterprise)功能差异大高写入吞吐量、原生支
- 初中学习机推荐:从功能、内容到用户体验的深度解析
资讯分享周
ux人工智能
在教育信息化持续深化的背景下,初中阶段的学习辅助设备正逐步成为家长和学生关注的重点。尤其在“双减”政策推动下,传统补习班的作用被削弱,越来越多家庭开始依赖智能学习工具来提升学习效率和自主性。其中,初中学习机因其集视频课程、AI辅导、错题整理、学习反馈等多功能于一体,成为当前市场热度最高的教育硬件之一。本文将围绕市场上主流的几款初中学习机进行客观分析,重点介绍简单一百、学而思、科大讯飞、作业帮四款产
- DeepSeek-V3 通俗详解:从诞生到优势,以及与 GPT-4o 的对比
码事漫谈
AIai
前些天发现了一个巨牛的人工智能学习网站,通俗易懂,风趣幽默,忍不住分享一下给大家。点击跳转到网站1.DeepSeek的前世今生1.1什么是DeepSeek?DeepSeek是一家专注于人工智能技术研发的公司,致力于打造高性能、低成本的AI模型。它的目标是让AI技术更加普惠,让更多人能够用上强大的AI工具。1.2DeepSeek-V3的诞生DeepSeek-V3是DeepSeek公司推出的最新一代A
- 企业级AI开发利器:Spring AI框架深度解析与实战_spring ai实战
AI大模型-海文
人工智能springpython算法开发语言java机器学习
企业级AI开发利器:SpringAI框架深度解析与实战一、前言:Java生态的AI新纪元在人工智能技术爆发式发展的今天,Java开发者面临着一个新的挑战:如何将大语言模型(LLMs)和生成式AI(GenAI)无缝融入企业级应用。传统的Java生态缺乏统一的AI集成方案,开发者往往需要为不同AI供应商(如OpenAI、阿里云、HuggingFace)编写大量重复的接口适配代码,这不仅增加了开发成本,
- Linux操作系统,故障排查
月堂
linux运维服务器
案例1:GRUB引导故障故障现象:系统启动卡在"GRUB>"提示符,无法进入系统原因分析:GRUB配置文件损坏(/boot/grub/grub.cfg)引导文件被误删或磁盘损坏解决步骤:在GRUB命令行依次执行:insmodxfssetroot=(hd0,msdos1)linux/vmlinuz-root=/dev/sda1initrd/initramfs-.imgboot进入系统后执行:grub
- Python编程:使用Opencv进行图像处理
【参考】https://github.com/opencv/opencv/tree/4.x/samples/pythonPython使用OpenCV进行图像处理OpenCV(OpenSourceComputerVisionLibrary)是一个开源的计算机视觉和机器学习软件库。下面将从基础到高阶介绍如何使用Python中的OpenCV进行图像处理。一、安装首先需要安装OpenCV库:pipinst
- 智力题——5L的桶和3L的桶如何装4L的水
酒醉梦醒
算法数据结构java5升水和3升水图论bfs状态压缩
文章目录智力题——5L的桶和3L的桶如何装4L的水问题描述直观分析问题建模问题解决智力题——5L的桶和3L的桶如何装4L的水问题描述有一个5L的桶A和一个3L的桶B以及无限量的水,如何让5L的桶装4L的水。支持操作:加水,倒水,A倒入B,B倒入A,除此之外不再支持其他操作,例如做记号或者借助其他工具直观分析直观分析就是利用我们的直观思维在草纸上不停的模拟这些操作,这个很不好说,对于简单问题你可能可
- 【Linux】ghb工具
nanguochenchuan
Linux操作系统linux运维服务器
GDB简介GDB(GNUDebugger)是Linux系统中最强大的命令行调试工具,由GNU项目开发。作为程序员调试C/C++程序的利器,GDB能帮助你:定位程序崩溃原因分析程序运行状态跟踪变量值变化检测内存错误安装与配置安装方法#Ubuntu/Debiansudoaptinstallgdb#CentOS/RHELsudoyuminstallgdb#ArchLinuxsudopacman-Sgdb
- Vue SPA 路由跳转无法回到顶部问题排查与解决
浪裡遊
vue.jsjavascriptecmascriptpiniarouterhtml
VueSPA路由跳转无法回到顶部问题排查与解决1.问题现象描述在使用Vue3+VueRouter4开发单页应用(SPA)时,遇到如下问题:点击导航栏或页脚的路由跳转后,页面没有自动回到顶部。即使配置了VueRouter的scrollBehavior,页面依然没有回到顶部的效果。有时内容会被导航栏遮住,看起来像"没有回到顶部"。2.常见原因分析内容区没有为导航栏预留空间导航栏是fixed或stick
- Java常用排序算法/程序员必须掌握的8大排序算法
cugfy
java
分类:
1)插入排序(直接插入排序、希尔排序)
2)交换排序(冒泡排序、快速排序)
3)选择排序(直接选择排序、堆排序)
4)归并排序
5)分配排序(基数排序)
所需辅助空间最多:归并排序
所需辅助空间最少:堆排序
平均速度最快:快速排序
不稳定:快速排序,希尔排序,堆排序。
先来看看8种排序之间的关系:
1.直接插入排序
(1
- 【Spark102】Spark存储模块BlockManager剖析
bit1129
manager
Spark围绕着BlockManager构建了存储模块,包括RDD,Shuffle,Broadcast的存储都使用了BlockManager。而BlockManager在实现上是一个针对每个应用的Master/Executor结构,即Driver上BlockManager充当了Master角色,而各个Slave上(具体到应用范围,就是Executor)的BlockManager充当了Slave角色
- linux 查看端口被占用情况详解
daizj
linux端口占用netstatlsof
经常在启动一个程序会碰到端口被占用,这里讲一下怎么查看端口是否被占用,及哪个程序占用,怎么Kill掉已占用端口的程序
1、lsof -i:port
port为端口号
[root@slave /data/spark-1.4.0-bin-cdh4]# lsof -i:8080
COMMAND PID USER FD TY
- Hosts文件使用
周凡杨
hostslocahost
一切都要从localhost说起,经常在tomcat容器起动后,访问页面时输入http://localhost:8088/index.jsp,大家都知道localhost代表本机地址,如果本机IP是10.10.134.21,那就相当于http://10.10.134.21:8088/index.jsp,有时候也会看到http: 127.0.0.1:
- java excel工具
g21121
Java excel
直接上代码,一看就懂,利用的是jxl:
import java.io.File;
import java.io.IOException;
import jxl.Cell;
import jxl.Sheet;
import jxl.Workbook;
import jxl.read.biff.BiffException;
import jxl.write.Label;
import
- web报表工具finereport常用函数的用法总结(数组函数)
老A不折腾
finereportweb报表函数总结
ADD2ARRAY
ADDARRAY(array,insertArray, start):在数组第start个位置插入insertArray中的所有元素,再返回该数组。
示例:
ADDARRAY([3,4, 1, 5, 7], [23, 43, 22], 3)返回[3, 4, 23, 43, 22, 1, 5, 7].
ADDARRAY([3,4, 1, 5, 7], "测试&q
- 游戏服务器网络带宽负载计算
墙头上一根草
服务器
家庭所安装的4M,8M宽带。其中M是指,Mbits/S
其中要提前说明的是:
8bits = 1Byte
即8位等于1字节。我们硬盘大小50G。意思是50*1024M字节,约为 50000多字节。但是网宽是以“位”为单位的,所以,8Mbits就是1M字节。是容积体积的单位。
8Mbits/s后面的S是秒。8Mbits/s意思是 每秒8M位,即每秒1M字节。
我是在计算我们网络流量时想到的
- 我的spring学习笔记2-IoC(反向控制 依赖注入)
aijuans
Spring 3 系列
IoC(反向控制 依赖注入)这是Spring提出来了,这也是Spring一大特色。这里我不用多说,我们看Spring教程就可以了解。当然我们不用Spring也可以用IoC,下面我将介绍不用Spring的IoC。
IoC不是框架,她是java的技术,如今大多数轻量级的容器都会用到IoC技术。这里我就用一个例子来说明:
如:程序中有 Mysql.calss 、Oracle.class 、SqlSe
- 高性能mysql 之 选择存储引擎(一)
annan211
mysqlInnoDBMySQL引擎存储引擎
1 没有特殊情况,应尽可能使用InnoDB存储引擎。 原因:InnoDB 和 MYIsAM 是mysql 最常用、使用最普遍的存储引擎。其中InnoDB是最重要、最广泛的存储引擎。她 被设计用来处理大量的短期事务。短期事务大部分情况下是正常提交的,很少有回滚的情况。InnoDB的性能和自动崩溃 恢复特性使得她在非事务型存储的需求中也非常流行,除非有非常
- UDP网络编程
百合不是茶
UDP编程局域网组播
UDP是基于无连接的,不可靠的传输 与TCP/IP相反
UDP实现私聊,发送方式客户端,接受方式服务器
package netUDP_sc;
import java.net.DatagramPacket;
import java.net.DatagramSocket;
import java.net.Ine
- JQuery对象的val()方法执行结果分析
bijian1013
JavaScriptjsjquery
JavaScript中,如果id对应的标签不存在(同理JAVA中,如果对象不存在),则调用它的方法会报错或抛异常。在实际开发中,发现JQuery在id对应的标签不存在时,调其val()方法不会报错,结果是undefined。
- http请求测试实例(采用json-lib解析)
bijian1013
jsonhttp
由于fastjson只支持JDK1.5版本,因些对于JDK1.4的项目,可以采用json-lib来解析JSON数据。如下是http请求的另外一种写法,仅供参考。
package com;
import java.util.HashMap;
import java.util.Map;
import
- 【RPC框架Hessian四】Hessian与Spring集成
bit1129
hessian
在【RPC框架Hessian二】Hessian 对象序列化和反序列化一文中介绍了基于Hessian的RPC服务的实现步骤,在那里使用Hessian提供的API完成基于Hessian的RPC服务开发和客户端调用,本文使用Spring对Hessian的集成来实现Hessian的RPC调用。
定义模型、接口和服务器端代码
|---Model
&nb
- 【Mahout三】基于Mahout CBayes算法的20newsgroup流程分析
bit1129
Mahout
1.Mahout环境搭建
1.下载Mahout
http://mirror.bit.edu.cn/apache/mahout/0.10.0/mahout-distribution-0.10.0.tar.gz
2.解压Mahout
3. 配置环境变量
vim /etc/profile
export HADOOP_HOME=/home
- nginx负载tomcat遇非80时的转发问题
ronin47
nginx负载后端容器是tomcat(其它容器如WAS,JBOSS暂没发现这个问题)非80端口,遇到跳转异常问题。解决的思路是:$host:port
详细如下:
该问题是最先发现的,由于之前对nginx不是特别的熟悉所以该问题是个入门级别的:
? 1 2 3 4 5
- java-17-在一个字符串中找到第一个只出现一次的字符
bylijinnan
java
public class FirstShowOnlyOnceElement {
/**Q17.在一个字符串中找到第一个只出现一次的字符。如输入abaccdeff,则输出b
* 1.int[] count:count[i]表示i对应字符出现的次数
* 2.将26个英文字母映射:a-z <--> 0-25
* 3.假设全部字母都是小写
*/
pu
- mongoDB 复制集
开窍的石头
mongodb
mongo的复制集就像mysql的主从数据库,当你往其中的主复制集(primary)写数据的时候,副复制集(secondary)会自动同步主复制集(Primary)的数据,当主复制集挂掉以后其中的一个副复制集会自动成为主复制集。提供服务器的可用性。和防止当机问题
mo
- [宇宙与天文]宇宙时代的经济学
comsci
经济
宇宙尺度的交通工具一般都体型巨大,造价高昂。。。。。
在宇宙中进行航行,近程采用反作用力类型的发动机,需要消耗少量矿石燃料,中远程航行要采用量子或者聚变反应堆发动机,进行超空间跳跃,要消耗大量高纯度水晶体能源
以目前地球上国家的经济发展水平来讲,
- Git忽略文件
Cwind
git
有很多文件不必使用git管理。例如Eclipse或其他IDE生成的项目文件,编译生成的各种目标或临时文件等。使用git status时,会在Untracked files里面看到这些文件列表,在一次需要添加的文件比较多时(使用git add . / git add -u),会把这些所有的未跟踪文件添加进索引。
==== ==== ==== 一些牢骚
- MySQL连接数据库的必须配置
dashuaifu
mysql连接数据库配置
MySQL连接数据库的必须配置
1.driverClass:com.mysql.jdbc.Driver
2.jdbcUrl:jdbc:mysql://localhost:3306/dbname
3.user:username
4.password:password
其中1是驱动名;2是url,这里的‘dbna
- 一生要养成的60个习惯
dcj3sjt126com
习惯
一生要养成的60个习惯
第1篇 让你更受大家欢迎的习惯
1 守时,不准时赴约,让别人等,会失去很多机会。
如何做到:
①该起床时就起床,
②养成任何事情都提前15分钟的习惯。
③带本可以随时阅读的书,如果早了就拿出来读读。
④有条理,生活没条理最容易耽误时间。
⑤提前计划:将重要和不重要的事情岔开。
⑥今天就准备好明天要穿的衣服。
⑦按时睡觉,这会让按时起床更容易。
2 注重
- [介绍]Yii 是什么
dcj3sjt126com
PHPyii2
Yii 是一个高性能,基于组件的 PHP 框架,用于快速开发现代 Web 应用程序。名字 Yii (读作 易)在中文里有“极致简单与不断演变”两重含义,也可看作 Yes It Is! 的缩写。
Yii 最适合做什么?
Yii 是一个通用的 Web 编程框架,即可以用于开发各种用 PHP 构建的 Web 应用。因为基于组件的框架结构和设计精巧的缓存支持,它特别适合开发大型应
- Linux SSH常用总结
eksliang
linux sshSSHD
转载请出自出处:http://eksliang.iteye.com/blog/2186931 一、连接到远程主机
格式:
ssh name@remoteserver
例如:
ssh
[email protected]
二、连接到远程主机指定的端口
格式:
ssh name@remoteserver -p 22
例如:
ssh i
- 快速上传头像到服务端工具类FaceUtil
gundumw100
android
快速迭代用
import java.io.DataOutputStream;
import java.io.File;
import java.io.FileInputStream;
import java.io.FileNotFoundException;
import java.io.FileOutputStream;
import java.io.IOExceptio
- jQuery入门之怎么使用
ini
JavaScripthtmljqueryWebcss
jQuery的强大我何问起(个人主页:hovertree.com)就不用多说了,那么怎么使用jQuery呢?
首先,下载jquery。下载地址:http://hovertree.com/hvtart/bjae/b8627323101a4994.htm,一个是压缩版本,一个是未压缩版本,如果在开发测试阶段,可以使用未压缩版本,实际应用一般使用压缩版本(min)。然后就在页面上引用。
- 带filter的hbase查询优化
kane_xie
查询优化hbaseRandomRowFilter
问题描述
hbase scan数据缓慢,server端出现LeaseException。hbase写入缓慢。
问题原因
直接原因是: hbase client端每次和regionserver交互的时候,都会在服务器端生成一个Lease,Lease的有效期由参数hbase.regionserver.lease.period确定。如果hbase scan需
- java设计模式-单例模式
men4661273
java单例枚举反射IOC
单例模式1,饿汉模式
//饿汉式单例类.在类初始化时,已经自行实例化
public class Singleton1 {
//私有的默认构造函数
private Singleton1() {}
//已经自行实例化
private static final Singleton1 singl
- mongodb 查询某一天所有信息的3种方法,根据日期查询
qiaolevip
每天进步一点点学习永无止境mongodb纵观千象
// mongodb的查询真让人难以琢磨,就查询单天信息,都需要花费一番功夫才行。
// 第一种方式:
coll.aggregate([
{$project:{sendDate: {$substr: ['$sendTime', 0, 10]}, sendTime: 1, content:1}},
{$match:{sendDate: '2015-
- 二维数组转换成JSON
tangqi609567707
java二维数组json
原文出处:http://blog.csdn.net/springsen/article/details/7833596
public class Demo {
public static void main(String[] args) { String[][] blogL
- erlang supervisor
wudixiaotie
erlang
定义supervisor时,如果是监控celuesimple_one_for_one则删除children的时候就用supervisor:terminate_child (SupModuleName, ChildPid),如果shutdown策略选择的是brutal_kill,那么supervisor会调用exit(ChildPid, kill),这样的话如果Child的behavior是gen_