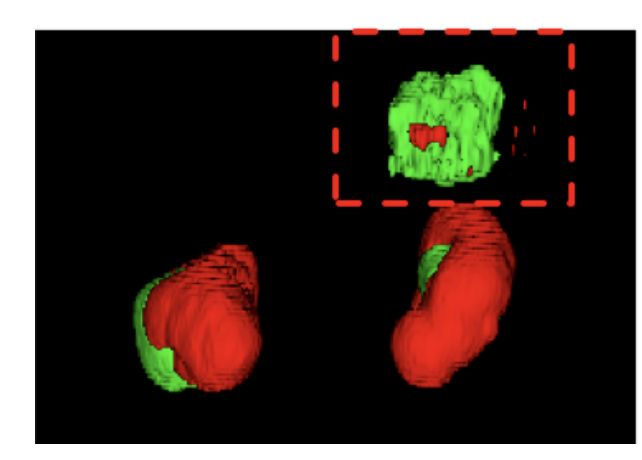
深度学习,分割后处理之通过连通成分分析去除假阳性区域,提高分割准确度
用深度学习方法得到的分割结果,会有一些假阳性区域。通过去除这些假阳性区域,可以提高分割结果。
比如说做肾分割,大家都知道,肾只有左右两边有,如果分割结果出现了三个区域,则可以根据常识,去除那个假阳性区域。
用到的方法就是 连通成分分析Connected-Components。
这里提供三种方法:
1. opencv-python 提供的方法
安装: pip install opencv-python cv2.connectedComponents & cv2.connectedComponentsWithStats
1.1 cv2.connectedComponents
实例:
import cv2
import numpy as np
img = np.array([[0, 255, 0, 0],
[0, 0, 255, 255],
[0, 0, 0, 255],
[255, 0, 0, 0]], np.uint8)
res, labels = cv2.connectedComponents(img, connectivity=4)
res
Out[5]: 4
labels
Out[6]:
array([[0, 1, 0, 0],
[0, 0, 2, 2],
[0, 0, 0, 2],
[3, 0, 0, 0]], dtype=int32)
假设 img 是分割结果,值为255的是目标区域,可以发现,目标区域有3块。res代表区域的数量 一共有4块。
labels为经过连通区域分析后的标记,把 4邻域 内相同的值标记为一个类别。这样一共产生了4个区域,分别用【0,1,2,3】表示。
上述例子例子中,参数 connectivity=4 表示在4邻域范围内查找元素,也是可以改成8邻域对比一下
4邻域:A点的上下左右中,假设存在B点和它的值一样,就表示 AB 点属于同一区域。
这样标记后,如果我们觉得3那个区域是假阳性,那我们就可以把3那个区域的值变为0,其余区域的值标记为255,这样就消除掉3这个区域的阳性值了。
label = np.where(labels > 2, 0, labels)
# 把labels中,大于2的值,赋值为0, 其余的就是labels原来的值。这样就剩下了两个区域
print(label)
结果如下:
[[0 1 0 0]
[0 0 2 2]
[0 0 0 2]
[0 0 0 0]]
1.2 cv2.connectedComponentsWithStats
stats 是 bounding box 的信息,N*5的矩阵,行对应每个label,五列分别为 [x0, y0, width, height, area]
参考链接[1]
总结:该方法对二维图像去除假阳性区域很好用,但是无法对三维图像进行操作。
2 cc3d 提供的方法
cc3d 提供了二维和三维的方法实现连通成分分析
-
3D 方法: 提供 26、18 或 6 个连通邻域划分区域
-
2D 方法: 提供 4 和 8 个连通域分析。
cc3d github 地址[2]
如何安装
pip install connected-components-3d
测试
测试代码及地址[3]
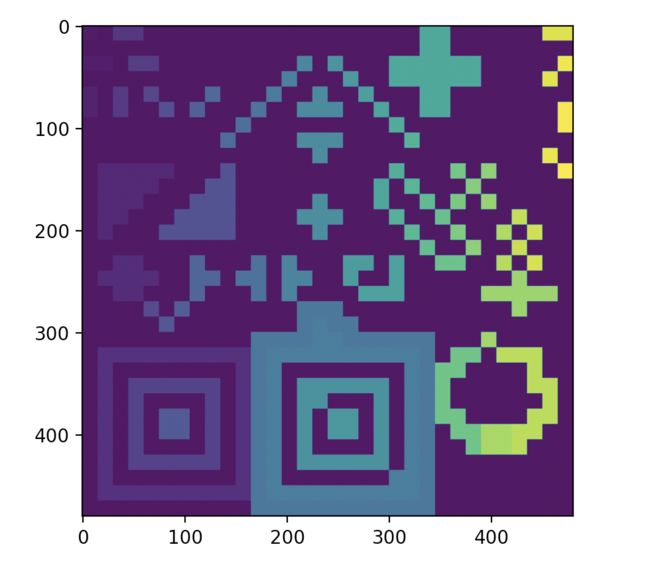
比如二维图像:
原始图像有 [0, 31, 199] 3个值 背景是0,绿色是31, 199是紫色。
img = np.array(Image.open('./testing_img/test2d.png'))[:,:,0]
print(np.unique(img))
labels = cc3d.connected_components(labels, connectivity=4)
使用4邻域后,图片多了很多种颜色,每种颜色都代表一个区域,一共有78个区域。
划分成不同区域了,自然能提取出想要的区域。比如把 区域像素<阈值的置为0,从而去除假阳性。或者只保留前两个最大的区域,其余置为0.
最后,附上我真实处理三维数据的代码
import cc3d
import nibabel as nib
from pathlib2 import Path
from tqdm import tqdm
import numpy as np
import os
def main(data, output):
data = Path(data).resolve()
output = Path(output).resolve()
assert data != output, f'postprocess data will replace original data, use another output path'
if not output.exists():
output.mkdir(parents=True)
predictions = sorted(data.glob('*_seg.nii.gz'))
for pred in tqdm(predictions):
if not pred.name.startswith('.'):
vol_nii = nib.load(str(pred))
affine = vol_nii.affine
vol = vol_nii.get_fdata()
vol = post_processing(vol)
vol_nii = nib.Nifti1Image(vol, affine)
vol_nii_filename = output / pred.name
vol_nii.to_filename(str(vol_nii_filename))
def post_processing(vol):
vol_ = vol.copy()
vol_[vol_ > 0] = 1
vol_ = vol_.astype(np.int64)
vol_cc = cc3d.connected_components(vol_)
cc_sum = [(i, vol_cc[vol_cc == i].shape[0]) for i in range(vol_cc.max() + 1)]
cc_sum.sort(key=lambda x: x[1], reverse=True)
cc_sum.pop(0) # remove background
reduce_cc = [cc_sum[i][0] for i in range(1, len(cc_sum)) if cc_sum[i][1] < cc_sum[0][1] * 0.1]
for i in reduce_cc:
vol[vol_cc == i] = 0
return vol
if __name__ == '__main__':
data = 'output/' # 分割结果地址,图像为nii.gz
output = 'output_remove/' # 移除假阳性后保存地址
if not os.path.exists(output):
os.makedirs(output)
main(data, output)
总结:这种方法较为复杂,但是可以实现 3d 去除假阳性。
3 sitk 提供的方法
这种方法也是首先进行连通成分分析,切分成不同的连通域。
如何安装 pip install SimpleITK
3.1 连通成分分析
sitk_maskimg = sitk.ReadImage('X.nii.gz', sitk.sitkUInt8)
# 其中sitk.sitkUInt8必须注明,否则使用 sitk.ConnectedComponent 报错
import SimpleITK as sitk
cc = sitk.ConnectedComponent(sitk_maskimg)
stats = sitk.LabelIntensityStatisticsImageFilter()
stats.Execute(cc, sitk_maskimg)
maxlabel = 0 # 获取最大连通域的索引
maxsize = 0 # 获取最大连通域的体素大小
# 遍历每一个连通域, 获取最大连通域的体素大小和索引
for l in stats.GetLabels(): # stats.GetLabels() (1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12)
size = stats.GetPhysicalSize(l) # stats.GetPhysicalSize(5)=75 表示第5个连通域的体素有75个
if maxsize < size:
maxlabel = l
maxsize = size
这样通过连通成分分析,获得了不同大小的连通域。至于怎么删除假阳性,可以根据自己的需求来。比如只保留最大的一个连通域,保留最大的两个连通域,保留体素不少于100的连通域等等
这里,我们通过一个比率来删除假阳性,假设最大的连通域有 x个 体素, 如果连通域的体素 < rate * x, rate设置为0.5,则被删除。
not_remove = []
for l in stats.GetLabels():
size = stats.GetPhysicalSize(l)
if size >= maxsize * rate: # 判断体素个数
not_remove.append(l)
labelmaskimage = sitk.GetArrayFromImage(cc)
outmask = labelmaskimage.copy()
outmask[labelmaskimage != maxlabel] = 0
for i in range(len(not_remove)):
outmask[labelmaskimage == not_remove[i]] = 1
# 这里的 outmask 就是最后保留的连通域
到这里已经得到了删除了部分假阳性的图像矩阵,最后提供一个完整的代码,包括如何保存处理后的图像。
# 通过连通成分分析,移除小区域
import SimpleITK as sitk
import os
import argparse
from pathlib import Path
def RemoveSmallConnectedCompont(sitk_maskimg, rate=0.5):
'''
two steps:
step 1: Connected Component analysis: 将输入图像分成 N 个连通域
step 2: 假如第 N 个连通域的体素小于最大连通域 * rate,则被移除
:param sitk_maskimg: input binary image 使用 sitk.ReadImage(path, sitk.sitkUInt8) 读取,
其中sitk.sitkUInt8必须注明,否则使用 sitk.ConnectedComponent 报错
:param rate: 移除率,默认为0.5, 小于 1/2最大连通域体素的连通域被移除
:return: binary image, 移除了小连通域的图像
'''
# step 1 Connected Component analysis
cc = sitk.ConnectedComponent(sitk_maskimg)
stats = sitk.LabelIntensityStatisticsImageFilter()
stats.Execute(cc, sitk_maskimg)
maxlabel = 0 # 获取最大连通域的索引
maxsize = 0 # 获取最大连通域的体素大小
# 遍历每一个连通域, 获取最大连通域的体素大小和索引
for l in stats.GetLabels(): # stats.GetLabels() (1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12)
size = stats.GetPhysicalSize(l) # stats.GetPhysicalSize(5)=75 表示第5个连通域的体素有75个
if maxsize < size:
maxlabel = l
maxsize = size
# step 2 获取每个连通域的大小,保留 size >= maxsize * rate 的连通域
not_remove = []
for l in stats.GetLabels():
size = stats.GetPhysicalSize(l)
if size >= maxsize * rate:
not_remove.append(l)
labelmaskimage = sitk.GetArrayFromImage(cc)
outmask = labelmaskimage.copy()
outmask[labelmaskimage != maxlabel] = 0
for i in range(len(not_remove)):
outmask[labelmaskimage == not_remove[i]] = 1
# 保存图像
outmask = outmask.astype('float32')
out = sitk.GetImageFromArray(outmask)
out.SetDirection(sitk_maskimg.GetDirection())
out.SetSpacing(sitk_maskimg.GetSpacing())
out.SetOrigin(sitk_maskimg.GetOrigin()) # 使 out 的层厚等信息同输入一样
return out # to save image: sitk.WriteImage(out, 'largecc.nii.gz')
if __name__ == '__main__':
parser = argparse.ArgumentParser(description="remove small connected domains")
parser.add_argument('--input', type=str, default="./123.nii.gz")
parser.add_argument("--output", type=str, default='./123.nii.gz')
args = parser.parse_args()
# for single image
sitk_maskimg = sitk.ReadImage(args.input, sitk.sitkUInt8)
out = RemoveSmallConnectedCompont(sitk_maskimg, rate=0.5) # 可以设置不同的比率
sitk.WriteImage(out, args.output)
使用 python sitkcc3d.py --input ./largecc.nii.gz --output ./largecc1.nii.gz
参考资料
[1]
参考链接: https://www.jianshu.com/p/a9cc11af270c
[2]
cc3d github 地址: https://github.com/seung-lab/connected-components-3d/tree/2da2547570ba5dc83fda9b17e68ea34b9fdc01d1
[3]
测试代码及地址: https://github.com/seung-lab/connected-components-3d/tree/2da2547570ba5dc83fda9b17e68ea34b9fdc01d1/manual_testing
[1]
参考链接:https://www.jianshu.com/p/a9cc11af270c
[2]
cc3d github 地址:https://github.com/seung-lab/connected-components-3d/tree/2da2547570ba5dc83fda9b17e68ea34b9fdc01d1
[3]
测试代码及地址:https://github.com/seung-lab/connected-components-3d/tree/2da2547570ba5dc83fda9b17e68ea34b9fdc01d1/manual_testing
深度学习,分割后处理之通过填补孔洞,提高分割准确度
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连 ![]()