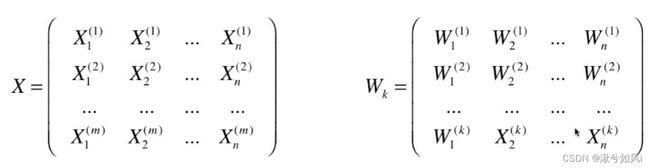机器学习之PCA
一、PCA基本介绍
PCA(Principal Components Analysis)-主成分分析算法
- 用于数据降维、可视化、去噪
- 非监督学习算法
二、PCA工作原理
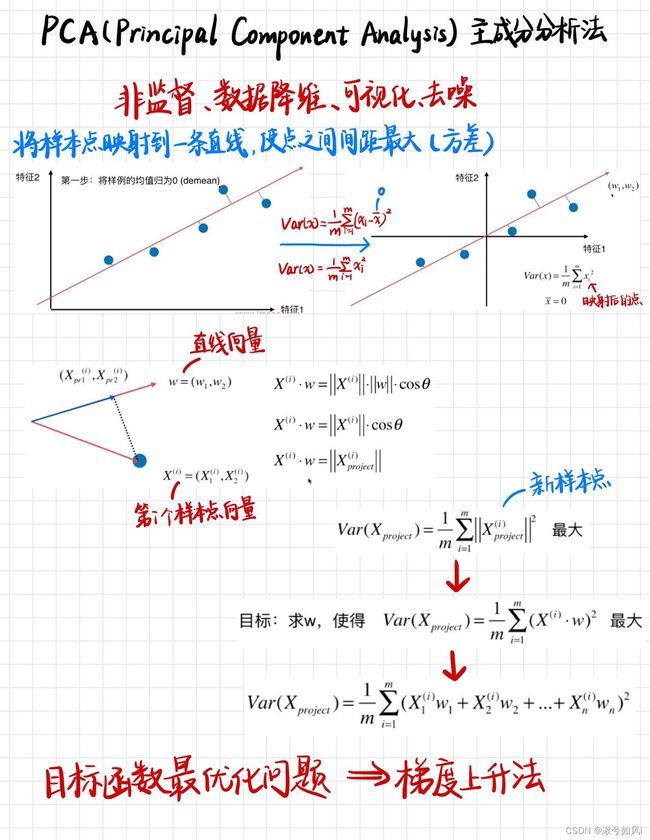
目标:每次将空间中多维的样本点映射到一条直线上(要保证样本点之间的间距最大,用方差衡量,即保证样本点的特征丢失得最少),从而实现降了1维。重复上面操作,得到许多条直线,其实就是得到一个新的坐标轴。然后将所有样本点映射到新的坐标轴上,即实现了数据降维,并且最好地保留了样本特征。
将上图进行步骤总结:
- 将所有样本点的均值归零化(每一个样本点的特征都减去该特征对应的均值)(为了方便求方差)
- 将方差中的每一部分,转化成直线向量与样本点向量的点积(均值为0,坐标轴中心是原点)
- 将方差设为一个目标函数,转化成了目标函数最优化(越大越好)问题,可使用梯度上升法解决
- 求出梯度,通过梯度上升法得到第一主成分w1,是一个向量
- 对所有样本点,利用向量减法去掉第一主成分w1上的分量
- 重复上述操作找出第二主成分w2,以此类推
- 最后得出主成分矩阵,即新的坐标轴
- 通过矩阵相乘,将样本点数据映射到新的坐标轴上,最终实现降维
三、sklearn实现PCA
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
digits = datasets.load_digits()
X = digits.data
y = digits.target
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=666)
# X_train.shape --> (1347, 64)
from sklearn.neighbors import KNeighborsClassifier
knn_clf = KNeighborsClassifier()
knn_clf.fit(X_train, y_train)
print(knn_clf.score(X_test, y_test)) # 0.9866666666666667 精度高但64维数据训练时间长
# 数据降维
from sklearn.decomposition import PCA
pca = PCA(n_components=2) # 降到2维
pca.fit(X_train)
X_train_reduction = pca.transform(X_train) # 将训练数据集降维
X_test_reduction = pca.transform(X_test) # 将测试数据集也降维
knn_clf = KNeighborsClassifier()
knn_clf.fit(X_train_reduction, y_train)
print(knn_clf.score(X_test_reduction, y_test)) # 0.6066666666666667 精度低但时间快很多
"""
pca.explained_variance_ratio_ 返回每一个主成分能解释的方差所占比例,主成分越多和越接近1
array([1.45668166e-01, 1.37354688e-01, 1.17777287e-01, 8.49968861e-02,
5.86018996e-02, 5.11542945e-02, 4.26605279e-02, 3.60119663e-02,
3.41105814e-02, 3.05407804e-02, 2.42337671e-02, 2.28700570e-02,
1.80304649e-02, 1.79346003e-02, 1.45798298e-02, 1.42044841e-02,
1.29961033e-02, 1.26617002e-02, 1.01728635e-02, 9.09314698e-03,
8.85220461e-03, 7.73828332e-03, 7.60516219e-03, 7.11864860e-03,
6.85977267e-03, 5.76411920e-03, 5.71688020e-03, 5.08255707e-03,
4.89020776e-03, 4.34888085e-03, 3.72917505e-03, 3.57755036e-03,
3.26989470e-03, 3.14917937e-03, 3.09269839e-03, 2.87619649e-03,
2.50362666e-03, 2.25417403e-03, 2.20030857e-03, 1.98028746e-03,
1.88195578e-03, 1.52769283e-03, 1.42823692e-03, 1.38003340e-03,
1.17572392e-03, 1.07377463e-03, 9.55152460e-04, 9.00017642e-04,
5.79162563e-04, 3.82793717e-04, 2.38328586e-04, 8.40132221e-05,
5.60545588e-05, 5.48538930e-05, 1.08077650e-05, 4.01354717e-06,
1.23186515e-06, 1.05783059e-06, 6.06659094e-07, 5.86686040e-07,
1.71368535e-33, 7.44075955e-34, 7.44075955e-34, 7.15189459e-34])
"""
pca = PCA(n_components=2) 可以指定主成分数量即降到几维。降到2维方便数据可视化,也极大提高了训练速度,但会导致精度下降很多,因为丢失了很多维度的特征。可以通过分析pca.explained_variance_ratio_返回的数组,将其可视化查看多少维主成分保留信息较多,然后选择降到合适的维度。
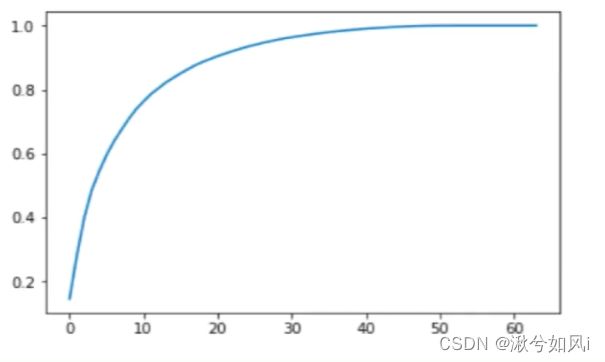
sklearn中的PCA封装了这一过程,可直接指定一个比例,保留这个比例的特征信息。
n_components是>=1的整数时,表示期望PCA降维后的特征维度数n_components是[0,1]的小数时,表示主成分的方差和所占的最小比例阈值,PCA类自己去根据样本特征方差来决定降维到的维度
pca = PCA(n_components=0.95)
pca.fit(X_train)
X_train_reduction = pca.transform(X_train) # 降维训练集
X_test_reduction = pca.transform(X_test) # 降维测试集
knn_clf = KNeighborsClassifier()
knn_clf.fit(X_train_reduction, y_train)
print(knn_clf.score(X_test_reduction, y_test)) # 0.98
print(pca.n_components_) # 28
输出结果,我们可以打印一下pca.n_components_,发现保留了28个主成分即压缩成28维就能保留95%的样本信息,精度高达0.98,和训练全部样本的精度所差无几并且极大地减少了训练时间!
总结
主成分分析目的是用较少的变量来代替原来较多的变量,并可以反映原来多个变量的大部分信息。
但经过运算之后的主成分特征维度的含义具有模糊性,解释性差(我们最多可以理解成主成分只是由原来的坐标维度线性相加的结果,但加出来之后它到底是啥就不好说了)。
问题
- 待完善