医学图像分割常见评价指标(单目标)——包含源码讲解和指标缺陷
单目标分割常见评价指标
-
- 1 知道4个常见指标,TP,TN,FP,FN
- 2 评价分割区域准确率
-
- 2.1 Recall Sensitivity TPR(True Positive Rate)
- 2.2 Specificity (True Negative Rate)
- 2.3 Precision (PPV)
- 2.4 Dice Coefficient
- 2.5 Jaccard Coefficient (IOU)
- 3 对分割出的边界进行评价
-
- 3.1 Hausdorff_95
- 3.2 持续更新中
- 4 源码
-
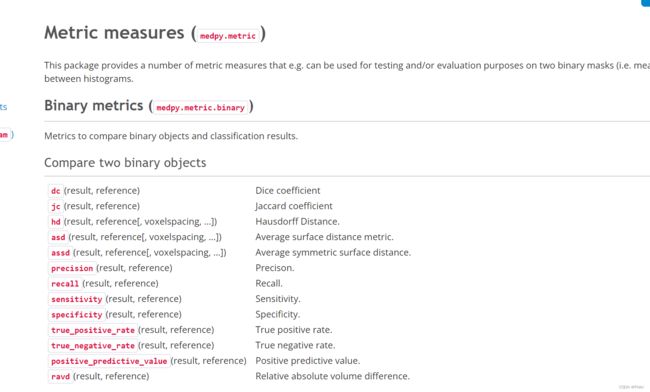
- 4.1 直接第三方包使用
- 4.2 手写代码
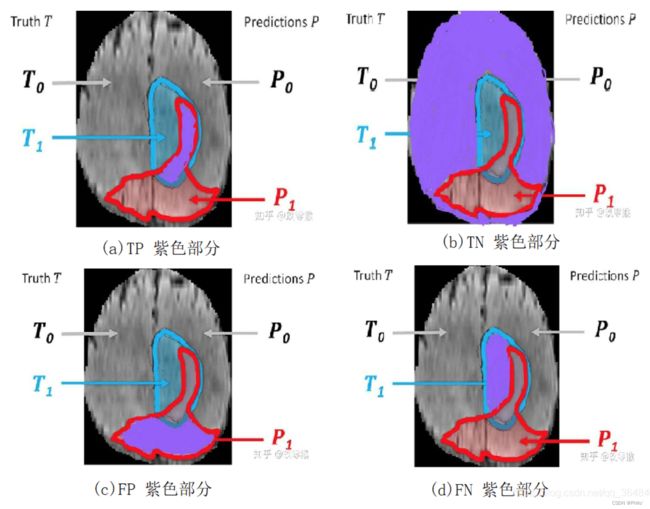
1 知道4个常见指标,TP,TN,FP,FN
- TP:True Positive,被判定为正样本,事实上也是正样本 ,即蓝色与红色的交集
- TN:True Negative,被判定为负样本,事实上也是负样本,即红色与蓝色以外区域
- FP:False Positive,被判定为正样本,但事实上是负样本,即红色中除了蓝色部分
- FN:False Negative,被判定为负样本,但事实上是正样本,即蓝色中除了红色部分
2 评价分割区域准确率
2.1 Recall Sensitivity TPR(True Positive Rate)
定义:

白话:
就是预测的区域中真正是目标区域的面积占据总目标区域的比例,1最好,0最拉;
优点:
可以清晰的得到正确的分割区域占据真区域的面积比例。
缺陷:
指标高不高?高,分割好不好?不好,这就是缺陷。
2.2 Specificity (True Negative Rate)
定义:
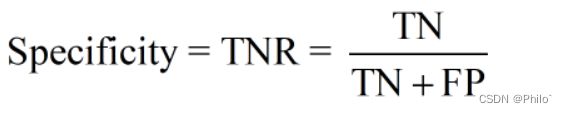
白话:
是Recall的另外一面,针对单目标分割,Recall和Specificity就是一个转置,也是1最好,0最拉。
优点:
可以从侧面印证自己分割的区域外面积目标区域外的总面积比例
缺陷:

假设绿色100,红色10, 蓝色20, 则TN为0.81多,指标如何,还撮合,实际一塌糊涂
2.3 Precision (PPV)
定义:

白话:
实际预测正确的部分占据预测部分总面积的比例,也是1最好,0最拉。
优点:
可以很清晰的知道自己预测正确的部分占据自己预测部分的比例
缺陷:
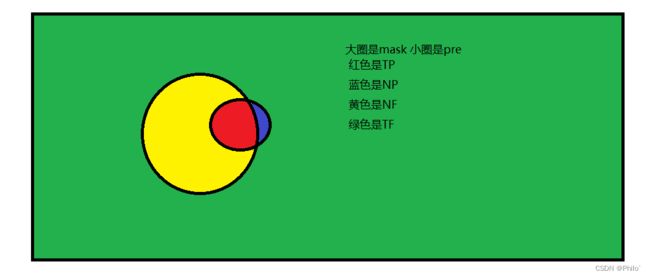
PPV高不高?很高了,红色占据整个小圈的面积比例很高,但是分割效果好不好?不好。
2.4 Dice Coefficient
定义:

白话:
就是两个物体相交的面积占总面积的比值,也是1最好,0最拉。
优点:
看着公式大家自己都能说出来,主要是我再想,为什么要用2TP呢?直接上下两个都是TP也是能看出指标的啊!
缺陷:

缺陷:
这里和Recall的缺陷比较相似,但是引入了FP,会稍微中和掉recall的缺陷,但是也是可以看出这个指标也是可以的,但实际效果分割效果也是不太好的。
2.5 Jaccard Coefficient (IOU)
白话:
和Dice很相似,也回答了我之前的疑惑,哈哈,是可以用TP的,就是换一个名字,也可以计算出重叠区域指标的,也是1最好,0最拉。
优点:
参考Dice
缺陷:
参考Dice
以上的所有参考指标都是有所侧重的,因此存在一些固有缺陷,这种缺陷是比较特殊的例子,这是没啥问题的,在评价分割效果的时候可以多用几个,如果几个指标都好,那就证明分割效果确实很好,如果有的指标很好,有的很差,就可以自己动手画一下图,看看是那种特殊情况了。
3 对分割出的边界进行评价
3.1 Hausdorff_95
定义:

就是计算两个边界之间的距离,具体计算过程不太清除,但是能get到点,这个值越大越差,越小越好。
3.2 持续更新中
4 源码
4.1 直接第三方包使用
pip install medpy
使用:
from medpy import metric
def calculate_metric_percase(pred, gt):
dice = metric.binary.dc(pred, gt)
jc = metric.binary.jc(pred, gt)
hd = metric.binary.hd95(pred, gt)
asd = metric.binary.asd(pred, gt)
return dice, jc, hd, asd
重点是,这里传入的pred和gt从模型中预测出来后,需要使用pred.cpu().detach().numpy(),将数据转化为cpu上的numpy数据类型,同时在单目标分割中,
传入的数据需要做二值化处理,否则出现的数据有问题,具体参看下面手写的代码。
4.2 手写代码
- 以下代码都是基于predict在传入的时候已经二值化处理了,且target也已经是二值矩阵
- 以下代码在自己的模型中都是跑过的,是没问题的,重点是如何将其用到自己的模型中
- 重点在于如何理解指标的实现,帮助理解这些评价指标的计算过程
Recall:
def recall(predict, target): #Sensitivity, Recall, true positive rate都一样
if torch.is_tensor(predict): # 模型本身有sigmoid函数
predict = predict.data.cpu().numpy()
if torch.is_tensor(target):
target = target.data.cpu().numpy()
total_recall = 0.0
for i in range(len(predict[0])): # 因为存在batch_size通道,需要切割一下
pre_split = predict[i]
tar_split = target[i]
pre_split = numpy.atleast_1d(pre_split.astype(numpy.bool)) # 非0为True 0为False
tar_split = numpy.atleast_1d(tar_split.astype(numpy.bool))
tp = numpy.count_nonzero(pre_split & tar_split) # 计算tp
fn = numpy.count_nonzero(~pre_split & tar_split) # 计算fn
try:
recall = tp / float(tp + fn) # 根据recall公式计算recall
except ZeroDivisionError:
recall = 0.0
total_recall += recall
return total_recall/len(predict[0]) # 进行均值化处理
Dice:
def dice(predict, target):
if torch.is_tensor(predict):
predict = predict.data.cpu().numpy()
if torch.is_tensor(target):
target = target.data.cpu().numpy()
total_dice = 0.0
for i in range(len(predict[0])):
pre_split = predict[i]
tar_split = target[i]
pre_split = numpy.atleast_1d(pre_split.astype(numpy.bool))
tar_split = numpy.atleast_1d(tar_split.astype(numpy.bool))
intersection = numpy.count_nonzero(pre_split & tar_split) #计算非零个数
size_i1 = numpy.count_nonzero(pre_split) # 计算gt面积
size_i2 = numpy.count_nonzero(tar_split) # 这里直接计算pre面积
try:
dice = 2. * intersection / float(size_i1 + size_i2)
except ZeroDivisionError:
dice = 0.0
total_dice += dice
return total_dice/len(predict[0])
IOU:
def iou(predict, target):
if torch.is_tensor(predict):
predict = predict.data.cpu().numpy()
if torch.is_tensor(target):
target = target.data.cpu().numpy()
total_iou = 0.0
for i in range(len(predict[0])):
pre_split = predict[i]
tar_split = target[i]
pre_split = numpy.atleast_1d(pre_split.astype(numpy.bool))
tar_split = numpy.atleast_1d(tar_split.astype(numpy.bool))
tp = numpy.count_nonzero(pre_split & tar_split)
fn = numpy.count_nonzero(~pre_split & tar_split)
fp = numpy.count_nonzero(~tar_split & pre_split)
try:
iou = tp / float(tp + fn + fp)
except ZeroDivisionError:
iou = 0.0
total_iou += iou
return total_iou/len(predict[0])
最近一直在弄医学图像分割模型,特此记录,有错误大家可以积极讨论,我也会不断改进的!