脑肿瘤分割学习(4):Multi-step Cascaded Networks for Brain Tumor Segmentation
Multi-step Cascaded Networks for Brain Tumor Segmentation[多步级联的脑肿瘤分割网络]
- Abstract
- 1 Introduction
- 2. Methodology
-
- 2.1 Multi-step Cascaded Network
- 2.2 3D U-Net Architecture with Deep Supervisions
- 3. Experiments
-
- 3.1 Preprocessing
- 3.2 Implementation Details
-
- 3.3 Segmentation Results
- 4. Disussing and Conclusion
- 写在前面,这个文章类似于BraTS2019冠军的风格,其实就是一个多步集成的网络。
Abstract
- 还是老样子,开始先介绍整个工作的重要性
- 脑肿瘤自动分割方法在脑肿瘤诊断和治疗的整个过程中起着极其重要的作用。
- 然后顺势推出我们的工作是啥
- 在本文中,我们提出了一种多步级联网络,该网络考虑了脑肿瘤子结构的层次拓扑结构,并对子结构进行了从粗到细的分割。
- 在分割过程中,将前一步的结果作为下一步的先验信息,以指导更精细的分割过程。整个网络都以端到端的方式进行训练。【从这里来看,这个结构和BraTS2019冠军方案思想极其相似,或者这是一个多阶段分割的主流思想与步骤】
- 此外,为了缓解梯度消失问题和减少过拟合度,我们在每一步增加了几个辅助输出作为一种深度监督,并分别引入了几种数据增强策略,对脑肿瘤分割证明是非常有效的。【似乎这种过深的模型都会有深度监督这个东西】
- 最后,利用focal loss损失来解决肿瘤区域和背景明显不平衡的问题。【这个可以考虑一下】
- 结果
- 我们的模型在Brats 2019年验证数据集上进行了测试,对于整个肿瘤、肿瘤核心和增强型肿瘤,平均骰子系数的初步结果分别为0.886、0.813和0.771。代码可在https://github.com/JohnleeHIT/Brats2019上找到【但是从这里来看的话,其实这个模型的整体精度并没有很高】
1 Introduction
- 介绍脑肿瘤分割的重要作用与意义,以及胶质瘤的一些背景情况,主要还是介绍做脑肿瘤分割的临床意义。
- 脑肿瘤是最严重的脑部疾病之一,其中恶性胶质瘤是最常见的类型。根据严重程度,胶质瘤可简单分为两类:侵袭性胶质瘤(即HGG),平均预期寿命近2年;温和胶质瘤(即LGG),平均预期寿命数年。
- 由于其死亡率相当高,因此对胶质瘤的早期诊断具有重要意义,这大大提高了治疗概率,尤其是对于LGG。目前,治疗胶质瘤最可能的方法是手术、化疗和放疗。
- 对于任何一种治疗策略,治疗前后都必须对病变区域进行准确的成像和分割,以评估特定策略的有效性。
- 介绍为啥会用到MRI来评估脑肿瘤,其实就是变相介绍数据集
- 在所有现有的成像仪器中,MRI以其高分辨率、高对比度和目前未知的健康威胁成为脑肿瘤分析的首选。
- 在目前的临床常规中,手动分割大量的MRI图像是一种常见的做法,结果证明这非常耗时,而且评分员容易出错。因此,提出一种自动分割方法将具有巨大的潜在价值。
- 开始介绍目前在自动脑肿瘤分割上的一些研究情况
- 许多研究人员提出了几种基于深度学习或机器学习的有效方法来解决这个问题。
- 在这些提出的方法中,Zikic等人[1]使用浅层CNN网络对以滑动窗口方式从MRI数据体捕获的2D图像块进行分类。
- Zhao等人[2]将3D肿瘤分割任务转换为3维平面的2D分割,并通过裁剪不同大小的patches的方式引入多尺度。[三个平面的2D,这种做法就是所谓的2.5D吧]
- Havaei等人[3]提出了一种级联卷积网络,可以同时捕获局部和全局信息。[本文主要思想可能来自于这个点]
- Cicek等人[4]将传统的2D U-net分割网络扩展到3D实现,使体积分割成为体素方式。
- Kamnitsas等人[5]提出了一种名为DeepMedic的双路径3D卷积网络,以整合多尺度上下文信息,并使用3D全连接CRF作为后处理方法来细化分割结果。
- Chen等人[6]对DeepMedic进行了改进,首先从原始DeepMedic中选择的多层中裁剪3D补丁,然后合并这些补丁,以在网络中学习更多信息,此外,在网络中引入深度监督,以更好地传播梯度。[多尺度带来的提升?]
- Ma等人[7]采用特征表示学习策略,通过使用特定于模态的随机森林作为特征学习核,有效地从多模态图像中探索局部和上下文信息,用于组织分割。
- 许多研究人员提出了几种基于深度学习或机器学习的有效方法来解决这个问题。
- 介绍本文思想的来源
- 受Havaei和Cicek的启发,我们提出了一种多步级联网络来分割脑肿瘤亚结构。该网络以3D U-net作为基本的分割结构,整个网络从粗到细,可以看作是一种空间注意机制。【?这里的说法可能比较牵强】
2. Methodology
2.1 Multi-step Cascaded Network
- 介绍什么是多步级联网络,以及这种设置的理由
- 所提出的多级级联网络如图2所示,该方法以从粗到细的方式分割肿瘤亚结构的层次结构。
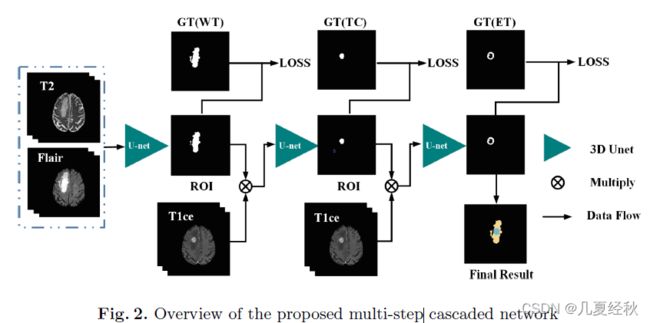
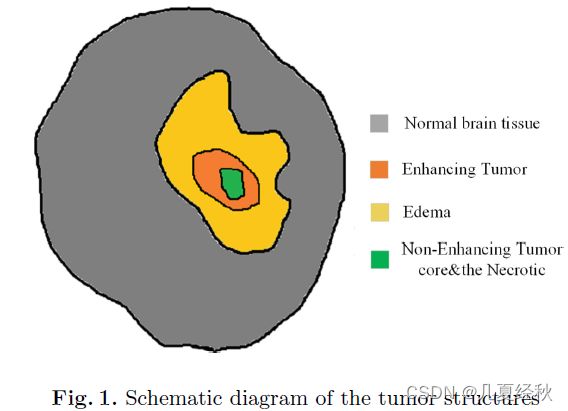
- 在第一步中,为了与在[8]中详细描述的手动注释协议保持一致,使用了MRI肿瘤体素的两种模式(FLAIR和T1CE)。然后将两个通道的数据体送入第一分割网络以粗略分割整个肿瘤(WT),它包含了脑瘤的所有亚结构;
- 在第二步中,同样地,我们选择T1ce通道作为数据源对肿瘤核心结构进行分割。此外,第一个粗略步骤的结果可以被用作第二个步骤的先验信息。通过将第一步生成的掩码与T1ce体素相乘,第二分割网络将更集中于相应的掩码区域,从而更容易分割TC结构。然后由第二网络对被掩蔽的卷进行处理,从而引入TC结构(前景)【这里的相乘操作其实就是把区域缩小到第一阶段的结果内进行搜索,以得到第二阶段的结果】
- 在最后一步,也是最精细的一步,通过同样的策略,我们也可以从数据体中得到增强的肿瘤(ET)亚结构,最后将这三个步骤的结果结合在一起,得到最终的脑肿瘤分割图。【只用到了四个模态中的三个模态】
- 所提出的多级级联网络如图2所示,该方法以从粗到细的方式分割肿瘤亚结构的层次结构。
2.2 3D U-Net Architecture with Deep Supervisions
- 介绍自己的model,拥有深度监督的model,作为一个这么深的网络,如果不用深度监督估计很难训练的好
- 在我们的多步级联网络中,我们采用了3D U-Net的一种变体作为基本的分割结构,如图3所示。典型的3D U-Net网络由两个路径组成:收缩途径和扩张途径。【其实也就是所谓的编码和解码】
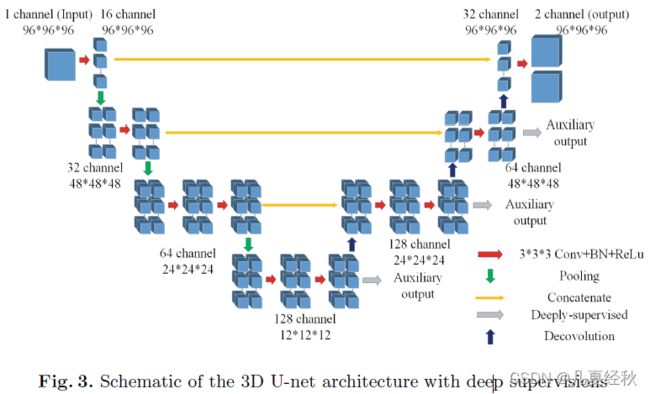
- 收缩路径主要用于对输入体素进行编码,并引入层次化特征,而扩展路径则用于对收缩路径中编码的信息进行解码
- 这两条路径采用跳跃连接,使得网络能够同时捕获局部和全局信息【这个说法有点牵强,只能说跳跃连接可以带来更多的信息,同时弥补一定的下采样和上采样损失,我个人如此理解】
- 我们的基本分割网络以3D U-Net为原型,并在此基础上进行了一些改进。3D U-Net与所提出的基本分割网络的主要区别如下:
- 1.与传统的3D U-net结构相比,我们提出的基本分割网络在扩展路径中引入了三个辅助输出,目的是为了更好地传播梯度,并降低相对较深的分割网络的梯度消失的概率。因此,对于基本的分割过程,我们需要最小化包括主分支和辅助损失函数的总体损失函数。【这里难不成是主分支和辅助分支的损失函数之和作为整体的损失函数】
- 2.我们将focal 损失[9]引入整个训练过程的损失函数,目的是减轻训练数据中正负样本的显着不平衡。焦点损失可表示如下:
F L ( p t ) = − α t ( 1 − p t ) γ log ( p t ) p t = { p if y = 1 1 − p otherwise \begin{gathered} \mathrm{FL}\left(p_{\mathrm{t}}\right)=-\alpha_{\mathrm{t}}\left(1-p_{\mathrm{t}}\right)^{\gamma} \log \left(p_{\mathrm{t}}\right) \\ p_{\mathrm{t}}= \begin{cases}p & \text { if } y=1 \\ 1-p & \text { otherwise }\end{cases} \end{gathered} FL(pt)=−αt(1−pt)γlog(pt)pt={p1−p if y=1 otherwise
这里 p ∈ [ 0 , 1 ] p \in[0,1] p∈[0,1] 是带有标注的类为 y = 1 \mathrm{y}=1 y=1 的模型估计概率 . γ ⩾ 0 \gamma \geqslant 0 γ⩾0 代表调整focal的参数, 它可以顺畅地调整简单示例的权重降低比率. α t \alpha_{t} αt 指的是平衡正负样本重要性的平衡因子。
- 在我们的多步级联网络中,我们采用了3D U-Net的一种变体作为基本的分割结构,如图3所示。典型的3D U-Net网络由两个路径组成:收缩途径和扩张途径。【其实也就是所谓的编码和解码】
3. Experiments
3.1 Preprocessing
- 介绍一些BraTS数据的预处理方法(归一化)
- 本文以BRATS 2019数据集为训练数据,包括259个HGG和76个LGG MRI卷,提供四种模式(T1、T2、T1CE和FLAIR)。根据数据集的官方声明,所有数据集都已按照相同的注释协议进行了手动分割。
- 此外,还对这些数据集进行了一些预处理操作,例如,所有MRI体积都被联合配准到相同的解剖模板上,内插到相同的分辨率,并剥离了头骨。
- 然而,由于MRI设备的不完善和患者的特殊性,图像数据中存在强度不均匀,也称为偏场,因此需要对原始数据集进行额外的预处理。这种强度不均匀或偏场对训练过程有很大影响。为了消除偏置场效应,人们提出了多种校正方法。
- 在所提出的偏置场校正方法中,最有效的是N4偏置场校正[14]。本文将N4偏场校正法作为分割前的一个重要的预处理步骤。最后,我们还使用归一化方法将所有数据归一化为单位方差为零的均值。[有必要了解一下这个啥叫N4偏置场校正,最后这个归一化感觉大体上就是减去均值然后除以标准差]
3.2 Implementation Details
- 主要讲一下model的实现工具与实现细节
- 我们混合了Brats 2019训练数据集中的所有数据,包括HGG和LGG,然后用混合数据集训练我们的模型。
- 在训练过程中,我们首先通过得到包含大脑的最大矩形来从体素中提取大脑区域。然后,由于内存的限制,我们将原始数据卷随机裁剪成子卷,并根据经验选择补丁的大小为969696。在训练过程中,我们在每次迭代中从患者的数据量中提取一个补丁。
- 而在测试阶段,对于单个数据体,我们将子体素按顺序排列,以便根据预测重建整个数据体,并且patches的大小与训练过程中的相同。
- 我们为每个患者数据得到不同数量的补丁,因为我们从体积中提取的大脑区域是不同的。
- 为了减少过拟合度,我们引入了一些数据增强的方法,如随机旋转角度,水平和垂直翻转,以及以一定的概率对子体积添加高斯模糊。事实证明,数据增强对于脑肿瘤分割任务是非常重要的,因为网络容易在训练数据相对较少的情况下过度拟合。
- 我们使用ADAM优化器来更新网络的权重。最初的学习率被设置为0.001,当损失曲线趋于平坦时,学习率下降到0.0005。在整个训练过程中,将批次大小设置为1。
- 我们的模型在NVIDIA RTX 2080钛图形处理器上进行了50个周期的训练,大约需要13小时。
3.3 Segmentation Results
- 这里主要是介绍评价指标
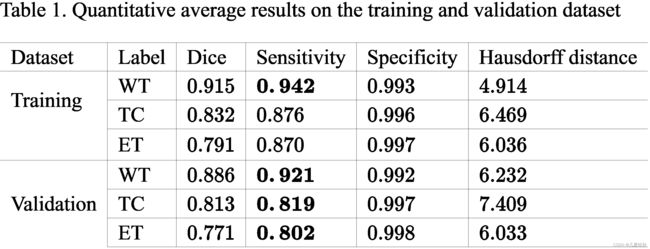
- 为了对我们提出的方法进行评估,我们在训练集和验证集上测试了我们的算法,通过将推理结果上传到在线评估平台(CBICB的IPP),最终得到了分别针对整个肿瘤(WT)、肿瘤核心(TC)和增强肿瘤(ET)的Dice sore、Hausdorff距离、敏感度和特异度的评估结果。上述指标的定义如下: Dice ( P , T ) = ∣ P 1 ∧ T 1 ∣ ( ∣ P 1 ∣ + ∣ T 1 ∣ ) / 2 Sensitivity ( P , T ) = ∣ P 1 ∧ T 1 ∣ ∣ T 1 ∣ Specificity ( P , T ) = ∣ P 0 ∧ T 0 ∣ ∣ T 0 ∣ Haus ( P , T ) = max { sup p ∈ ∂ P 1 inf t ∈ ∂ T 1 d ( p , t ) , sup t ∈ ∂ T 1 inf p ∈ ∂ P 1 d ( t , p ) } \begin{aligned} &\operatorname{Dice}(P, T)=\frac{\left|P_{1} \wedge T_{1}\right|}{\left(\left|P_{1}\right|+\left|T_{1}\right|\right) / 2}\\ &\text { Sensitivity }(P, T)=\frac{\left|P_{1} \wedge T_{1}\right|}{\left|T_{1}\right|}\\ &\operatorname{Specificity}(P, T)=\frac{\left|P_{0} \wedge T_{0}\right|}{\left|T_{0}\right|}\\ &\operatorname{Haus}(P, T)=\max \left\{\sup _{p \in \partial P_{1}} \inf _{t \in \partial T_{1}} d(p, t), \sup _{t \in \partial T_{1}} \inf _{p \in \partial P_{1}} d(t, p)\right\} \end{aligned} Dice(P,T)=(∣P1∣+∣T1∣)/2∣P1∧T1∣ Sensitivity (P,T)=∣T1∣∣P1∧T1∣Specificity(P,T)=∣T0∣∣P0∧T0∣Haus(P,T)=max{p∈∂P1supt∈∂T1infd(p,t),t∈∂T1supp∈∂P1infd(t,p)}
- 这里 P P P代表算法的预测图, T T T 经过专家手动分割的真实值(label). ∧ \wedge ∧ 逻辑与操作, ∣ ⋅ ∣ |\cdot| ∣⋅∣ 代表集合中体素的数量, P 1 , F 0 P_{1}, F_{0} P1,F0分别代表预测图中正的体素以及负的体素, T 1 , T 0 T_{1}, T_{0} T1,T0 分别代表真实标签中的正值和负值。, d ( p , t ) d(p, t) d(p,t) 代表两个点p,t之间的距离 p , t . ∂ P 1 p, t . \partial P_{1} p,t.∂P1 是预测集 P 1 P_{1} P1 的表面, ∂ T 1 \partial T_{1} ∂T1 是真实标签集 T 1 T_{1} T1的表面.
- 介绍结果
- 分析算法的性能
- 为了更好的分析提出算法的整体性能,我们将所有的验证集和训练集结果画了 ,如图4所示。显然,该方法可以很好地分割两个数据集中的几乎所有体积,除了少数离群点。
- 此外,通过比较验证数据集和训练数据集的盒图,我们注意到验证数据集的骰子系数、敏感度、特异度和Hausdorff距离等评价指标的方差都大于训练数据集的方差,这意味着我们的方法在一定程度上仍然存在过拟合问题。
- 最后,从四个子图中我们可以看到,对于训练和验证数据集,整个肿瘤的骰子系数的方差都小于肿瘤核心和增强的肿瘤亚结构,对于敏感度和Hausdorff距离度量是相同的,而对于特异性度量是相反的,这符合我们的预期。
- 然而,最令我们惊讶的是,对于这两个数据集,在大多数指标上,肿瘤核心(TC)的方差都大于增强肿瘤核心(ET)的方差,最可能的解释是网络有时会受到LGG肿瘤样本的影响而错误地预测整个肿瘤作为肿瘤核心,这使得方差急剧增加。
- 结果的可视化
4. Disussing and Conclusion
-
介绍一下可视化的分析
- 通过可视化所有验证结果,我们发现有趣的是,肿瘤核心区域的大量不良分割病例是那些将整个肿瘤误认为肿瘤核心区域的病例。
- 最可能的解释可能是尽管模式相同,不同 MRI 体素之间的差异。因此,如果在训练过程之前采取了一些可以减少这些变化的预处理方法,结果可能会增加,例如直方图均衡。
-
介绍一下训练策略上的总结
- 此外,我们还尝试了逐步训练网络而不是端到端训练的课程学习策略,结果结果并不比端到端训练的效果好。
- 这很可能是因为如果网络中的所有参数都可以更新,网络可以更好地拟合训练数据。
- 最后,我们尝试对级联网络的三个步骤进行加权,令人惊讶的是,我们发现最终结果对于训练步骤的增量、减量甚至权重都没有太大差异。
-
最后对全文进行总结
- 总之,我们提出了一个非常有效的多步网络来分割所有的肿瘤子结构。
- 我们首先为每个步骤选择特定的模态,以使自动分割过程与哺乳动物协议保持一致,与使用所有模态的方法相比,这大大提高了我们的结果。
- 之后,我们使用 N4 偏置场校正和归一化对输入体积进行预处理。
- 由于内存限制,我们从原始数据中随机裁剪体积块并在这些块上引入数据增强,我们发现数据增强对于减少过度拟合非常重要,尤其是在训练数据稀缺时。
- 最后,训练补丁在多步网络中进行训练,这已被证明比单步网络更有效,因为它以粗到细的方式训练网络并将棘手的多分类问题分为三个更容易二进制分类问题。我们在 BraTS 2019 验证数据集上评估了所提出的方法,结果表明我们的方法在所有三个子结构上都表现良好。
-
个人总结
- 本文的多步级联其实就是一种基于区域的分割策略,其实目的主要是将多类分割转为多个2类分割问题,可以在一定程度上提升整体的精度,但是这种多步级联的方式会带来较大的显存消耗以及难以训练,所以作者用了深度监督思想进行辅助训练。整体训练成本偏高,可以尝试采用一拖多的单一网络进行改进。
- 同时根据数据并没有采用全部的模态数据,我目前观察到的是T1数据并不能从肉眼上看出来对于整个分割有何影响,后续可以尝试以下剔除T1数据看看效果是否会更好。