- 如何修改Python安装路径
壹只小小码农
python学习开发语言
在安装软件时,很多人都会发现默认的安装路径不是他们想要的,于是就想要修改安装路径。那么如何修改安装路径呢?本文将从多个角度为大家进行分析。一、在安装向导中更改一般情况下,我们在安装软件时会看到安装向导,其中会有一个“安装路径”选项,我们可以在这里手动更改安装路径。不同软件的安装向导可能略有不同,但是一般都会有这个选项。二、使用修改器有些软件虽然没有提供修改安装路径的选项,但是我们可以使用一些修改器
- Python中类基础知识详解和应用
点云SLAM
Pythonpython开发语言深度学习人工智能计算机视觉python中的类学习
Python类知识详解类的定义语法class类名:#类体(属性、方法)示例:classPerson:pass创建类的实例(对象)p=Person()#创建一个类的对象(实例)类的构造方法(__init__)__init__是类的构造函数,在实例化对象时自动调用,用于初始化属性。classPerson:def__init__(self,name,age):self.name=nameself.age
- python+requests+excel 接口测试
鱼鱼说测试
postmanpython开发语言
1、EXCEL文件接口保存方式,如图。2、然后就是读取EXCEL文件中的数据方法,如下:1importxlrd234classreadExcel(object):5def__init__(self,path):6self.path=path78@property9defgetSheet(self):10#获取索引11xl=xlrd.open_workbook(self.path)12sheet=x
- Flask(二) 路由routes
@昵称不存在
Flaskflask
文章目录基本路由定义路由参数路由规则设置请求方法(GET/POST)路由函数返回静态文件和模板Blueprint(模块化路由)显示当前所有路由Flask路由是Web应用程序中将URL映射到Python函数的机制。定义路由:使用@app.route(‘/path’)装饰器定义URL和视图函数的映射。路由参数:通过动态部分在URL中传递参数。路由规则:使用类型转换器指定URL参数的类型。请求方法:指定
- python中random中uniform怎么用_Python中的random.uniform()函数教程与实例解析
weixin_39763640
random.uniform()函数教程与实例解析1.uniform()函数说明random.uniform(x,y)方法将随机生成一个实数,它在[x,y]范围内。2.uniform()的语法与参数2.1语法#_*_coding:utf-8_*_importrandomrandom.uniform(x,y)或#_*_coding:utf-8_*_fromrandomimportuniformuni
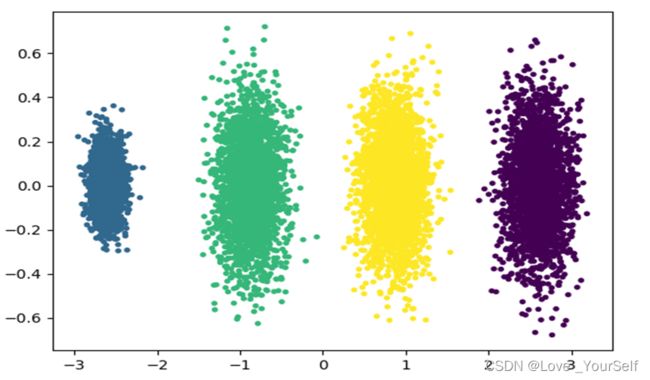
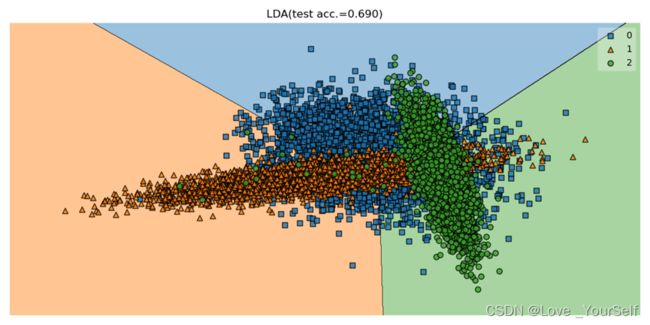
- Python实例题:基于 KNN 算法的手写数字识别
目录Python实例题题目要求:解题思路:代码实现:Python实例题题目基于KNN算法的手写数字识别要求:实现一个基于K-NearestNeighbors(KNN)算法的手写数字识别系统。支持以下功能:使用MNIST数据集训练和测试模型实现KNN分类算法可视化手写数字样本评估模型性能(准确率、混淆矩阵等)添加用户交互界面,允许用户绘制数字并进行识别。解题思路:使用sklearn加载MNIST数据
- Python实例题:基于遗传算法的旅行商问题求解
狐凄
实例python开发语言
目录Python实例题题目要求:解题思路:代码实现:Python实例题题目基于遗传算法的旅行商问题求解要求:使用遗传算法解决旅行商问题(TSP)。支持以下功能:随机生成城市坐标或导入预定义城市实现遗传算法的基本操作(选择、交叉、变异)可视化进化过程和最终路径统计进化过程中的适应度变化允许用户调整遗传算法参数(种群大小、迭代次数、交叉率、变异率等)。解题思路:用列表表示城市访问顺序作为染色体。使用欧
- Python Flask Web教程004:Flask 变量规则
若北辰
flaskpython前端
FlaskWeb教程004:Flask变量规则1.Flask变量规则2.实例3.转换器构建规则4.规范的URL5.路由尾部有无斜杠的区别路由尾部斜杠的影响推荐使用带尾斜杠的路由结论1.Flask变量规则通过向规则参数添加变量部分,可以动态构建URL。此变量部分标记为。它作为关键字参数传递给与规则相关联的函数。2.实例在以下示例中,route()装饰器的规则参数包含附加到URL'/hello’的。因
- Club_IntelliMatch_Development_Guide
Joseit
pythonpythonpygamedjangoflask
ClubIntelliMatch系统-全栈开发流程文档概述ClubIntelliMatch系统是一个现代化的社团活动智能匹配平台,采用前后端分离架构。系统基于PythonFlask构建RESTfulAPI后端,Vue.js3+Vite构建现代化前端,MySQL作为持久化数据存储。本文档深入分析了整个开发流程的技术架构、设计原则和实现细节。系统架构流程图后端API架构前端组件架构app.pyFlas
- Python实例题:基于 Flask 的博客系统
狐凄
实例python开发语言
目录Python实例题题目要求:解题思路:代码实现:1.base.html2.index.html3.post.html4.create_post.html5.login.html6.register.htmlPython实例题题目基于Flask的博客系统要求:使用Flask框架构建一个简单的博客系统。实现用户认证(注册、登录、注销)。支持博客文章的创建、编辑、删除和查看。使用SQLite数据库存
- Python助力自动驾驶:深度学习模型优化全攻略
Echo_Wish
Python!实战!python自动驾驶深度学习
Python助力自动驾驶:深度学习模型优化全攻略说起自动驾驶,大家第一反应往往是“高精地图”“传感器融合”“路径规划”等等,背后真正的“大脑”其实是各式各样的深度学习模型。它们负责感知环境、识别路况、预测行为,甚至实时做出决策。可是,跑在车上的这些模型不仅要精准,还得轻量、实时、稳定,这可不是简单的“丢GPU就能解决”的问题。今天,咱们就从Python开发者的视角,聊聊自动驾驶里深度学习模型的优化
- Ansible部署MySQL实操
码农运维知识
运维mysqlansiblemysql
一、Ansible概述Ansible是一款开源的自动化运维工具,由MichaelDeHaan于2012年创建,2015年被红帽(RedHat)收购(收购金额超1亿美元)。它基于Python开发,通过SSH协议实现远程节点管理,无需在被控端安装任何客户端代理(Agentless)。这种设计使其成为轻量级、易部署的自动化解决方案,特别适合批量系统配置、应用程序部署和任务编排等场景。核心特点无代理架构:
- AI绘画背后的技术:Stable Diffusion原理详解与实战
AI学长带你学AI
ai
AI绘画背后的技术:StableDiffusion原理详解与实战关键词:StableDiffusion、扩散模型、AI绘画、潜在空间、文本生成图像摘要:本文将带你揭开AI绘画“魔法”背后的核心技术——StableDiffusion的神秘面纱。我们会用“给小学生讲故事”的方式,从生活中的例子出发,逐步解释扩散模型的底层逻辑、StableDiffusion的关键创新,并用Python代码实战演示如何生
- matplotlib 绘制热力图
扶子
pythonmatplotlib绘图代码matplotlibpython经验分享热力图
1、功能介绍:使用了matplotlib和seaborn两个python库来创建并显示一个热力图。热力图是一种通过颜色变化来表示二维表格数据集中值分布的图形,适合用于展示矩阵数据或数据分析结果中的模式和趋势。2、代码部分:importmatplotlib.pyplotaspltimportseabornassnsimportnumpyasnp#设置中文字体plt.rcParams['font.sa
- open3d 使用 RANSAC 算法拟合平面
扶子
python点云处理平面pythonopen3d经验分享点云拟合平面
1、功能介绍:一个python代码演示了如何使用open3d和numpy来完成一个完整的点云平面拟合任务。它包括以下几个主要部分:生成符合某一平面方程的随机点云数据、使用RANSAC算法对这些点云进行平面拟合、可视化原始点云和平面拟合结果2、代码部分:importnumpyasnpimportopen3daso3d#生成随机点云np.random.seed(42)n_points=100#假设这些
- pycharm——djiango之数据迁移,终端操作
Pop–
python
首先在pycharm中找到terminal(终端),输入指令:pythonmanage.pymakemigrations之后你会看到如下图:这表示创建成功。接着输入指令:pythonmanage.pymigrate就能看到好多ok,你在数据库中也能看到很多表你可以在终端打开数据库查看表,也可以使用客户端的可视化界面查看,还可以在pycharm中右边的database里边打开查看,如下图:之后你就可
- python 百度云api_Python使用百度API上传文件到百度网盘代码分享
weixin_39775577
python百度云api
#coding:UTF-8importurllibimporturllib2__author__='Administrator'fromposter.encodeimportmultipart_encodefromposter.streaminghttpimportregister_openersregister_openers()defupload(fileName):"""通过百度开发者API
- Flask入门基础1
浅清陌
Flaskflaskpython后端
1Flask简介Flask诞生于2010年,是Arminronacher(阿明·罗纳彻)用Python语言基于Werkzeug工具箱编写的轻量级Web开发框架。Flask本身相当于一个内核,其他几乎所有的功能都要用到扩展(邮件扩展Flask-Mail,用户认证Flask-Login,数据库Flask-SQLAlchemy),都需要用第三方的扩展来实现。比如可以用Flask扩展加入ORM、窗体验证工
- C++封装python调用库
技术大白
c++开发语言
传结构体中间用空字符串问题使用callback传输结构体,中间出现\0字符,使用std::vector类型voidPyProcessInterface::ProcessContent(constchar*buff,UINT32size,boolfromSelf){if(callback){std::vectordataVec(buff,buff+size);callback(std::move(d
- 量化价值投资入门:Fama-French三因子模型详解与实战应用
量化价值投资入门到精通
ai
量化价值投资入门:Fama-French三因子模型详解与实战应用关键词:量化投资、Fama-French三因子模型、价值投资、因子投资、资产定价、Python实现、投资组合管理摘要:本文深入解析Fama-French三因子模型的理论基础、数学原理和实际应用。作为现代金融学最重要的资产定价模型之一,三因子模型通过市场因子、规模因子和价值因子解释股票收益差异。我们将从模型起源开始,详细讲解其数学表达和
- Python操作百度网盘指南
weixin_47233946
编程python开发语言
##介绍百度网盘是中国流行的云存储服务,通过API可以实现自动化操作。本指南介绍如何使用Python操作百度网盘,包括上传、下载、管理文件等功能。##准备工作###1.获取百度网盘开发者权限1.访问[百度开发者中心](https://pan.baidu.com/union/home)2.注册开发者账号并创建应用3.获取API密钥(AppKey和SecretKey)###2.安装必要的Python库
- python读取sas数据集_SASpy模块,利用Python操作SAS
SASpy模块打通了Python与SAS之间的连接。有了SASpy模块,我们就能够在Python中操控SAS。本文将首先介绍SASpy模块的一些基本方法,最后通过一个聚类分析的例子,来展示如何在Python中调用SAS的机器学习过程,以及对聚类结果的可视化。SASpy模块特点1、需要Python3.X及以上,SAS9.4及以上,需要Java环境;2、无论是本地SAS还是远程服务器上的SAS,都可以
- 从数据抓取到分析:用Python爬虫获取、清洗与可视化数据
程序员威哥
python爬虫c++
在数据科学领域,数据的获取、清洗与分析是整个数据处理过程中的关键步骤。随着互联网上数据的不断增多,使用Python爬虫抓取网站数据并进行分析已成为数据科学家和分析师的常见任务。本篇文章将通过具体的实例,展示如何使用Python从零开始抓取数据,清洗数据,并进行数据分析和可视化。1.数据抓取:用Python爬虫获取网页数据1.1选择爬虫工具Python提供了多个强大的爬虫框架和库,常用的工具包括:r
- Python基础(吃洋葱小游戏)
aaiier
pythonpygame开发语言
下面我将为你设计一个"吃洋葱小游戏"的Python实现方案,使用Pygame库开发。这个游戏模拟吃洋葱的过程,玩家需要收集不同种类的洋葱以获得高分,同时避免吃到辣椒。吃洋葱小游戏-Python实现方案1.游戏设计概念游戏目标:玩家控制角色吃掉尽可能多的洋葱获得高分核心机制:洋葱从屏幕上方随机下落玩家左右移动角色接住洋葱不同洋葱有不同分值(普通洋葱+10,红洋葱+20,紫洋葱+50)辣椒会扣减生命值
- 模拟工作队列 - 华为OD机试真题(JavaScript卷)
什码情况
算法面试javascript数据结构华为od
华为OD机试题库《C++》限时优惠9.9华为OD机试题库《Python》限时优惠9.9华为OD机试题库《JavaScript》限时优惠9.9针对刷题难,效率慢,我们提供一对一算法辅导,针对个人情况定制化的提高计划(全称1V1效率更高)。看不懂有疑问需要答疑辅导欢迎私VX:code5bug题目描述让我们来模拟一个工作队列的运作,有一个任务提交者和若干任务执行者,执行者从1开始编号。提交者会在给定的时
- 数据分类 - 华为OD机试真题(JavaScript 题解)
什码情况
华为odjavascript开发语言数据结构算法机试
华为OD机试题库《C++》限时优惠9.9华为OD机试题库《Python》限时优惠9.9华为OD机试题库《JavaScript》限时优惠9.9针对刷题难,效率慢,我们提供一对一算法辅导,针对个人情况定制化的提高计划(全称1V1效率更高)。看不懂有疑问需要答疑辅导欢迎私VX:code5bug题目描述对一个数据a进行分类,分类方法为:此数据a(四个字节大小)的四个字节相加对一个给定的值b取模,如果得到的
- odrive软件的版本
m0_55305757
stm32电机嵌入式硬件odrive
odrive软件的版本0.4.0通信方面引入一个fibre变复杂了(节点还是手工生成的),cpp程序开始变多了。(sensorless我看到变成独立文件了)pythontool开始使用pip安装形式。0.5.0开始支持spi的encoder,as5047之类0.5.1据说之后的版本controlloop开始变化0.5.2开始应该是大修改了//RequiredtouseOC4forADCtrigge
- 说说自己Python 代码优化实践
chilavert318
大数据linux运维python
今年上半年在外省做一个大数据相关的项目,在review项目组成员的代码时,发现一段处理大数据集的模块存在明显性能瓶颈:10万条数据的清洗流程耗时近20分钟,CPU占用率却始终在30%以下。深入分析后发现,看似简洁的Python代码背后,隐藏着诸多可以优化的细节——这并非个例,我们的程序在追求代码可读性时,往往忽略了Python特有的性能陷阱。今天抽点时间,从我实践中的代码就python开发,从内存
- ROS学习笔记5:常用API和模块导入
前言本人ROS小白,利用寒假时间学习ROS,在此以笔记的方式记录自己每天的学习过程。争取写满15篇(5/15)。环境:Ubuntu20.04、ROS1:noetic环境配置:严格按照下方学习链接的教程配置,基本一次成功。学习链接:【Autolabor初级教程】ROS机器人入门对应链接文档:ROS机器人入门课程《ROS理论与实践》笔记绝大部分代码使用Python语言编写。本期关键词:初始化,话题服务
- 一个简单测试Deepseek吞吐量的脚本,国内环境可跑
谢平康
深度学习pytorch人工智能
一个简单测试Deepseek吞吐量的脚本,这里用DeepSeek-R1-Distill-Qwen-32B,支持单卡409024G可跑,具体看你的硬件情况做调整,理论支持所有的模型,看你需要,可以修改模型名称,重点是pip使用国内的源,模型下载用阿里的ModelScope,无障碍下载,使用.最后可以生成一个txt与html报表.前提是你安装了python与python-venv,你可以不用venv来
- sql统计相同项个数并按名次显示
朱辉辉33
javaoracle
现在有如下这样一个表:
A表
ID Name time
------------------------------
0001 aaa 2006-11-18
0002 ccc 2006-11-18
0003 eee 2006-11-18
0004 aaa 2006-11-18
0005 eee 2006-11-18
0004 aaa 2006-11-18
0002 ccc 20
- Android+Jquery Mobile学习系列-目录
白糖_
JQuery Mobile
最近在研究学习基于Android的移动应用开发,准备给家里人做一个应用程序用用。向公司手机移动团队咨询了下,觉得使用Android的WebView上手最快,因为WebView等于是一个内置浏览器,可以基于html页面开发,不用去学习Android自带的七七八八的控件。然后加上Jquery mobile的样式渲染和事件等,就能非常方便的做动态应用了。
从现在起,往后一段时间,我打算
- 如何给线程池命名
daysinsun
线程池
在系统运行后,在线程快照里总是看到线程池的名字为pool-xx,这样导致很不好定位,怎么给线程池一个有意义的名字呢。参照ThreadPoolExecutor类的ThreadFactory,自己实现ThreadFactory接口,重写newThread方法即可。参考代码如下:
public class Named
- IE 中"HTML Parsing Error:Unable to modify the parent container element before the
周凡杨
html解析errorreadyState
错误: IE 中"HTML Parsing Error:Unable to modify the parent container element before the child element is closed"
现象: 同事之间几个IE 测试情况下,有的报这个错,有的不报。经查询资料后,可归纳以下原因。
- java上传
g21121
java
我们在做web项目中通常会遇到上传文件的情况,用struts等框架的会直接用的自带的标签和组件,今天说的是利用servlet来完成上传。
我们这里利用到commons-fileupload组件,相关jar包可以取apache官网下载:http://commons.apache.org/
下面是servlet的代码:
//定义一个磁盘文件工厂
DiskFileItemFactory fact
- SpringMVC配置学习
510888780
springmvc
spring MVC配置详解
现在主流的Web MVC框架除了Struts这个主力 外,其次就是Spring MVC了,因此这也是作为一名程序员需要掌握的主流框架,框架选择多了,应对多变的需求和业务时,可实行的方案自然就多了。不过要想灵活运用Spring MVC来应对大多数的Web开发,就必须要掌握它的配置及原理。
一、Spring MVC环境搭建:(Spring 2.5.6 + Hi
- spring mvc-jfreeChart 柱图(1)
布衣凌宇
jfreechart
第一步:下载jfreeChart包,注意是jfreeChart文件lib目录下的,jcommon-1.0.23.jar和jfreechart-1.0.19.jar两个包即可;
第二步:配置web.xml;
web.xml代码如下
<servlet>
<servlet-name>jfreechart</servlet-nam
- 我的spring学习笔记13-容器扩展点之PropertyPlaceholderConfigurer
aijuans
Spring3
PropertyPlaceholderConfigurer是个bean工厂后置处理器的实现,也就是BeanFactoryPostProcessor接口的一个实现。关于BeanFactoryPostProcessor和BeanPostProcessor类似。我会在其他地方介绍。PropertyPlaceholderConfigurer可以将上下文(配置文件)中的属性值放在另一个单独的标准java P
- java 线程池使用 Runnable&Callable&Future
antlove
javathreadRunnablecallablefuture
1. 创建线程池
ExecutorService executorService = Executors.newCachedThreadPool();
2. 执行一次线程,调用Runnable接口实现
Future<?> future = executorService.submit(new DefaultRunnable());
System.out.prin
- XML语法元素结构的总结
百合不是茶
xml树结构
1.XML介绍1969年 gml (主要目的是要在不同的机器进行通信的数据规范)1985年 sgml standard generralized markup language1993年 html(www网)1998年 xml extensible markup language
- 改变eclipse编码格式
bijian1013
eclipse编码格式
1.改变整个工作空间的编码格式
改变整个工作空间的编码格式,这样以后新建的文件也是新设置的编码格式。
Eclipse->window->preferences->General->workspace-
- javascript中return的设计缺陷
bijian1013
JavaScriptAngularJS
代码1:
<script>
var gisService = (function(window)
{
return
{
name:function ()
{
alert(1);
}
};
})(this);
gisService.name();
&l
- 【持久化框架MyBatis3八】Spring集成MyBatis3
bit1129
Mybatis3
pom.xml配置
Maven的pom中主要包括:
MyBatis
MyBatis-Spring
Spring
MySQL-Connector-Java
Druid
applicationContext.xml配置
<?xml version="1.0" encoding="UTF-8"?>
&
- java web项目启动时自动加载自定义properties文件
bitray
javaWeb监听器相对路径
创建一个类
public class ContextInitListener implements ServletContextListener
使得该类成为一个监听器。用于监听整个容器生命周期的,主要是初始化和销毁的。
类创建后要在web.xml配置文件中增加一个简单的监听器配置,即刚才我们定义的类。
<listener>
<des
- 用nginx区分文件大小做出不同响应
ronin47
昨晚和前21v的同事聊天,说到我离职后一些技术上的更新。其中有个给某大客户(游戏下载类)的特殊需求设计,因为文件大小差距很大——估计是大版本和补丁的区别——又走的是同一个域名,而squid在响应比较大的文件时,尤其是初次下载的时候,性能比较差,所以拆成两组服务器,squid服务于较小的文件,通过pull方式从peer层获取,nginx服务于较大的文件,通过push方式由peer层分发同步。外部发布
- java-67-扑克牌的顺子.从扑克牌中随机抽5张牌,判断是不是一个顺子,即这5张牌是不是连续的.2-10为数字本身,A为1,J为11,Q为12,K为13,而大
bylijinnan
java
package com.ljn.base;
import java.util.Arrays;
import java.util.Random;
public class ContinuousPoker {
/**
* Q67 扑克牌的顺子 从扑克牌中随机抽5张牌,判断是不是一个顺子,即这5张牌是不是连续的。
* 2-10为数字本身,A为1,J为1
- 翟鸿燊老师语录
ccii
翟鸿燊
一、国学应用智慧TAT之亮剑精神A
1. 角色就是人格
就像你一回家的时候,你一进屋里面,你已经是儿子,是姑娘啦,给老爸老妈倒怀水吧,你还觉得你是老总呢?还拿派呢?就像今天一样,你们往这儿一坐,你们之间是什么,同学,是朋友。
还有下属最忌讳的就是领导向他询问情况的时候,什么我不知道,我不清楚,该你知道的你凭什么不知道
- [光速与宇宙]进行光速飞行的一些问题
comsci
问题
在人类整体进入宇宙时代,即将开展深空宇宙探索之前,我有几个猜想想告诉大家
仅仅是猜想。。。未经官方证实
1:要在宇宙中进行光速飞行,必须首先获得宇宙中的航行通行证,而这个航行通行证并不是我们平常认为的那种带钢印的证书,是什么呢? 下面我来告诉
- oracle undo解析
cwqcwqmax9
oracle
oracle undo解析2012-09-24 09:02:01 我来说两句 作者:虫师收藏 我要投稿
Undo是干嘛用的? &nb
- java中各种集合的详细介绍
dashuaifu
java集合
一,java中各种集合的关系图 Collection 接口的接口 对象的集合 ├ List 子接口 &n
- 卸载windows服务的方法
dcj3sjt126com
windowsservice
卸载Windows服务的方法
在Windows中,有一类程序称为服务,在操作系统内核加载完成后就开始加载。这里程序往往运行在操作系统的底层,因此资源占用比较大、执行效率比较高,比较有代表性的就是杀毒软件。但是一旦因为特殊原因不能正确卸载这些程序了,其加载在Windows内的服务就不容易删除了。即便是删除注册表中的相 应项目,虽然不启动了,但是系统中仍然存在此项服务,只是没有加载而已。如果安装其他
- Warning: The Copy Bundle Resources build phase contains this target's Info.plist
dcj3sjt126com
iosxcode
http://developer.apple.com/iphone/library/qa/qa2009/qa1649.html
Excerpt:
You are getting this warning because you probably added your Info.plist file to your Copy Bundle
- 2014之C++学习笔记(一)
Etwo
C++EtwoEtwoiterator迭代器
已经有很长一段时间没有写博客了,可能大家已经淡忘了Etwo这个人的存在,这一年多以来,本人从事了AS的相关开发工作,但最近一段时间,AS在天朝的没落,相信有很多码农也都清楚,现在的页游基本上达到饱和,手机上的游戏基本被unity3D与cocos占据,AS基本没有容身之处。so。。。最近我并不打算直接转型
- js跨越获取数据问题记录
haifengwuch
jsonpjsonAjax
js的跨越问题,普通的ajax无法获取服务器返回的值。
第一种解决方案,通过getson,后台配合方式,实现。
Java后台代码:
protected void doPost(HttpServletRequest req, HttpServletResponse resp)
throws ServletException, IOException {
String ca
- 蓝色jQuery导航条
ini
JavaScripthtmljqueryWebhtml5
效果体验:http://keleyi.com/keleyi/phtml/jqtexiao/39.htmHTML文件代码:
<!DOCTYPE html>
<html xmlns="http://www.w3.org/1999/xhtml">
<head>
<title>jQuery鼠标悬停上下滑动导航条 - 柯乐义<
- linux部署jdk,tomcat,mysql
kerryg
jdktomcatlinuxmysql
1、安装java环境jdk:
一般系统都会默认自带的JDK,但是不太好用,都会卸载了,然后重新安装。
1.1)、卸载:
(rpm -qa :查询已经安装哪些软件包;
rmp -q 软件包:查询指定包是否已
- DOMContentLoaded VS onload VS onreadystatechange
mutongwu
jqueryjs
1. DOMContentLoaded 在页面html、script、style加载完毕即可触发,无需等待所有资源(image/iframe)加载完毕。(IE9+)
2. onload是最早支持的事件,要求所有资源加载完毕触发。
3. onreadystatechange 开始在IE引入,后来其它浏览器也有一定的实现。涉及以下 document , applet, embed, fra
- sql批量插入数据
qifeifei
批量插入
hi,
自己在做工程的时候,遇到批量插入数据的数据修复场景。我的思路是在插入前准备一个临时表,临时表的整理就看当时的选择条件了,临时表就是要插入的数据集,最后再批量插入到数据库中。
WITH tempT AS (
SELECT
item_id AS combo_id,
item_id,
now() AS create_date
FROM
a
- log4j打印日志文件 如何实现相对路径到 项目工程下
thinkfreer
Weblog4j应用服务器日志
最近为了实现统计一个网站的访问量,记录用户的登录信息,以方便站长实时了解自己网站的访问情况,选择了Apache 的log4j,但是在选择相对路径那块 卡主了,X度了好多方法(其实大多都是一样的内用,还一个字都不差的),都没有能解决问题,无奈搞了2天终于解决了,与大家分享一下
需求:
用户登录该网站时,把用户的登录名,ip,时间。统计到一个txt文档里,以方便其他系统调用此txt。项目名
- linux下mysql-5.6.23.tar.gz安装与配置
笑我痴狂
mysqllinuxunix
1.卸载系统默认的mysql
[root@localhost ~]# rpm -qa | grep mysql
mysql-libs-5.1.66-2.el6_3.x86_64
mysql-devel-5.1.66-2.el6_3.x86_64
mysql-5.1.66-2.el6_3.x86_64
[root@localhost ~]# rpm -e mysql-libs-5.1