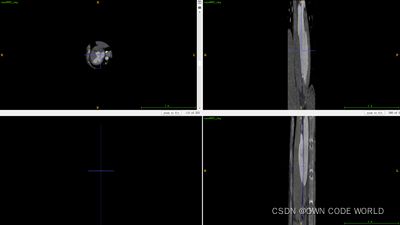
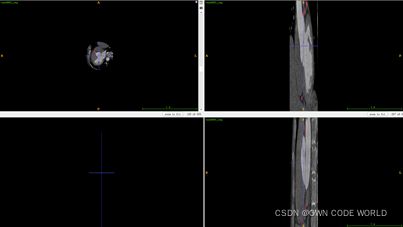
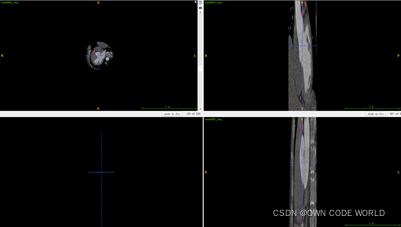
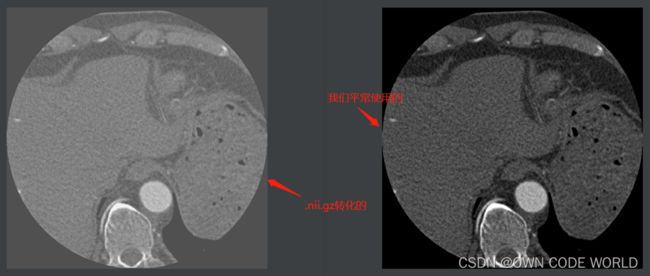
- 系统学习Python——并发模型和异步编程:进程、线程和GIL
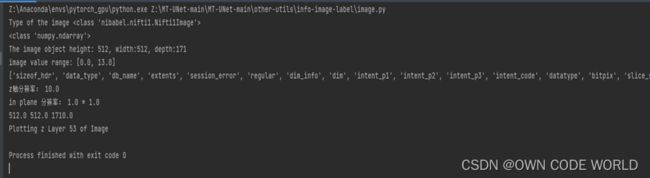
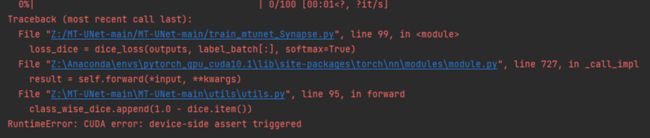
分类目录:《系统学习Python》总目录在文章《并发模型和异步编程:基础知识》我们简单介绍了Python中的进程、线程和协程。本文就着重介绍Python中的进程、线程和GIL的关系。Python解释器的每个实例都是一个进程。使用multiprocessing或concurrent.futures库可以启动额外的Python进程。Python的subprocess库用于启动运行外部程序(不管使用何种
- Flask框架入门:快速搭建轻量级Python网页应用
「已注销」
python-AIpython基础网站网络pythonflask后端
转载:Flask框架入门:快速搭建轻量级Python网页应用1.Flask基础Flask是一个使用Python编写的轻量级Web应用框架。它的设计目标是让Web开发变得快速简单,同时保持应用的灵活性。Flask依赖于两个外部库:Werkzeug和Jinja2,Werkzeug作为WSGI工具包处理Web服务的底层细节,Jinja2作为模板引擎渲染模板。安装Flask非常简单,可以使用pip安装命令
- Python Flask 框架入门:快速搭建 Web 应用的秘诀
Python编程之道
Python人工智能与大数据Python编程之道pythonflask前端ai
PythonFlask框架入门:快速搭建Web应用的秘诀关键词Flask、微框架、路由系统、Jinja2模板、请求处理、WSGI、Web开发摘要想快速用Python搭建一个灵活的Web应用?Flask作为“微框架”代表,凭借轻量、可扩展的特性,成为初学者和小型项目的首选。本文将从Flask的核心概念出发,结合生活化比喻、代码示例和实战案例,带你一步步掌握:如何用Flask搭建第一个Web应用?路由
- 上位机知识篇---SD卡&U盘镜像
常用的镜像烧录软件balenaEtcherbalenaEtcher是一个开源的、跨平台的工具,用于将操作系统镜像文件(如ISO和IMG文件)烧录到SD卡和USB驱动器中。以下是其使用方法、使用场景和使用注意事项的介绍:使用方法下载安装:根据自己的操作系统,从官方网站下载对应的安装包。Windows系统下载.exe文件后双击安装;Linux系统若下载的是.deb文件,可在终端执行“sudodpkg-
- python_虚拟环境
阿_焦
python
第一、配置虚拟环境:virtualenv(1)pipvirtualenv>安装虚拟环境包(2)pipinstallvirtualenvwrapper-win>安装虚拟环境依赖包(3)c盘创建虚拟目录>C:\virtualenv>配置环境变量【了解一下】:(1)如何使用virtualenv创建虚拟环境a、cd到C:\virtualenv目录下:b、mkvirtualenvname>创建虚拟环境nam
- PyTorch & TensorFlow速成复习:从基础语法到模型部署实战(附FPGA移植衔接)
阿牛的药铺
算法移植部署pytorchtensorflowfpga开发
PyTorch&TensorFlow速成复习:从基础语法到模型部署实战(附FPGA移植衔接)引言:为什么算法移植工程师必须掌握框架基础?针对光学类产品算法FPGA移植岗位需求(如可见光/红外图像处理),深度学习框架是算法落地的"桥梁"——既要用PyTorch/TensorFlow验证算法可行性,又要将训练好的模型(如CNN、目标检测)转换为FPGA可部署的格式(ONNX、TFLite)。本文采用"
- Python爱心光波
系列文章序号直达链接Tkinter1Python李峋同款可写字版跳动的爱心2Python跳动的双爱心3Python蓝色跳动的爱心4Python动漫烟花5Python粒子烟花Turtle1Python满屏飘字2Python蓝色流星雨3Python金色流星雨4Python漂浮爱心5Python爱心光波①6Python爱心光波②7Python满天繁星8Python五彩气球9Python白色飘雪10Pyt
- Python流星雨
Want595
python开发语言
文章目录系列文章写在前面技术需求完整代码代码分析1.模块导入2.画布设置3.画笔设置4.颜色列表5.流星类(Star)6.流星对象创建7.主循环8.流星运动逻辑9.视觉效果10.总结写在后面系列文章序号直达链接表白系列1Python制作一个无法拒绝的表白界面2Python满屏飘字表白代码3Python无限弹窗满屏表白代码4Python李峋同款可写字版跳动的爱心5Python流星雨代码6Python
- Python之七彩花朵代码实现
PlutoZuo
Pythonpython开发语言
Python之七彩花朵代码实现文章目录Python之七彩花朵代码实现下面是一个简单的使用Python的七彩花朵。这个示例只是一个简单的版本,没有很多高级功能,但它可以作为一个起点,你可以在此基础上添加更多功能。importturtleastuimportrandomasraimportmathtu.setup(1.0,1.0)t=tu.Pen()t.ht()colors=['red','skybl
- Python 脚本最佳实践2025版
前文可以直接把这篇文章喂给AI,可以放到AI角色设定里,也可以直接作为提示词.这样,你只管提需求,写脚本就让AI来.概述追求简洁和清晰:脚本应简单明了。使用函数(functions)、常量(constants)和适当的导入(import)实践来有逻辑地组织你的Python脚本。使用枚举(enumerations)和数据类(dataclasses)等数据结构高效管理脚本状态。通过命令行参数增强交互性
- (Python基础篇)了解和使用分支结构
EternityArt
基础篇python
目录一、引言二、Python分支结构的类型与语法(一)if语句(单分支)(二)if-else语句(双分支)(三)if-elif-else语句(多分支)三、分支结构的应用场景(一)提示用户输入用户名,然后再提示输入密码,如果用户名是“admin”并且密码是“88888”则提示正确,否则,如果用户名不是admin还提示用户用户名不存在,(二)提示用户输入用户名,然后再提示输入密码,如果用户名是“adm
- (Python基础篇)循环结构
EternityArt
基础篇python
一、什么是Python循环结构?循环结构是编程中重复执行代码块的机制。在Python中,循环允许你:1.迭代处理数据:遍历列表、字典、文件内容等。2.自动化重复任务:如批量处理数据、生成序列等。3.控制执行流程:根据条件决定是否继续或终止循环。二、为什么需要循环结构?假设你需要打印1到100的所有偶数:没有循环:需手动编写100行print()语句。print(0)print(2)print(4)
- (Python基础篇)字典的操作
EternityArt
基础篇python开发语言
一、引言在Python编程中,字典(Dictionary)是一种极具灵活性的数据结构,它通过“键-值对”(key-valuepair)的形式存储数据,如同现实生活中的字典——通过“词语(键)”快速查找“释义(值)”。相较于列表和元组的有序索引访问,字典的优势在于基于键的快速查找,这使得它在处理需要频繁通过唯一标识获取数据的场景中极为高效。掌握字典的操作,能让我们更高效地组织和管理复杂数据,是Pyt
- Python七彩花朵
Want595
python开发语言
系列文章序号直达链接Tkinter1Python李峋同款可写字版跳动的爱心2Python跳动的双爱心3Python蓝色跳动的爱心4Python动漫烟花5Python粒子烟花Turtle1Python满屏飘字2Python蓝色流星雨3Python金色流星雨4Python漂浮爱心5Python爱心光波①6Python爱心光波②7Python满天繁星8Python五彩气球9Python白色飘雪10Pyt
- php SPOF
贵哥的编程之路(热爱分享 为后来者)
PHP语言经典程序100题php开发语言
1.什么是单点故障(SPOF)?单点故障指的是系统中某个组件一旦失效,整个系统或服务就会不可用。常见的单点有:数据库、缓存、Web服务器、负载均衡、网络设备等。2.常见单点故障场景只有一台数据库服务器,宕机后所有业务不可用只有一台Redis缓存,挂掉后缓存全部失效只有一台Web服务器,挂掉后网站无法访问只有一个负载均衡节点,挂掉后流量无法分发只有一条网络链路,断开后所有服务失联3.消除单点故障的主
- php 高并发下日志量巨大,如何高效采集、存储、分析
贵哥的编程之路(热爱分享 为后来者)
PHP语言经典程序100题php开发语言
1.问题背景高并发系统每秒产生大量日志(如访问日志、错误日志、业务日志等)。单机写入、存储、分析能力有限,容易成为瓶颈。需要支持实时采集、分布式存储、快速检索与分析。2.主流架构方案一、分布式日志采集架构[应用服务器(PHP等)]|v[日志采集Agent(如Filebeat、Fluentd、Logstash)]|v[消息队列/缓冲(如Kafka、Redis、RabbitMQ)]|v[日志存储(如E
- 霍夫变换(Hough Transform)算法原来详解和纯C++代码实现以及OpenCV中的使用示例
点云SLAM
算法图形图像处理算法opencv图像处理与计算机视觉算法直线提取检测目标检测霍夫变换算法
霍夫变换(HoughTransform)是一种经典的图像处理与计算机视觉算法,广泛用于检测图像中的几何形状,例如直线、圆、椭圆等。其核心思想是将图像空间中的“点”映射到参数空间中的“曲线”,从而将形状检测问题转化为参数空间中的峰值检测问题。一、霍夫变换基本思想输入:边缘图像(如经过Canny边缘检测)输出:一组满足几何模型的形状(如直线、圆)关键思想:图像空间中的一个点→参数空间中的一个曲线参数空
- 用OpenCV标定相机内参应用示例(C++和Python)
下面是一个完整的使用OpenCV进行相机内参标定(CameraCalibration)的示例,包括C++和Python两个版本,基于棋盘格图案标定。一、目标:相机标定通过拍摄多张带有棋盘格图案的图像,估计相机的内参:相机矩阵(内参)K畸变系数distCoeffs可选外参(R,T)标定精度指标(如重投影误差)二、棋盘格参数设置(根据自己的棋盘格设置):棋盘格角点数:9x6(内角点,9列×6行);每个
- Vue3+Vite+TS+Axios整合详细教程
老马聊技术
VueViteTSvue.js
1.Vite简介Vite是新一代的前端构建工具,在尤雨溪开发Vue3.0的时候诞生。类似于Webpack+Webpack-dev-server。其主要利用浏览器ESM特性导入组织代码,在服务器端按需编译返回,完全跳过了打包这个概念,服务器随起随用。生产中利用Rollup作为打包工具,号称下一代的前端构建工具。vite是一种新型的前端构建工具,能够显著的提升前端开发者的体验。它主要有俩部分组成:一个
- Anaconda 详细下载与安装教程
Anaconda详细下载与安装教程1.简介Anaconda是一个用于科学计算的开源发行版,包含了Python和R的众多常用库。它还包括了conda包管理器,可以方便地安装、更新和管理各种软件包。2.下载Anaconda2.1访问官方网站首先,打开浏览器,访问Anaconda官方网站。2.2选择适合的版本在页面中,你会看到两个主要的下载选项:AnacondaIndividualEdition:适用于
- Linux/Centos7离线安装并配置MySQL 5.7
有事开摆无事百杜同学
LInux/CentOS7linuxmysql运维
Linux/Centos7离线安装并配置MySQL5.7超详细教程一、环境准备1.下载MySQL5.7离线包2.使用rpm工具卸载MariaDB(避免冲突)3.创建系统级别的MySQL专用用户二、安装与配置1.解压并重命名MySQL目录2.创建数据目录和配置文件3.设置目录权限4.初始化MySQL5.配置启动脚本6.配置环境变量三、启动与验证1.启动MySQL服务2.获取初始密码3.登录并修改密码
- python中 @注解 及内置注解 的使用方法总结以及完整示例
慧一居士
Pythonpython
在Python中,装饰器(Decorator)使用@符号实现,是一种修改函数/类行为的语法糖。它本质上是一个高阶函数,接受目标函数作为参数并返回包装后的函数。Python也提供了多个内置装饰器,如@property、@staticmethod、@classmethod等。一、核心概念装饰器本质:@decorator等价于func=decorator(func)执行时机:在函数/类定义时立即执行装饰
- Python中的静态方法和类方法详解
在Python中,`@staticmethod`和`@classmethod`是两种装饰器,它们用于定义类中的方法,但是它们的行为和用途有所不同。###@staticmethod`@staticmethod`装饰器用于定义一个静态方法。静态方法不接收类或实例的引用作为第一个参数,因此它不能访问类的状态或实例的状态。静态方法可以看作是与类关联的普通函数,但它们可以通过类名直接调用。classMath
- Linux操作系统磁盘管理
CZZDg
linux运维服务器
目录一.硬盘介绍1.硬盘的物理结构2.CHS编号3.磁盘存储划分4.开机流程5.要点6.磁盘存储数据的形式二.Linux文件系统1.根文件系统2.虚拟文件系统3.真文件系统4.伪文件系统三.磁盘分区与挂载1.磁盘分区方式2.分区命令3.查看与识别命令4.格式化命令5.挂载命令四.LVM逻辑卷1.概述2.管理命令五.磁盘配额1.概述usrquota:支持对用户的磁盘配额grpquota:支持对组的磁
- Python中类静态方法:@classmethod/@staticmethod详解和实战示例
在Python中,类方法(@classmethod)和静态方法(@staticmethod)是类作用域下的两种特殊方法。它们使用装饰器定义,并且与实例方法(deffunc(self))的行为有所不同。1.三种方法的对比概览方法类型是否访问实例(self)是否访问类(cls)典型用途实例方法✅是❌否访问对象属性类方法@classmethod❌否✅是创建类的替代构造器,访问类变量等静态方法@stati
- Python多版本管理与pip升级全攻略:解决冲突与高效实践
码界奇点
Pythonpythonpip开发语言python3.11源代码管理虚拟现实依赖倒置原则
引言Python作为最流行的编程语言之一,其版本迭代速度与生态碎片化给开发者带来了巨大挑战。据统计,超过60%的Python开发者需要同时维护基于Python3.6+和Python2.7的项目。本文将系统解决以下核心痛点:如何安全地在同一台机器上管理多个Python版本pip依赖冲突的根治方案符合PEP标准的生产环境最佳实践第一部分:Python多版本管理核心方案1.1系统级多版本共存方案Wind
- 基于Python的健身数据分析工具的搭建流程day1
weixin_45677320
python开发语言数据挖掘爬虫
基于Python的健身数据分析工具的搭建流程分数据挖掘、数据存储和数据分析三个步骤。本文主要介绍利用Python实现健身数据分析工具的数据挖掘部分。第一步:加载库加载本文需要的库,如下代码所示。若库未安装,请按照python如何安装各种库(保姆级教程)_python安装库-CSDN博客https://blog.csdn.net/aobulaien001/article/details/133298
- tcpdump交叉编译
weixin_45673259
tcpdump测试工具网络
1.下载路径官网:https://www.tcpdump.org/2.编译解压:tar-xflibpcap-1.10.4.tar.xztar-xftcpdump-4.99.4.tar.xz编译libpcap./configure--host=mips-v720s229-linux--target=mips-v720s229-linuxCC=/opt/A1/mips-gcc720-uclibc229
- 小林渗透入门:burpsuite+proxifier抓取小程序流量
ξ流ぁ星ぷ132
小程序web安全安全性测试网络安全安全
目录前提:代理:proxifier:步骤:bp证书安装bp设置代理端口:proxifier设置规则:proxifier应用规则:结果:前提:在介绍这两个工具具体实现方法之前,有个很重要的技术必须要大概了解才行---代理。代理:个人觉得代理,简而言之,就是在你和服务器中间的一个中间人,来转达信息。那为什么要代理呢,因为这里的burpsuite要抓包,burpsuite只有做为中间代理人才可以进行拦截
- 【Linux内核模块】Linux内核模块程序结构
byte轻骑兵
#嵌入式Linux驱动开发实战linux运维服务器
如果你已经写过第一个"HelloWorld"内核模块,可能会好奇:为什么那个几行代码的程序能被内核识别?那些module_init、MODULE_LICENSE到底是什么意思?今天咱们就来扒一扒内核模块的程序结构,搞清楚一个合格的内核模块到底由哪些部分组成,每个部分又承担着什么角色。目录一、内核模块的"骨架":最简化结构解析二、头文件:内核模块的"说明书"2.1最常用的三个头文件2.2按需添加的其
- jdk tomcat 环境变量配置
Array_06
javajdktomcat
Win7 下如何配置java环境变量
1。准备jdk包,win7系统,tomcat安装包(均上网下载即可)
2。进行对jdk的安装,尽量为默认路径(但要记住啊!!以防以后配置用。。。)
3。分别配置高级环境变量。
电脑-->右击属性-->高级环境变量-->环境变量。
分别配置 :
path
&nbs
- Spring调SDK包报java.lang.NoSuchFieldError错误
bijian1013
javaspring
在工作中调另一个系统的SDK包,出现如下java.lang.NoSuchFieldError错误。
org.springframework.web.util.NestedServletException: Handler processing failed; nested exception is java.l
- LeetCode[位运算] - #136 数组中的单一数
Cwind
java题解位运算LeetCodeAlgorithm
原题链接:#136 Single Number
要求:
给定一个整型数组,其中除了一个元素之外,每个元素都出现两次。找出这个元素
注意:算法的时间复杂度应为O(n),最好不使用额外的内存空间
难度:中等
分析:
题目限定了线性的时间复杂度,同时不使用额外的空间,即要求只遍历数组一遍得出结果。由于异或运算 n XOR n = 0, n XOR 0 = n,故将数组中的每个元素进
- qq登陆界面开发
15700786134
qq
今天我们来开发一个qq登陆界面,首先写一个界面程序,一个界面首先是一个Frame对象,即是一个窗体。然后在这个窗体上放置其他组件。代码如下:
public class First { public void initul(){ jf=ne
- Linux的程序包管理器RPM
被触发
linux
在早期我们使用源代码的方式来安装软件时,都需要先把源程序代码编译成可执行的二进制安装程序,然后进行安装。这就意味着每次安装软件都需要经过预处理-->编译-->汇编-->链接-->生成安装文件--> 安装,这个复杂而艰辛的过程。为简化安装步骤,便于广大用户的安装部署程序,程序提供商就在特定的系统上面编译好相关程序的安装文件并进行打包,提供给大家下载,我们只需要根据自己的
- socket通信遇到EOFException
肆无忌惮_
EOFException
java.io.EOFException
at java.io.ObjectInputStream$PeekInputStream.readFully(ObjectInputStream.java:2281)
at java.io.ObjectInputStream$BlockDataInputStream.readShort(ObjectInputStream.java:
- 基于spring的web项目定时操作
知了ing
javaWeb
废话不多说,直接上代码,很简单 配置一下项目启动就行
1,web.xml
<?xml version="1.0" encoding="UTF-8"?>
<web-app xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance"
xmlns="h
- 树形结构的数据库表Schema设计
矮蛋蛋
schema
原文地址:
http://blog.csdn.net/MONKEY_D_MENG/article/details/6647488
程序设计过程中,我们常常用树形结构来表征某些数据的关联关系,如企业上下级部门、栏目结构、商品分类等等,通常而言,这些树状结构需要借助于数据库完成持久化。然而目前的各种基于关系的数据库,都是以二维表的形式记录存储数据信息,
- maven将jar包和源码一起打包到本地仓库
alleni123
maven
http://stackoverflow.com/questions/4031987/how-to-upload-sources-to-local-maven-repository
<project>
...
<build>
<plugins>
<plugin>
<groupI
- java IO操作 与 File 获取文件或文件夹的大小,可读,等属性!!!
百合不是茶
类 File
File是指文件和目录路径名的抽象表示形式。
1,何为文件:
标准文件(txt doc mp3...)
目录文件(文件夹)
虚拟内存文件
2,File类中有可以创建文件的 createNewFile()方法,在创建新文件的时候需要try{} catch(){}因为可能会抛出异常;也有可以判断文件是否是一个标准文件的方法isFile();这些防抖都
- Spring注入有继承关系的类(2)
bijian1013
javaspring
被注入类的父类有相应的属性,Spring可以直接注入相应的属性,如下所例:1.AClass类
package com.bijian.spring.test4;
public class AClass {
private String a;
private String b;
public String getA() {
retu
- 30岁转型期你能否成为成功人士
bijian1013
成长励志
很多人由于年轻时走了弯路,到了30岁一事无成,这样的例子大有人在。但同样也有一些人,整个职业生涯都发展得很优秀,到了30岁已经成为职场的精英阶层。由于做猎头的原因,我们接触很多30岁左右的经理人,发现他们在职业发展道路上往往有很多致命的问题。在30岁之前,他们的职业生涯表现很优秀,但从30岁到40岁这一段,很多人
- 【Velocity四】Velocity与Java互操作
bit1129
velocity
Velocity出现的目的用于简化基于MVC的web应用开发,用于替代JSP标签技术,那么Velocity如何访问Java代码.本篇继续以Velocity三http://bit1129.iteye.com/blog/2106142中的例子为基础,
POJO
package com.tom.servlets;
public
- 【Hive十一】Hive数据倾斜优化
bit1129
hive
什么是Hive数据倾斜问题
操作:join,group by,count distinct
现象:任务进度长时间维持在99%(或100%),查看任务监控页面,发现只有少量(1个或几个)reduce子任务未完成;查看未完成的子任务,可以看到本地读写数据量积累非常大,通常超过10GB可以认定为发生数据倾斜。
原因:key分布不均匀
倾斜度衡量:平均记录数超过50w且
- 在nginx中集成lua脚本:添加自定义Http头,封IP等
ronin47
nginx lua csrf
Lua是一个可以嵌入到Nginx配置文件中的动态脚本语言,从而可以在Nginx请求处理的任何阶段执行各种Lua代码。刚开始我们只是用Lua 把请求路由到后端服务器,但是它对我们架构的作用超出了我们的预期。下面就讲讲我们所做的工作。 强制搜索引擎只索引mixlr.com
Google把子域名当作完全独立的网站,我们不希望爬虫抓取子域名的页面,降低我们的Page rank。
location /{
- java-3.求子数组的最大和
bylijinnan
java
package beautyOfCoding;
public class MaxSubArraySum {
/**
* 3.求子数组的最大和
题目描述:
输入一个整形数组,数组里有正数也有负数。
数组中连续的一个或多个整数组成一个子数组,每个子数组都有一个和。
求所有子数组的和的最大值。要求时间复杂度为O(n)。
例如输入的数组为1, -2, 3, 10, -4,
- Netty源码学习-FileRegion
bylijinnan
javanetty
今天看org.jboss.netty.example.http.file.HttpStaticFileServerHandler.java
可以直接往channel里面写入一个FileRegion对象,而不需要相应的encoder:
//pipeline(没有诸如“FileRegionEncoder”的handler):
public ChannelPipeline ge
- 使用ZeroClipboard解决跨浏览器复制到剪贴板的问题
cngolon
跨浏览器复制到粘贴板Zero Clipboard
Zero Clipboard的实现原理
Zero Clipboard 利用透明的Flash让其漂浮在复制按钮之上,这样其实点击的不是按钮而是 Flash ,这样将需要的内容传入Flash,再通过Flash的复制功能把传入的内容复制到剪贴板。
Zero Clipboard的安装方法
首先需要下载 Zero Clipboard的压缩包,解压后把文件夹中两个文件:ZeroClipboard.js
- 单例模式
cuishikuan
单例模式
第一种(懒汉,线程不安全):
public class Singleton { 2 private static Singleton instance; 3 pri
- spring+websocket的使用
dalan_123
一、spring配置文件
<?xml version="1.0" encoding="UTF-8"?><beans xmlns="http://www.springframework.org/schema/beans" xmlns:xsi="http://www.w3.or
- 细节问题:ZEROFILL的用法范围。
dcj3sjt126com
mysql
1、zerofill把月份中的一位数字比如1,2,3等加前导0
mysql> CREATE TABLE t1 (year YEAR(4), month INT(2) UNSIGNED ZEROFILL, -> day
- Android开发10——Activity的跳转与传值
dcj3sjt126com
Android开发
Activity跳转与传值,主要是通过Intent类,Intent的作用是激活组件和附带数据。
一、Activity跳转
方法一Intent intent = new Intent(A.this, B.class); startActivity(intent)
方法二Intent intent = new Intent();intent.setCla
- jdbc 得到表结构、主键
eksliang
jdbc 得到表结构、主键
转自博客:http://blog.csdn.net/ocean1010/article/details/7266042
假设有个con DatabaseMetaData dbmd = con.getMetaData(); rs = dbmd.getColumns(con.getCatalog(), schema, tableName, null); rs.getSt
- Android 应用程序开关GPS
gqdy365
android
要在应用程序中操作GPS开关需要权限:
<uses-permission android:name="android.permission.WRITE_SECURE_SETTINGS" />
但在配置文件中添加此权限之后会报错,无法再eclipse里面正常编译,怎么办?
1、方法一:将项目放到Android源码中编译;
2、方法二:网上有人说cl
- Windows上调试MapReduce
zhiquanliu
mapreduce
1.下载hadoop2x-eclipse-plugin https://github.com/winghc/hadoop2x-eclipse-plugin.git 把 hadoop2.6.0-eclipse-plugin.jar 放到eclipse plugin 目录中。 2.下载 hadoop2.6_x64_.zip http://dl.iteye.com/topics/download/d2b
- 如何看待一些知名博客推广软文的行为?
justjavac
博客
本文来自我在知乎上的一个回答:http://www.zhihu.com/question/23431810/answer/24588621
互联网上的两种典型心态:
当初求种像条狗,如今撸完嫌人丑
当初搜贴像条犬,如今读完嫌人软
你为啥感觉不舒服呢?
难道非得要作者把自己的劳动成果免费给你用,你才舒服?
就如同 Google 关闭了 Gooled Reader,那是
- sql优化总结
macroli
sql
为了是自己对sql优化有更好的原则性,在这里做一下总结,个人原则如有不对请多多指教。谢谢!
要知道一个简单的sql语句执行效率,就要有查看方式,一遍更好的进行优化。
一、简单的统计语句执行时间
declare @d datetime ---定义一个datetime的变量set @d=getdate() ---获取查询语句开始前的时间select user_id
- Linux Oracle中常遇到的一些问题及命令总结
超声波
oraclelinux
1.linux更改主机名
(1)#hostname oracledb 临时修改主机名
(2) vi /etc/sysconfig/network 修改hostname
(3) vi /etc/hosts 修改IP对应的主机名
2.linux重启oracle实例及监听的各种方法
(注意操作的顺序应该是先监听,后数据库实例)
&nbs
- hive函数大全及使用示例
superlxw1234
hadoophive函数
具体说明及示例参 见附件文档。
文档目录:
目录
一、关系运算: 4
1. 等值比较: = 4
2. 不等值比较: <> 4
3. 小于比较: < 4
4. 小于等于比较: <= 4
5. 大于比较: > 5
6. 大于等于比较: >= 5
7. 空值判断: IS NULL 5
- Spring 4.2新特性-使用@Order调整配置类加载顺序
wiselyman
spring 4
4.1 @Order
Spring 4.2 利用@Order控制配置类的加载顺序
4.2 演示
两个演示bean
package com.wisely.spring4_2.order;
public class Demo1Service {
}
package com.wisely.spring4_2.order;
public class