【三、对问题进行了解决】MT-Unet模型重新训练腹部和冠脉数据集【个人记录学习】
一、解决【二】中遗留了四个问题。
下面进行解决:
Q1:至于为什么没有val的部分,还没时间研究
Q2:训练和测试的数据痛的同一组,这个容易解决
Q3:用MT-Unet训练我们的数据的尺寸问题:只能用244*244的图像,这个要仔细研究甚至修改网络架构,才能判断是否能够结果。
Q5: 训练出来的.nii.gz偏白色,猜测是像素值的区间出现了问题,应该可以通过归一化到[0,255]
Q1:个人感觉没必要了,test就相当于val了,作用都是来看dice,什么时间收敛,我们可以什么时间停止训练了。(这个问题,如果有其他的想法,欢迎评论区留言,交流学习一下)
Q2:训练和测试的数据痛的同一组。这次在训练腹部数据集时,把30组数据,分为20组训练和10组测试。在训练自己的冠脉数据集时,采用了49组训练集,16组测试集。
Q3:这个目前修改了两个地方,宏观暂时可以跑通,而且效果比用224的好得多。因为个人比较菜,远远达不到可以自主根据论文模型来使用代码一点点复现的地步,因此不确定微观上面是否存在影响。
Q5: 确实是像素值的区间出现了问题。问题的根源主要是在根据项目作者的提示,预处理拿到的Synapse 数据集时将像素利用clip调整到了[-125, 275]范围,并进行了下面的操作。
normalised_image_data = (image_data - args.clip[0]) / (args.clip[1] - args.clip[0])因此,只要反向操作一波就好了。这里需要注意的是,当反向操作的时候,由于精度原因,只能将像素值调整到【0,254.99999523162842】范围,因此,需要在【0,254.99999523162842】的基础上再叠加一次归一化到【0,255】的操作。
二、训练调整好的腹部数据集
1.腹部数据集选取了0066中的30组数据,将其分为了20组训练,10组测试。
这里提一下,前段时间跟作者发邮件,一方面跟作者索要了Synapse处理好并且论文中使用的数据集,昨天发现邮件里,作者给了。另一方面,发现了如果训练ACDC,会出现dice一直固定为一个值,不变化的问题。反馈给作者,作者发现是上传的代码的原因,已经进行了修改,重新提交到GitHub上了。因此,训练ACDC数据集的UU们,可以重新down一下GitHub上的代码【Time:2022/3/28】。
2.训练的时候,batch_size=12,尺寸:224*224,class=14,epoch=200,skip=3剩下都是默认的。
3.最后的训练了89epoch,我看它的dice趋于稳定了,就停止了。
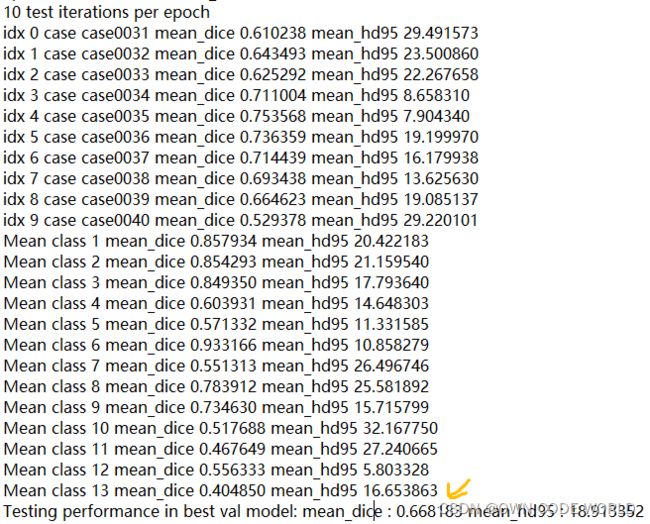
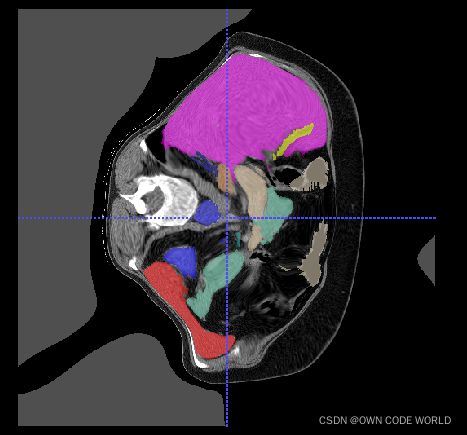
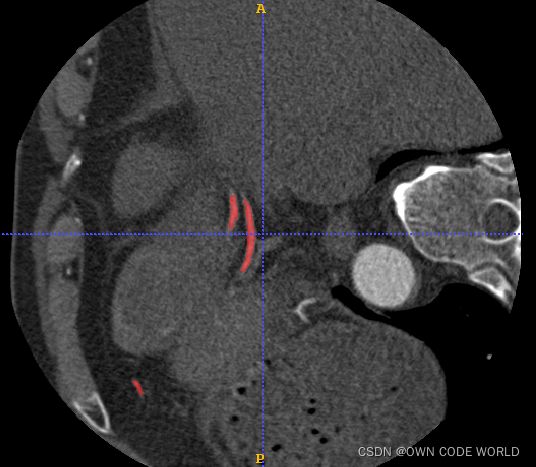
这是最好的一组结果,dice=0.6681.然后,训练出的可视化效果,是下面的样子:
gt pre
4.由于,因此使用的是划分14类的数据(论文中的数据是9类),数据和作者的不一样,因此,效果达不到论文中的程度。
三、训练自己的冠脉数据集-224*224*3
1.这一部分是训练冠脉数据集,在训练之前,由于需要将49组用作train和16组用作test的png数据集,都转化为.nii.gz的格式(其中用作gt的png还需要进行像素值从【0,255】到【0,1】的归一化处理),因此先写了一个处理数据的小脚本(虽然小,但是由于并没有学过python的基础,花费了好多时间,而且第一次处理好了以后,扔到服务器上跑,如果训练集和测试集相同,dice正常,如果不同,那么不同的dice一直为0或者0.000xx级别的。由于当训练集和测试集相同时,dice正常,所以认为数据是正常的,结果在经过两天各种改错尝试,小emo后,发现还是数据集的问题,写的小脚本,需要修改两行的内容(;´༎ຶД༎ຶ`) 哭死,两行==两天)。
2.将png都转化为.nii.gz的格式后,还需要按作者的要求,对数据进行预处理的操作。
3.将49组数据作为训练集,16组数据作为测试集。batch_size=12, 224*224,class=2,epoch=200,skip=3,其他默认。
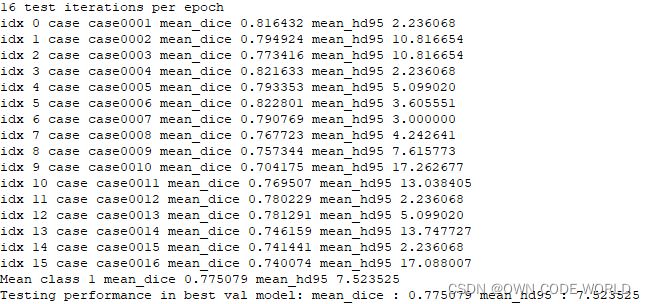
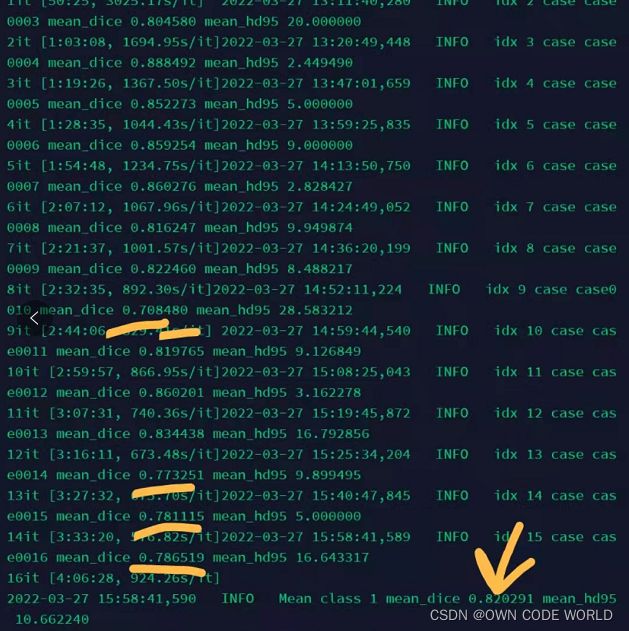
4.最后训练了143epoch,已经开始过拟合了,就停止了。
其中最好的在69epoch,dice=0.775079。不是很高,达不到想要的高度,但出现这个原因也正常。因为训练时的数据时224*224*3的数据集,回丢失很多的信息。512*512*3才是正确的道路,因此后面需要尝试尽心修改。
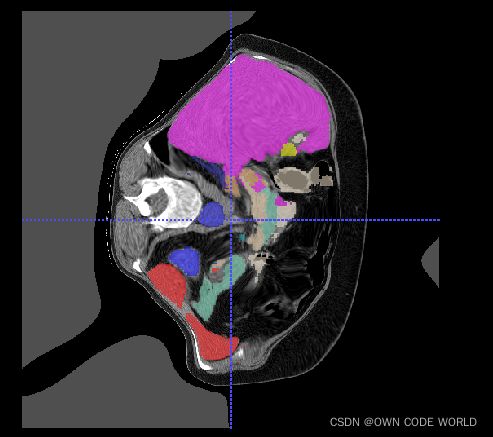
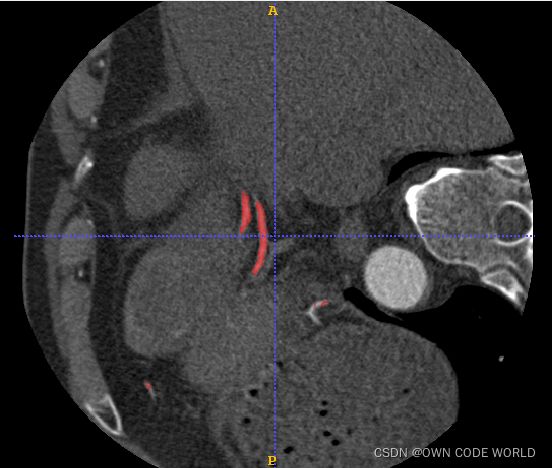
训练出的可视化效果,是下面的样子:
gt pre
四、训练自己的冠脉数据集-512*512*3
这一部分,目前还在跑,后面跑完以后,再继续来写。结果是比用224*224的好些,至少目前最好的dice已经到了0.82.
五、小啰嗦
虽然MT-unet还在跑,但是反思了很多,从EfficientUnet++,到Unet,再到MT-unet,均动手去配置环境,调试代码,然后,找数据,去跑复现作者的结果,但结果一直达不到作者的程度,用自己的数据集也到不了期望的水准。确实烦了,这种碰运气,想碰对一个模型,急功近利,自己的数据就能跑的很不错的想法,现在感觉不可能,甚至有些搞笑了。差不多行了,该去入门“黑盒子”了。一直在黑盒子外,逛荡,胡乱调参不会有什么收获的。那就从Unet开始吧,沉下去一段时间,好好摸索摸索。一个小目标:先能设计一个 能跑 的模型,然后代码实现出来。