生物基因结构
基因结构
最近需要对启动子区域进行预测,所以首先对启动子的结构特征进行了解,而说到启动子,那就一定要了解基因结构,所以,在网上查找了部分资料进行整理与学习。
首先,根据RNA合成的不同时期,从DNA到成熟mRNA,分为三个阶段了解基因结构的变化。
RNA合成
特点
- RNA 的合成是以反义链(模板链)为模板,以 5’→3’方向合成的,合成的 RNA 的序列是与 DNA 编码链(有意链)相同。
- 在合成的RNA中是以磷酸二酯键来连接碱基与嘌呤的。
- 在合成RNA的时候,需要RNA聚合酶RNA polymerase , 4种核糖核苷酸rNTPs, 转录因子 transcription factors,启动子 promoter & 终止子 terminator/模版 template
RNA聚合酶-RNA polymerase:
细菌 Bacteria:全酶 (Holoenzyme) 由一种核心酶(α2ββ’σω)和多种因子组成。
真核生物 Eukaryotes:三种 RNA 聚合酶 ,根据对
α-鹅膏覃碱分为三类。
酶 细胞内定位 转录产物 相对活性 对α-鹅膏覃碱的敏感程度 RNA 聚合酶Ⅰ 核仁 rRNA(28S, 18S, 5.8S) 50-70% 不敏感 RNA 聚合酶Ⅱ 核质 hnRNA*, snRNA, mRNA 20-40% 敏感 RNA 聚合酶Ⅲ 核质 tRNA, 5SRNA, 某些涉及 RNA 加工的 snRNA 约 10% 存在物种特异性 PS:细菌中研究得最为清楚的是大肠杆菌的RNA聚合酶,该酶是由五种亚基组成的六聚体(α2ββ’ωσ),该六聚体称之为核心酶(coreenzyme),σ因子与核心酶结合后称为全酶 (Holoenzyme)。
对RNA的分类
其中只有真核生物需要转化成前mRNA,而细菌与原核生物由于缺少内含子不需要这一步。
-
hnRNA: heterogeneous nuclear RNA, 核内不均一 RNA, RNA 的前体
-
snRNA:核小RNA是真核生物转录后加工过程中RNA剪接体(spliceosome)的主要成分,参与mRNA前体的加工过程。
-
snoRNA:核仁小RNA(small nucleolar RNA)由内含子编码,分布于真核生物细胞核仁的小分子非编码RNA,具有保守的结构元件。已证明有多种功能,主要参与rRNA的加工;反义snoRNA指导rRNA核糖甲基化。
-
scRNA:,胞质小RNA(small cytoplasmic RNA,scRNA),细胞质中的小分子RNA。通常指转移核糖核酸(tRNA)和小的核糖体RNA(rRNA),如5S rRNA、5.8S rRNA等。
-
tmRNA:转运-信使RNA(Transfer-messenger RNA),是一种细菌的RNA分子,是tRNA和信使RNA类似物。 tmRNA的用途十分广泛,它可用于回收停滞的核糖体,并有利于异常的信使RNA的降解。
DNA
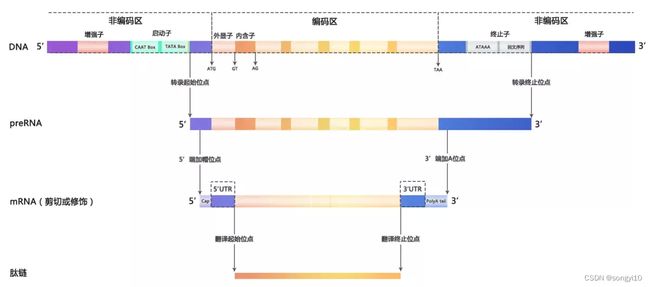
转录是从DNA聚合酶结合到模版链上开始的,用一个简单模型来概括就是将DNA分为两个部分,编码区与非编码区。下图是包含了一个最简单的转录单元(transcription unit),转录单元起始于启动子并终止于终止子。
PS:一个转录单元只包含一个基因,而转录本是由多个转录单元加上基因间隔区组成的。
由上图可以看出,基因结构分为编码区与非编码区,真核生物的编码区存在内含子与外显子,首先会生成前mRNA,然后将mRNA中的内含子切除,最后合并外显子形成mRNA。而原核生物没有内含子,可以直接生成mRNA。
编码区
外显子 Exon:外显子是在 preRNA 经过剪切或修饰后,被保留的DNA部分,并最终出现在成熟RNA的基因序列中。
内含子 Intron:在真核生物中,内含子作为阻断基因的线性表达的一段DNA序列,是在 preRNA 经过剪切或修饰后,被切除的DNA序列
非编码区
非编码区虽然不会被转录,但是对与基因的表达起到了重要的作用,启动子,终止子,增强子等都处于非编码区中,且非编码区在总RNA中占比超过90%。非编码区RNA可以转录为功能性RNA,如tRNA,rRNA等;也可以对转录起到控制与调控作用,甚至参与mRNA的加工。
启动子:是一段位于结构基因 5’端上游区的保守的 DNA 序列,能活化 RNA 聚合酶,使之与模板 DNA 准确地相结合并具有转录起始的特异性。启动子长约100-1000bp。在转录过程中,RNA聚合酶与转录因子可以识别并特异性结合到启动子特有的DNA序列(一般为保守序列),从而启动转录。启动子本身并不转录而且也不控制基因活动,而是通过转录因子结合来调控转录过程。在细胞核中,似乎启动子优先分布在染色体区域的边缘,可能是在不同染色体上共同表达基因。 此外,在人类中,启动子显示出每个染色体特有的某些结构特征。
原核生物启动子
原核生物的启动子最重要的是-10区与-35区,如果在原核生物中这两个区域之间的距离超过或小于16-19bp,都会降低转录活性,可能与RNA Pol本身构象有关。
-10区(-10 box,Pribnow 盒)
是由 5 个核苷酸组成的保守序列,是聚合酶结合位点,其中央大约位于起点上游 10bp 处,所以又称为 -10 区,是真核生物与古细菌的TATA 盒的原核同源物,具有较短共有序列TATAATAAT。
-10区特点:
- AT 较丰富,易于解链;
- 其保守序列为
TAtAaT,位于-10bp 左右,保守序列小写字母表示该碱基保守性略低; - 突变后会改变启动子效率;
- 与 RNA pol 紧密结合形成开放启动复合体;
- 使 RNA pol 定向转录。
研究发现,只有
-10 区是不能结合 RNA 聚合酶的。从噬菌体的左、右启动子 PL 及 PR 和 SV40 启动子的 - 35 bp 附近找到了另一段共同序列:TTGACA
-35区(35 box ( Sextama 盒 ))
其保守序列为 TTGACa, 与 -10 序列相隔 16-19bp。
为 RNA pol 的识别位点。
是 RNA 聚合酶与启动子的结合位点,能与 σ 因子相互识别而具有很高的亲和力。但不能被 RNA Pol 的核心酶识别,核心酶只能起到和模板结合和催化的功能。
- 原核生物启动子的共同特点
- 位置和距离都比较恒定,都在其控制基因的 5’端,常和操纵子相邻;
- -35 序列,-10 序列等特征序列都十分保守;
- 都含有识别 (R ) 、结合 (B) 和起始 (I) 三个位点;
- 直接和多聚酶相结合,与
σ 结合决定转录的特异性。
σ因子自身并不能与 DNA 结合,但与核心酶相互作用后暴露出σ因子的 DNA 结合域:β’ 亚基的氨基酸片段促进 σ因子与启动子 -10 框的非模板链的结合。
σ因子可以选择哪些基因将被转录:
- σ70 (RpoD)-“管家”σ因子/主要σ因子,转录生长细胞中的大多数基因。制造保持细胞存活所必需的蛋白质。
- σ54 (RpoN) -氮源缺陷应激σ因子
- σ38 (RpoS) -饥饿应激σ因子
- σ32 (RpoH) 热休克应激σ因子
- σ28 (RpoF) -鞭毛σ因子
- σ24 (RpoE) -极端/极端应激σ因子
- σ19 (FecI) -柠檬酸铁σ因子,调节用于铁运输的 fec 基因的转录
真核生物启动子
真核生物 RNA 聚合酶Ⅱ所识别的启动子区
TATA box(Hogness 区)
-25 ~ -30 bp 区,保守序列为 TATAAA。确定转录起始位点,使转录精确地起始:如果除去 TATA 区或进行碱基突变,转录产物下降的相对值不如 CAAT 区或 GC 区突变后明显,但发现所获得的 RNA 产物起始点不固定。
- 启始子 (initiator,
Inr):转录起始位点附近。 - 上游启动子元件 ( upstream promoter element,
UPE, 又称 上游激活序列 (upstream activating sequence,UAS) : TATA 区上游的保守序列。 CAAT box
CCAAT box(有时也缩写为CAAT box或CAT box):具有GGCCAATCT 共有序列的不同核苷酸序列 ,是真核生物基因常有的调节区,位于转录起始点上游约-80bp处,可能也是RNA聚合酶的一个结合处,控制着转录起始的频率。与之对应的就是原核的-35区。
CAAT框是最早被人们描述的常见启动子元件之一,常位于接近-80的位置,但是它可以在离起始点较远的距离仍能起作用,且在两种取向均可发挥作用。CAAT框的突变敏感性提示了它在决定转录效率上有很强的作用,但是突变对启动子的特异性没有影响。
GC box:-80 ~ -110 含有 GCCACACCC 或 GGGCGGG 序列。
CAAT 区和 GC 区主要控制转录起始频率,基本不参与起始位点的确定。
- 真核生物启动子特点
- 有多种元件:TATA 框,GC 框,CATT 框等;
- 结构不恒定。有的有多种框盒,如组蛋白 H2B; 有的只有TATA 框和 GC 框,如 SV40 早期转录蛋白;
- 它们的位置、序列、距离和方向都不完全相同;
- 有的有远距离的调控元件存在,如增强子,这些元件常常起到控制转录效率和选择起始位点的作用,不直接和 RNA pol 结合。转录时先和其它转录激活因子相结合,再和聚合酶结合。
增强子 Enhancer
增强子是位于转录起始位点或下游基因1Mbp的位置,长度50-1500bp的序列,其可以被转录激活因子结合从而增加特定基因转录发生的可能性,广泛的存在于原核与真核生物基因结构中。
增强子能大大增强启动子的活性。增强子有别于启动子处有两点:
- 增强子的位置相对于启动子而言不是固定的,而能有很大的变动;它能在两个方向产生相互作用。
- 一个增强子并不限于促进某一特殊启动子的转录,它能刺激在它附近的任一启动子。
终止子 Terminator
终止子与终止密码子的概念区分:二者在名称上相似,但是含义是截然不同的。终止子是处于基因的非编码区的一段DNA序列,用于终止转录。而终止密码子是在翻译过程中终止肽链合成的mRNA中的三联体碱基序列,一般情况下为UAA,UAG和UGA,不编码为氨基酸。
终止子处于基因或操纵子的末端,给RNA聚合酶提供转录终止信号的DNA序列。
- ATAAA
ATAAA 是 preRNA 在通过修剪后形成成熟mRNA 时在3’UTR产生ployA 是的加尾信号。但是这段序列并不是绝对保守,也可能为其他A富集的序列,比如AATAAA等。
- 回文序列 palindrome sequence
回文序列是双链DNA中的一段倒置重复序列,这段序列有个特点,它的碱基序列与其互补链之间正读和反读都相同。当该序列的双链被打开后,如果这段序列较短,有可能是限制性内切酶的识别序列,如果比较长,有可能形成发卡结构,这种结构的形成有助于DNA与特异性DNA与蛋白质的结合。
preRNA
- 转录起始位点 Transcription start sites (TSS)
转录起始位点是指与新生RNA链第一个核苷酸相对应的DNA链上的碱基,通常为一个嘌呤(A 或G),即5’UTR的上游第一个碱基。 通常在起始核苷酸的两侧为 C 和 T (i.e. CGT or CAT)。
- 转录终止位点 Transcription termination sites (TTS)
转录起始位点是指新生RNA链最后一个核苷酸相对应的DNA链上的碱基。当RNA链延伸到转录终止位点时,RNA聚合酶不再形成新的磷酸二酯键,RNA-DNA杂合物分离,转录泡瓦解,DNA恢复成双链状态,而RNA聚合酶和RNA链都被从模板上释放出来。
- 开放阅读框 Open reading frame(ORF)
ORF 是连续的一段密码子,其含有起始密码子(通常是AUG)和终止密码子(通常是UAA,UAG或UGA)。在真核基因中,ORF跨越内含子/外显子区域,其可以在 ORF 转录后拼接在一起以产生蛋白质翻译的最终mRNA。 由于读写位置不同(对应不同的起始位点),ORF 可能翻译为不同的多肽链。
mRNA
从上图可以看出,外显子不仅仅只有编码区域,还有非编码的区域
5'UTR与3'UTR。
UTR (Untranslated Region ),如果这段序列位于5’端,就称作5’UTR(5‘-untranslated region),也叫前导序列(leader)。相反若位于3’端,我们就叫它3’UTR(3‘-untranslated region),也叫尾随序列(trailer)。
5’UTR 位于从mRNA起点的甲基化鸟嘌呤核苷酸帽延伸至起始密码子AUG,3’UTR从编码区末端的终止密码子延伸至多聚A尾巴(Poly-A)的前端 。
原核生物和真核生物都可以看到UTR,但它们的长度和组成都有所不同。原核生物中,5′非翻译区通常为3至10个核苷酸的长度。但在真核生物中,5′非翻译区有成百上千个核苷酸的长度。与原核生物相比,真核生物的基因组的复杂性更高,3′非翻译区的长度也不同。虽然5′非翻译区和3′非翻译区在长度上有差异,但5′非翻译区的长度在演化过程中比3′非翻译区显得更保守。
5‘Cap
5‘Cap也被称为7-甲基鸟苷酸帽,缩写为m7G。这种结构在RNA进出细胞核起到识别作用;可以抗5’-核酸外切酶的截切;促进5’端内含子的切除;在翻译过程中有助于核糖体对mRNA的识别和结合。
3’ PolyA tail
Poly A tail 由多个腺苷一磷酸组成 ,也就是说它是一段仅含有腺嘌呤碱基的RNA 。这种结构可以避免细胞质中的酶促降解,并有助于转录终止,mRNA从细胞核中的输出和翻译。
CDS (coding dna sequence)
CDS 是基因中DNA或RNA为蛋白质编码区域,该区域通常开始于5‘末端的起始密码子并结束于3’端的终止密码子。生物体基因组编码区的总和称为外显子组。
CDS与ORF的区别与联系:
- CDS是Coding sequence的缩写,是指编码一段蛋白产物的序列,是与蛋白质密码子一一对应的序列。
- ORF是open reading frame的缩写,翻译成开放阅读框,是指从一个起始密码子开始到一个终止密码子结束的一段序列,但并不是所有读码框都能表达出蛋白产物(在我看来就是可能会包含内含子,读码框本省无法翻译为蛋白质,但是经过剪切后就可以)
- CDS必定是一个ORF,但也可能包括多个ORF,相反,每个ORF不一定都是CDS。(真核与原核)
参考资料
基因结构:https://zhuanlan.zhihu.com/p/49601643
转录:https://blog.csdn.net/zea408497299/article/details/124464842?ops_request_misc=%257B%2522request%255Fid%2522%253A%2522166081277516781432993626%2522%252C%2522scm%2522%253A%252220140713.130102334.pc%255Fall.%2522%257D&request_id=166081277516781432993626&biz_id=0&utm_medium=distribute.pc_search_result.none-task-blog-2allfirst_rank_ecpm_v1~rank_v33_ecpm-3-124464842-null-null.142




