系统问题解决
dcm图片到nrrd的格式转换(二维到三维)
dcm:DICOM(Digital Imaging and Communications in Medicine)即医学数字成像和通信,是医学图像和相关信息的国际标准(ISO 12052)。它定义了质量能满足临床需要的可用于数据交换的医学图像格式。DICOM被广泛应用于放射医疗,心血管成像以及放射诊疗诊断设备(X射线,CT,核磁共振,超声等),并且在眼科和牙科等其它医学领域得到越来越深入广泛的应用。在数以万计的在用医学成像设备中,DICOM是部署最为广泛的医疗信息标准之一。当
前大约有百亿级符合DICOM标准的医学图像用于临床使用。
nrrd:具体信息有: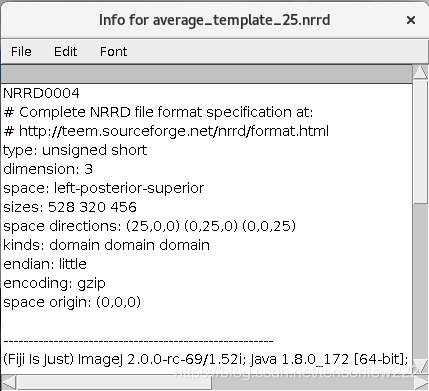
Line 1: NRRD0004
表示NRRD格式的版本,不同的版本在保存信息上存在差异。
Line 2 and 3 # 开头,代表注释
标注NRRD标准格式的网站,对某些细节感兴趣的读者,可以自己去探索。
Line 4: type: unsigned short
表示文件存储数据的数字范围,这里表示图像的像素值范围在0-255。
Line 5: dimension: 3
表示图像是三维的
Line 6: space: left-posterior-superior
表示物理空间的坐标系
Line 7: sizes: 528 320 456
表示图像在每个坐标中的像素个数。此时可以参照图1的大小和左上角的显示信息。
这里需要注意的是,与Matlab中保存三维矩阵不同,这里的528代表的是图像长度(从左到右),对应Matlab三维矩阵的列方向(column);320代表的是图像的宽度(从上到下),对应Matlab三维矩阵的行方向(row)。
如果,我们利用Matlab三维矩阵来保存这幅图像,那么我们需要的三维矩阵应该初始化为 data=zeros([320, 528, 456]);
Line 8: space directions: (25,0,0) (0,25,0) (0,0,25)
代表了图像中的坐标空间与物理空间的对应关系。
dcm到nrrd的处理过程:多张dcm图片形成一个dcm序列。调用sitk库的writeImage成为nrrd文件
dcms_name = sitk.ImageSeriesReader.GetGDCMSeriesFileNames(tidy_ct_path)
dcms_read = sitk.ImageSeriesReader() # 类似java里面的实例
dcms_read.SetFileNames(dcms_name) # 将list里面的元素设置
dcms_series = dcms_read.Execute()
# Execute()源码介绍execute(ImageSeriesReader self) -> Image
if not os.path.exists(sf):
print('sf目录不存在,请检查一下')
exit()
sitk.WriteImage(dcms_series, sf + '\\%d.nrrd' % (nb + 1))
sitk库:
ITK是一个功能很强大的医学图像处理公开库,搭配VTK用以显示图像,可以实现几乎所有医学图像处理的功能需要。ITK通常以C++包进行提供,当然也可以自己编译为Python包,不过编译过程比较繁琐耗时,而且很容易踩坑。但ITK官方进行的Python封装SimpleITK,则直接可以拿来使用;虽然有部分ITK的功能没有包含,但已基本够用了。我们在处理医学图像时,使用的基本都是SimpleITK。
simpleitk打开dicom文件
1.读取文件:读取DICOM序列
医学图像中一个CT序列包含很多张图片,即一个case包含许多slice,使用SimpleITK可以直接读取一个序列,并方便地得到各种参数,将图像数据转换成numpy Array:
import SimpleITK as sitk
import numpy as np
reader = sitk.ImageSeriesReader()
dicom_names = reader.GetGDCMSeriesFileNames(case_path)
reader.SetFileNames(dicom_names)
image = reader.Execute()
image_array = sitk.GetArrayFromImage(image) # z, y, x
origin = image.GetOrigin() # x, y, z
spacing = image.GetSpacing() # x, y, z
需要注意的是,SimpleITK读取的图像数据的坐标顺序为zyx,即从多少张切片到单张切片的宽和高;而据SimpleITK Image获取的origin和spacing的坐标顺序则是xyz。这些需要特别注意。
读取DICOM单张图片
可以将一个DICOM序列作为一个整体一次读入,也可以一张一张地读入每张切片:
import SimpleITK as sitk
import numpy as np
image = sitk.ReadImage(slice_path)
image_array = sitk.GetArrayFromImage(image) # z, y, x
这里需要注意的除了坐标顺序是zyx之外,还需注意,即使读取单张切片,所得到的结果也是3维的,只不过第一维是1。
读取mhd文件
涉及DICOM序列时,为了传输方便(从上百个dcm文件到一个mhd文件),很多情况下以mhd文件格式进行呈现,不过mhd文件只是一个很小的包含数据信息的文件,同时搭配的通常还有一个二进制的数据文件(格式为raw或zraw等)。使用SimpleITK读取这种文件也比较方便。
import SimpleITK as sitk
import numpy as np
image = sitk.ReadImage(mhd_path)
image_array = sitk.GetArrayFromImage(image) # z, y, x
origin = image.GetOrigin() # x, y, z
…
有时候不想整个读取数据(因为比较大,读取处理比较慢),想要读取的只是一些基本信息,比如origin、spacing等等。这时可以只读取mhd文件,据此获取信息
2.处理文件
处理DICOM文件主要有插值等操作,可以直接使用SimpleITK(或者说是ITK)的相关函数,并通过pipeline结构进行处理。
插值
import SimpleITK as sitk
reader = sitk.ImageSeriesReader()
dicom_names = reader.GetGDCMSeriesFileNames(case_path) reader.SetFileNames(dicom_names)
image = reader.Execute()
resample = sitk.ResampleImageFilter()
resample.SetOutputDirection(image.GetDirection())
resample.SetOutputOrigin(image.GetOrigin())
newspacing = [1, 1, 1]
resample.SetOutputSpacing(newspacing)
newimage = resample.Execute(image)
三维重建的原理
CT三维重建主要有六种基本后处理方法:
1.多层面重建(MPR)
多层面重建是最基本的“三维”重建成像方法,是二维的图像序列,和我们最熟悉的轴位图像是一个“家族”的。
MPR适用于任一平面的结构成像,以任意角度观察正常组织器官或病变,可以显示腔性结构的横截面以观察腔隙的狭窄程度、评价血管受侵情况、真实地反映器官间的位置关系等。
2.最大密度投影(MIP):
最大密度投影是将一定厚度(即CT层厚)中最大CT值的体素投影到背景平面上,以显示所有或部分的强化密度高的血管和/或器官,简单原理和图像是酱紫的:
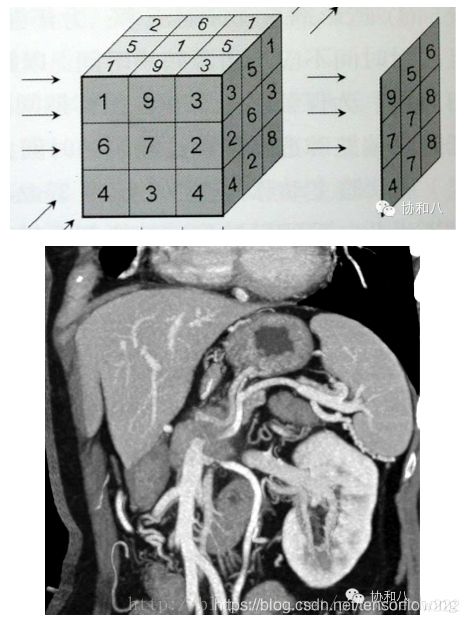
由于这种方法显示的是一定层厚图像中CT值最高的体素,所以变化层厚会对图像产生影响:
3.表面阴影遮盖(SSD)
将操作者的眼睛作为假设光源方向,投射到CT值在设定阈值以上的体素上则不再透过继续成像,仅呈现所有表面体素的集合立体图形,适用于显示CT值与其他结构相差较大的组织结构成像……
SSD图像就像是黑白的塑形图像,所以临床上主要用于显示骨骼病变或是结肠CT重建
容积漫游技术(VRT)
这种三维成像功能非常强大,形态及色彩逼真,绝对是CT三维重建中的“高富帅”,可以对动静脉血管、软组织及骨结构等进行立体塑形成像,也可以显示支气管树、结肠及内耳等结构,对于复杂结构的成像有一定优势,VRT图像直观生动,深受广大医生的喜爱,称得上是辅助诊断、显示病变的大杀器,但是我们要注意一点,VRT图像的伪彩设置很重要,不恰当的伪彩设置会将血管外层像素过滤掉,显示的血管狭窄的程度会比真实情况严重。
曲面重建(CPR)
这种重建技术是在一个维度上选择特定的曲线路径,将该路径上的所有体素在同一平面上进行显示,可以一次评价曲度较大的结构如脾动脉、胰管、冠状动脉等管状结构的全长情况,
虚拟内镜技术(VE)
这种CT重建图像可以模拟各种内镜检查的效果,它是假设视线位于所要观察的管“腔”内,通过设定一系列的参数范围,即可看到管“腔”内的结构
总结:所以CT扫描的时候直接能够扫描到厚度,因为其扫描的最小单位是体素,所以其实我们获取多张二维dcm格式的图像时已经有了三维的信息
用VTK实现CT图片的三维重建过程
1.读取数据:
首先,读取切片数据,并将其转换为我们的开发工具VTK所支持的一种数据表达形式。我们给CT数据建立的是比较抽象的等值面模型,最后将物理组件与抽象的模型结合在一起来建立对CT 数据的可视化,以帮助用户正确理解数据。利用VTK中的vtkDICOMImageReader 我们可以很方便的读取切片数据,读取数据的代码如下所示:
reader = vtkDICOMImageReader::New();//建立一个读取对象
reader->SetDataByteOrderToLittleEndian();
reader->SetDirectoryName(m_path); //设置读取切片数据文件的路径
shrink=vtkImageShrink3D::New();//抽取样点,显示数量减少速达加快
shrink->SetShrinkFactors(4,4,1);
shrink->AveragingOn();
shrink->SetInput((vtkDataObject *)reader->GetOutput());
2.提取等值面
接着我们就可以用算法对所读取的数据进行处理了。本人采用的经典MC的面绘制方法,首先利用vtkMarchingCubes 类来提取出某一CT 值的等值面,但这时的等值面其实仍只是一些三角面片,还必须由vtkStripper 类将其拼接起来形成连续的等值面。这样就把读取的原始数据经过处理转换为应用数据,也即由原始的点阵数据转换为多边形数据然后由vtkPolyDataMapper 将其映射为几何数据,并将其属性赋给窗口中代表它的演员,将结果显示出来。
vtkMarchingCubes *skinExtractor = vtkMarchingCubes::New();
//建立一个Marching Cubes 算法的对象,从CT切片数据中提取出皮肤
skinExtractor->SetValue(0,300); //提取出CT 值为300
skinExtractor->SetInputConnection(shrink->GetOutputPort());
vtkDecimatePro *deci=vtkDecimatePro::New(); //减少数据读取点,以牺牲数据量加速交互
deci->SetTargetReduction(0.3);
deci->SetInputConnection(skinExtractor->GetOutputPort());
vtkSmoothPolyDataFilter *smooth=vtkSmoothPolyDataFilter::New(); //使图像更加光滑
smooth->SetInputConnection(deci->GetOutputPort());
smooth->SetNumberOfIterations(200) ;
vtkPolyDataNormals * skinNormals = vtkPolyDataNormals::New(); //求法线
skinNormals->SetInputConnection(smooth->GetOutputPort());
skinNormals->SetFeatureAngle(60.0);
vtkStripper *stripper=vtkStripper::New(); //将三角形连接起来
stripper->SetInputConnection(skinNormals->GetOutputPort());
vtkPolyDataMapper *skinMapper = vtkPolyDataMapper::New(); //将几何数据映射成图像数据
skinMapper->SetInput(stripper->GetOutput());
skinMapper->ScalarVisibilityOff();
利用同样的方法,我们也可以提取出骨骼的等值面。骨骼的CT 值是1150 左右。所以只要在SetValue()方法中将参数设置为1150 就可以了。而且Visualization Toolkit支持多表面重建,所以在实际应用中我们可以设置多个参数值,提取出多个等值面并同时显示出来。在这个应用实例中我们只对血管等值面进行了重建。
3.显示及结果
通过前面这些工作,我们基本上已经完成了对数据的读取处理映射等步骤,下面我们就要对数据进行显示了。
//设置照相机
aCamera = vtkCamera::New();
aCamera->SetViewUp (0, 0, -1);
aCamera->SetPosition (0, 1, 0);
aCamera->SetFocalPoint (0, 0, 0);
aCamera->ComputeViewPlaneNormal();
//设置Actor相关系数
coneActor = vtkActor::New();
coneActor->SetMapper(skinMapper);
coneActor->GetProperty()->SetAmbient(0.5);
coneActor->GetProperty()->SetDiffuse(1);
coneActor->GetProperty()->SetSpecular(0.6);
//显示类
renderer = vtkRenderer::New();
renderer->AddActor(coneActor);//添加coneActor对象
renderer->AddActor2D(textActor);//添加textActor对象
renderer->SetBackground(0,0,0);
renderer->SetActiveCamera(aCamera);//添加照相机
renderer->ResetCamera ();
renWin = vtkRenderWindow::New();//设置绘图窗口renWin->AddRenderer(renderer);//装载绘图类
iren = vtkWin32RenderWindowInteractor::New();//设置绘图窗口交互
iren->SetRenderWindow(renWin);//装载绘图窗口
如何从一序列dcm格式获取三维信息
一种基于面片模型的X射线图像模拟方法,包含以下步骤:
从CT图像中分割获取各器官的三角面片模型;
对各器官的模型的正向面深度图和背向面深度图进行顺序无关的融混操作,获得器官模型各投影位置处的厚度值;
对每一器官模型使用朗伯-比尔定律模拟X射线的能量衰减;
对多个器官的X射线图像进行深度融合;
对融合后的X射线图像加入噪声。
所述从CT图像中分割获取各器官的三角面片模型包含以下步骤:
针对不同器官的特点,使用不同的图像分割算法进行器官模型的三维重建;
器官模型使用三角网格进行存储。
所述对器官模型的正向面深度图和背向面深度图进行顺序无关的融混操作包含以下步骤:
以射线源所在位置为相机位置,光线发射方向为观察方向进行相机参数设置;
将世界视点投影矩阵传入顶点着色程序中,并在其中计算每个点的投影坐标,投影坐标的Z值即为深度值;
在片段着色程序中将深度值进行归一化,再将深度值赋给颜色值,剔除深度值较大的片段,获得场景的正向面深度图;
剔除深度值较小的片段,获得场景的背向面深度图;
将两张深度图上的数据进行顺序无关的融混操作,得到的值就是器官模型对应投影位置处的厚度值。
所述对每一器官模型使用朗伯-比尔定律模拟X射线的能量衰减包含以下步骤:
针对每一器官模型的每一投影位置计算对应的厚度值;
设置每一器官对X射线的吸收系数;
使用朗伯-比尔定律模拟X射线的能量衰减。
更详细的内容见:http://www.xjishu.com/zhuanli/55/201910068600.html