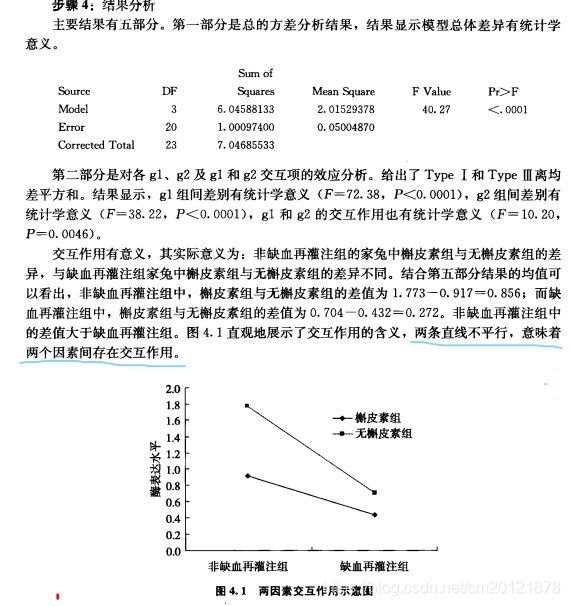
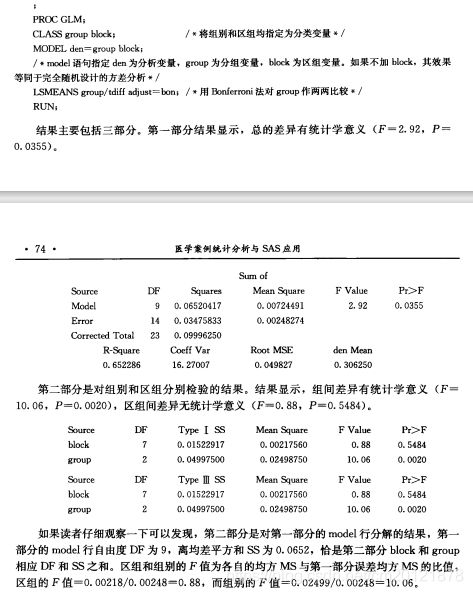
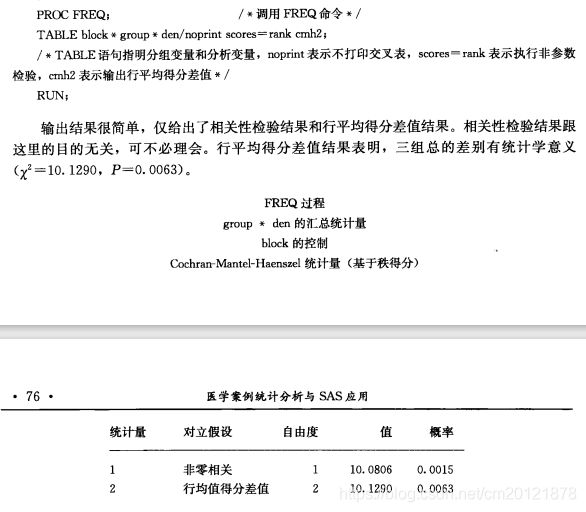
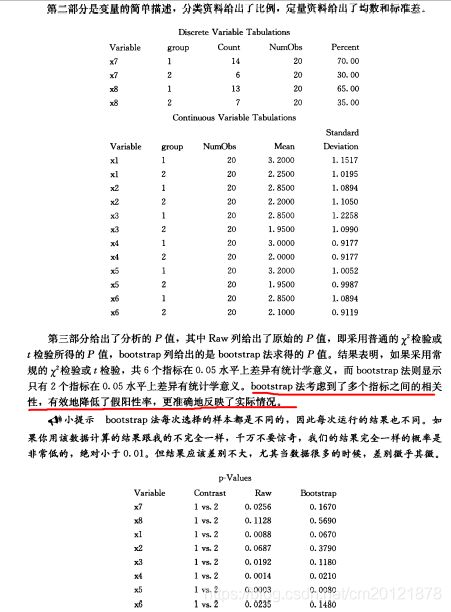
医学案例统计分析与SAS应用--自学笔记
目录
- 第二章医学研究设计与SAS实现
-
- 科研设计思路
-
- 样本含量估计
- 实验设计
- 科研设计的sas实现
-
- 完全随机设计
- 随机区组设计
- 析因设计
- 关系型研究
- 第三章 统计描述与SAS分析
-
- 统计描述及sas命令简介
-
- 定量资料的统计描述
- 分类资料的统计描述
- 第四章 定量资料的SAS统计分析
-
- 定量资料常用统计方法
-
- t检验
- 方差分析
- 秩和检验
- 多重检验
- 正态性检验
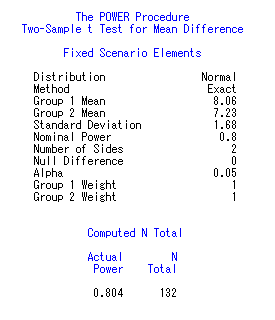
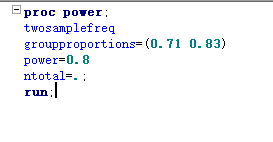
- 把握度的计算
- 定量资料的分析思路
-
- 析因设计资料
- 配对设计资料
- 随机区组资料
- 多指标的组间比较
- 第五章 分类资料的SAS统计分析
-
- 分类资料常用统计方法
- 分类资料的分析思路
-
- 四格表资料
- R*2表资料
- 趋势性检验
- 两两比较
- 2*C表无序资料
- 2*C表有序资料
- 配对资料
- 多层分类资料
- 第六章相关分析及SAS实现
-
- 相关分析
-
- 定量资料相关分析
- 分类资料相关分析
- 相关分析的思路
-
- 线性相关分析
-
- 偏相关分析
- 分类资料的相关性
- 配对资料的相关性
- 多分类指标的相关性
- 第七章 线性回归与SAS分析
-
- 线性回归简介
- 线性回归的分析思路
-
- 简单线性回归分析
- 多重线性回归分析
-
- 应用条件检验
- 单因素分析
- 多因素分析
- 线性回归的替代方法
-
- 主成分回归
-
- 共线性诊断
- 主成分分析
- 主成分回归
- 偏最小二乘回归
-
- VIP指标
- 稳健回归
- 非参数回归
- 第八章logistics回归与SAS分析
-
- logistics回归
- logistics的分析思路
-
- 单因素
- 多因素
- logistics回归的扩展
-
- 多项logistics回归
- 有序logistic回归
- 第九章 生存分析与SAS分析
-
- 生存分析简介
-
- 描述与比较
- 影响因素分析
- 生存分析思路
-
- 生存曲线的比较
- 等比例风险的cox回归
- 非等比例风险的cox回归
- 第十章 一般线性模型与广义xianxiang
-
- 简介
-
- 一般线性模型
- 广义线性模型
- sas实现
-
- 协方差分析
- poisson回归分析
- 第十章 多水平数据的SAS分析
-
- 多水平数据常用统计方法
-
- 多水平模型
- 广义估计方程
- 分析思路
-
- 定量资料的多水平数据分析
- 分类资料的多水平数据分析
第二章医学研究设计与SAS实现
科研设计思路
样本含量估计
样本含量估计之前需要实现确定以下因素
一类错误
二类错误
单侧检验/双侧检验
其他
实验设计
科研设计的sas实现
步骤1:确定设计方法
步骤2:样本含量估计
步骤3:研究对象随机分组
完全随机设计
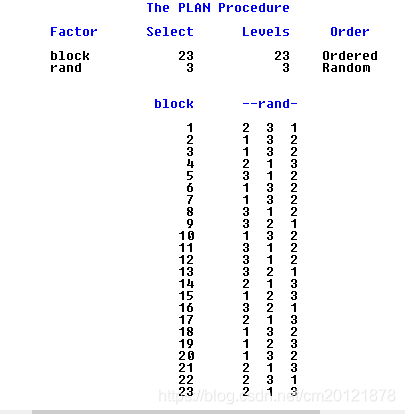
随机区组设计
析因设计
关系型研究
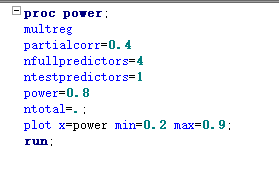
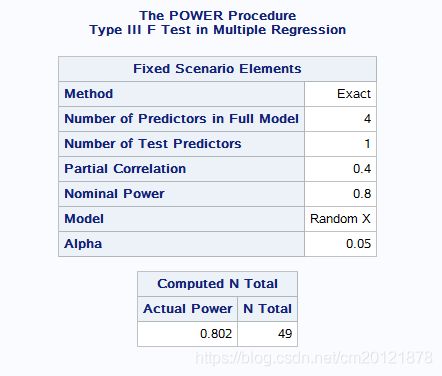

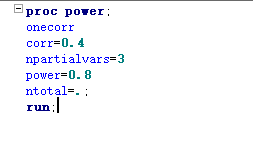
在样本量估计时,只要确定研究因素和校正因素,相关和回归所求的样本量估计值是一致的
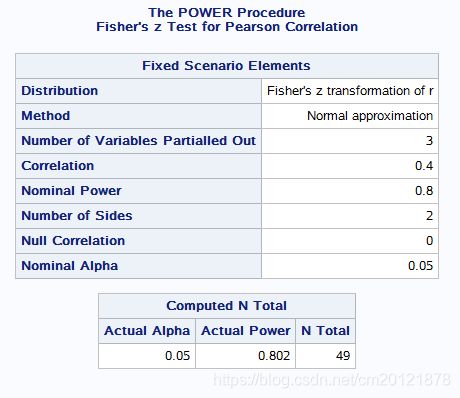
第三章 统计描述与SAS分析
统计描述及sas命令简介
定量资料的统计描述
分类资料的统计描述
第四章 定量资料的SAS统计分析
定量资料常用统计方法
t检验
proc ttest<选项>;
class 分组变量;
var 分析变量;
paired 变量1*变量2;
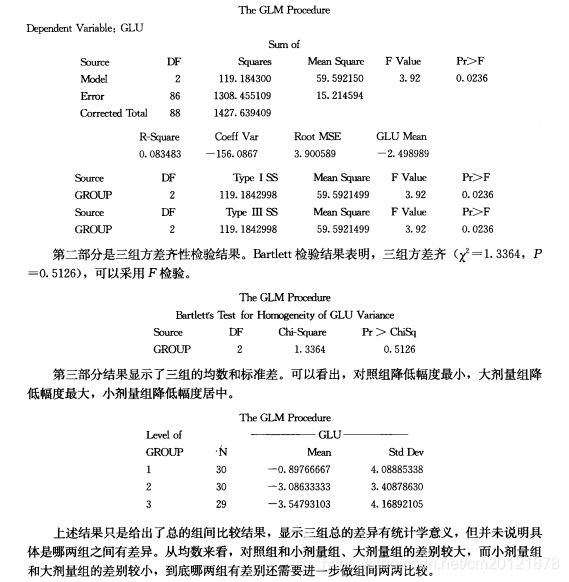
方差分析
proc glm<选项>;
class 分组变量;
model 分析变量=分组变量;
lsmeans 分组变量;
means 分组变量;
秩和检验
完全随机设计秩和检验:两组wilcoxon 多组kruskal-wallis
proc nparlway<选项>;
class 分组变量;
var 分析变量;
freq 频数;
随机区组设计秩和检验:friedman
proc freq<选项>;
table 区组分组变量分析变量/<选项>;
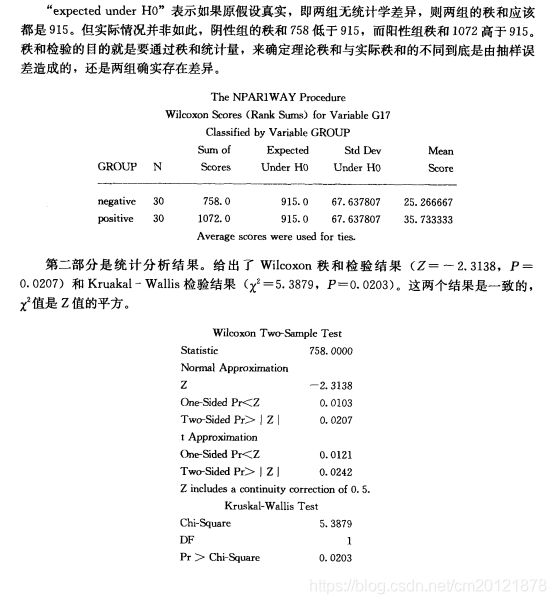
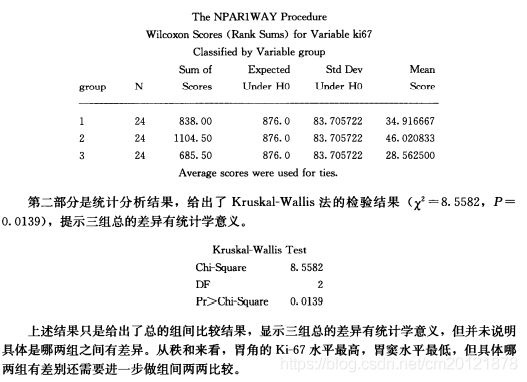
目前没有提供秩和检验两两比较的选项和菜单,可根据两两比较的公式自己编程来实现结果的输出,还可以采用基于秩的方差分析实现


多重检验
proc multtest<选项>;
class 分组变量;
test 分析变量<选项>;
正态性检验
对于多组数据比较,只有每组数据均符合正态分布,才算符合正态分布
proc univariate normal;
class 分组变量;
var 分析变量;
run;
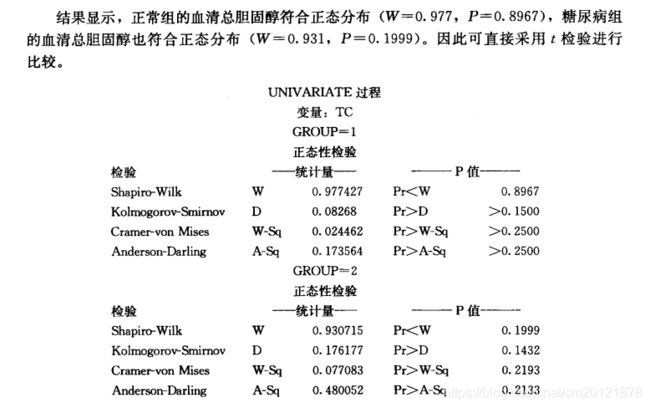
shapiro-wilk检验常用于50例以下的正态性检验,kolmogorov-smirnov检验多用于50例以上的正态性检验
把握度的计算
定量资料的分析思路
析因设计资料
配对设计资料
随机区组资料
多指标的组间比较
第五章 分类资料的SAS统计分析
分类资料常用统计方法
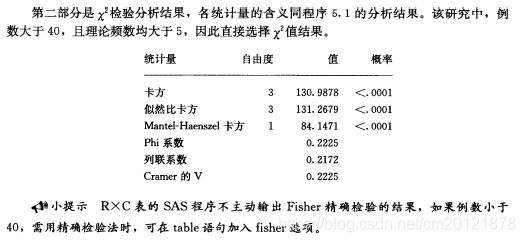
卡方检验的sas程序
proc freq<选项>;
table 行*列/ <选项>;
test 统计量关键字;
weight 权重变量;
by 分层变量;
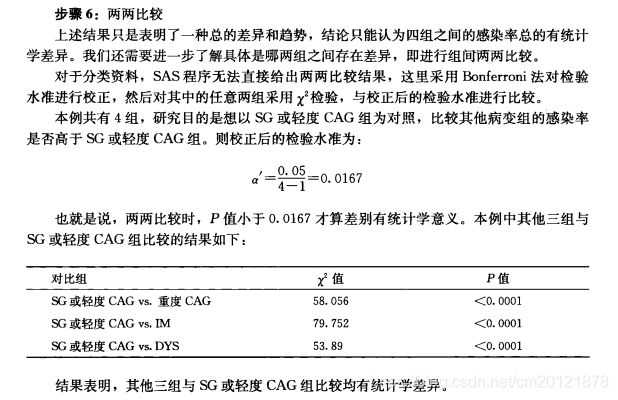
分类资料的分析思路
四格表资料
R*2表资料
趋势性检验
两两比较
2*C表无序资料
2*C表有序资料
配对资料
多层分类资料
第六章相关分析及SAS实现
相关分析
定量资料相关分析
proc corr<选项>;
var 变量1 变量2;
partial 变量1 变量2;
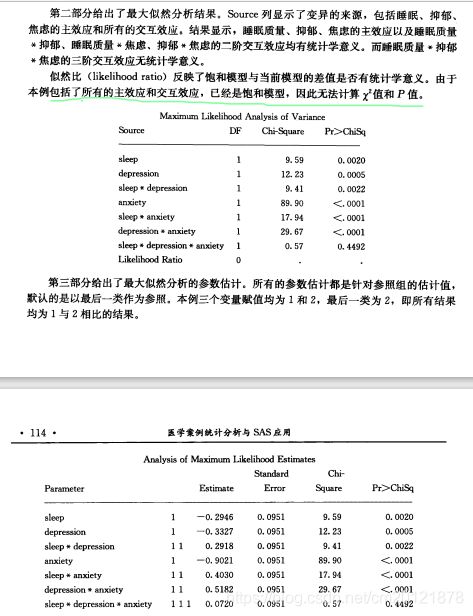
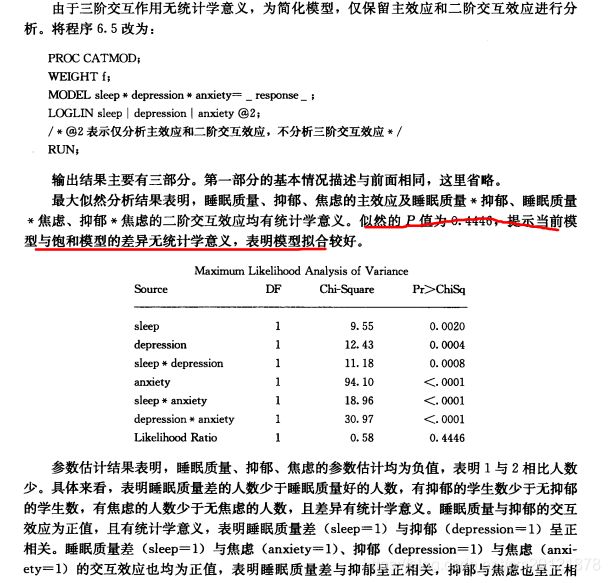
分类资料相关分析
对数线性模型
proc catmod<选项>;
weight 权重变量;
model 变量1变量2…=response;
loglin 效应;

相关分析的思路
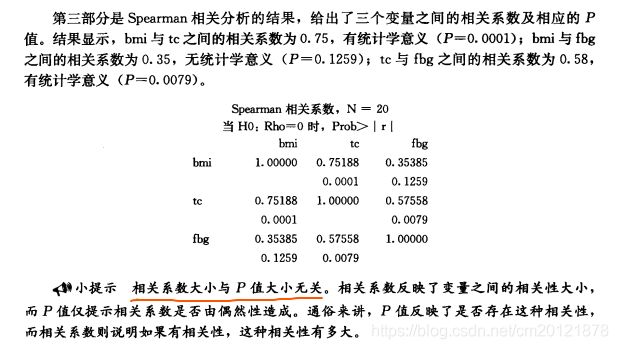
线性相关分析
proc corr pearson spearman;
var bmi tc fbg;
run;

偏相关分析
proc corr pearson spearman;
var bmi fbg;
partial tc;
run;

分类资料的相关性
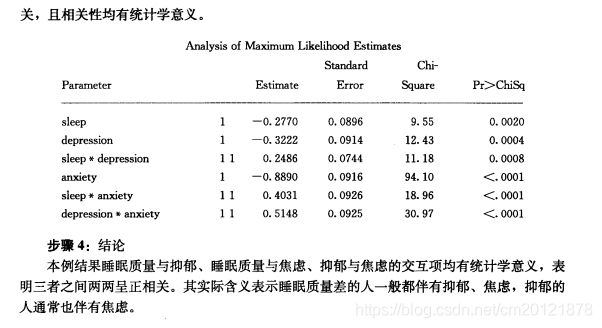
![]()
配对资料的相关性
多分类指标的相关性
第七章 线性回归与SAS分析
线性回归简介
proc reg<选项>;
model 因变量=自变量;
plot

线性回归的分析思路
简单线性回归分析
多重线性回归分析
应用条件检验
单因素分析
多因素分析
线性回归的替代方法
主成分回归
proc princomp<选项>;
var变量;
run;
共线性诊断
主成分分析
主成分回归
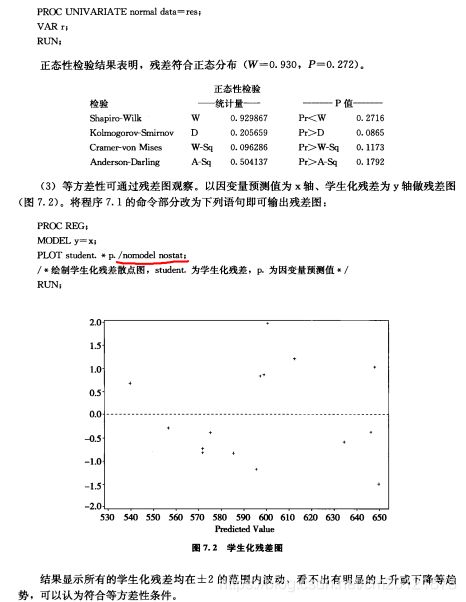
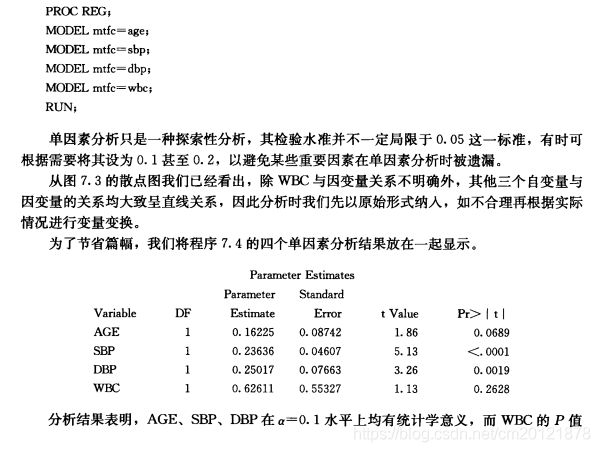
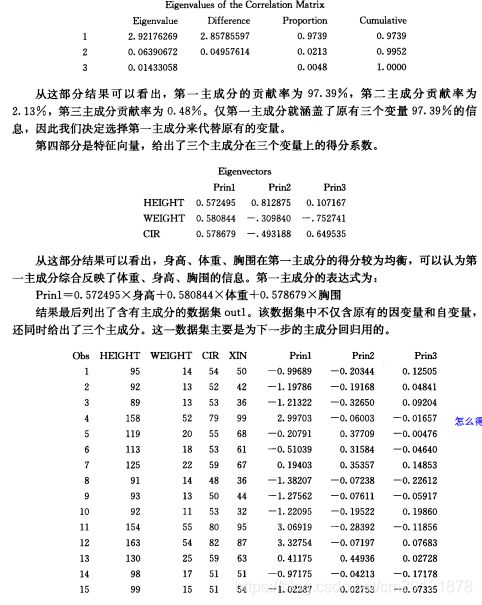
![]()
偏最小二乘回归
proc pls<选项>;
model 因变量=自变量;
run;
VIP指标
VIP代表自变量对模型拟合的重要程度
稳健回归
proc robustreg<选项>;
model 因变量=自变量;
run;

1.首先用线性回归对数据进行异常点诊断分析

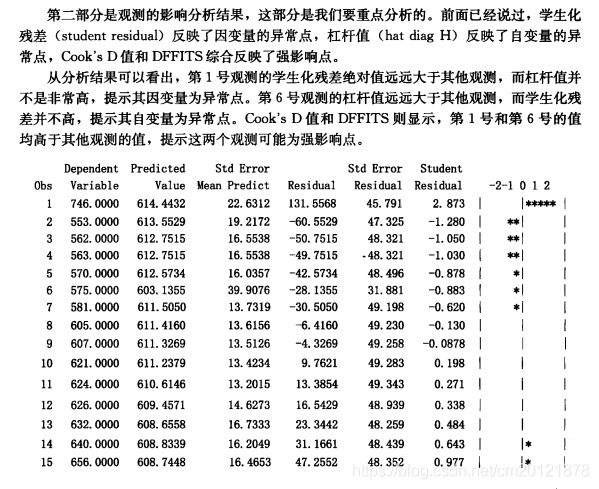
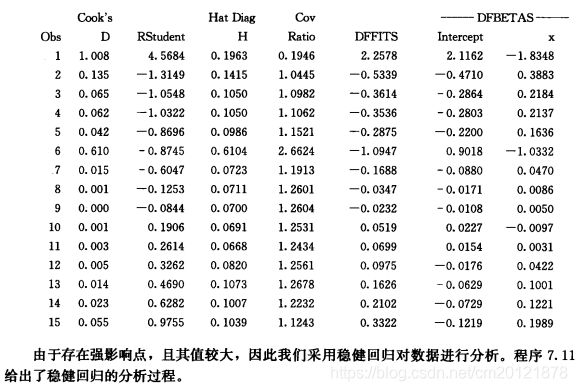
2.由于存在强影响点,且其值较大,进行稳健回归分析


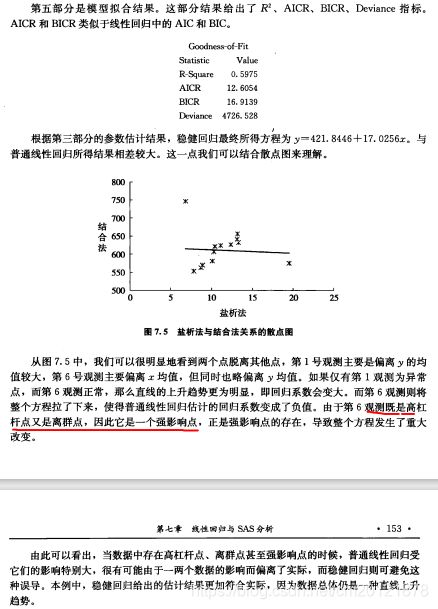
非参数回归
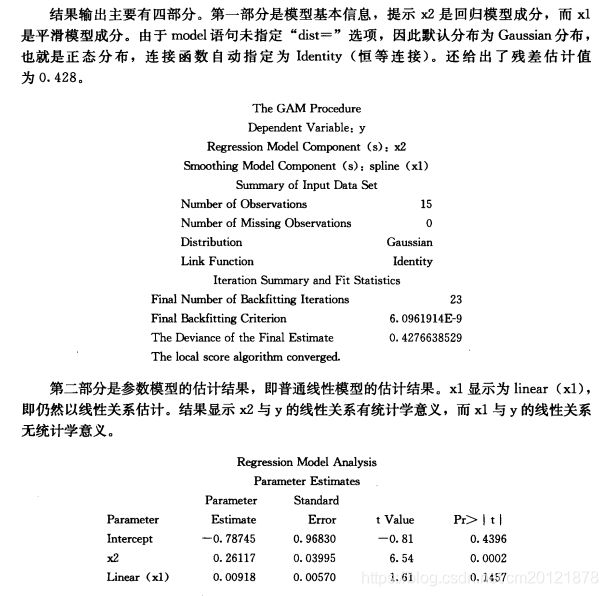
proc gam;
model 因变量=param(自变量)spline(自变量)loess(自变量)spline2(自变量);
score out=数据集;
run;
第八章logistics回归与SAS分析
logistics回归
proc logistic<选项>;
class 自变量;
model 因变量=自变量;
freq 变量;
logistics的分析思路
单因素
多因素
logistics回归的扩展
多项logistics回归
proc catmod<选项>;
direct 自变量;
model 因变量=自变量;




有序logistic回归
第九章 生存分析与SAS分析
生存分析简介
描述与比较
proc lifetest<选项>;
time 生存时间*生存状态(截尾值列表);
strata 分组变量;

影响因素分析
proc phreg<选项>;
model 生存时间*生存状态(截尾值列表)=分析变量<选项>;
output out=数据集名称 关键字1=名称1;

生存分析思路
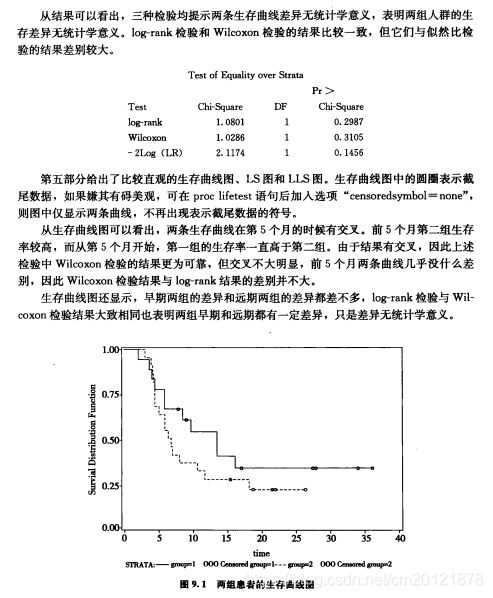
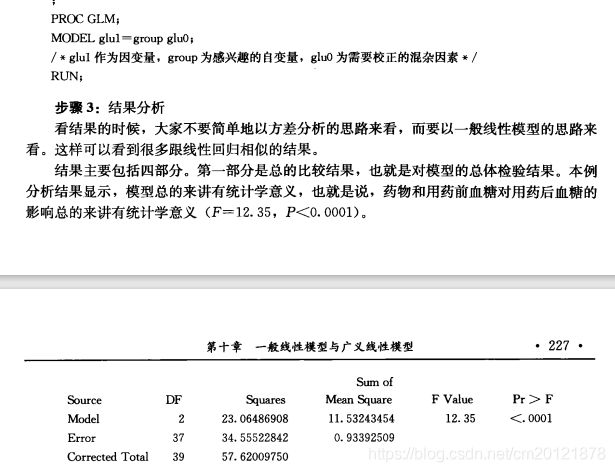
生存曲线的比较
等比例风险的cox回归
非等比例风险的cox回归
第十章 一般线性模型与广义xianxiang
简介
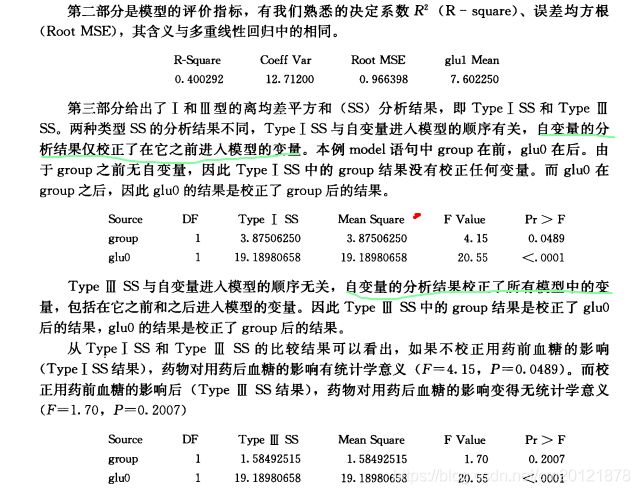
一般线性模型
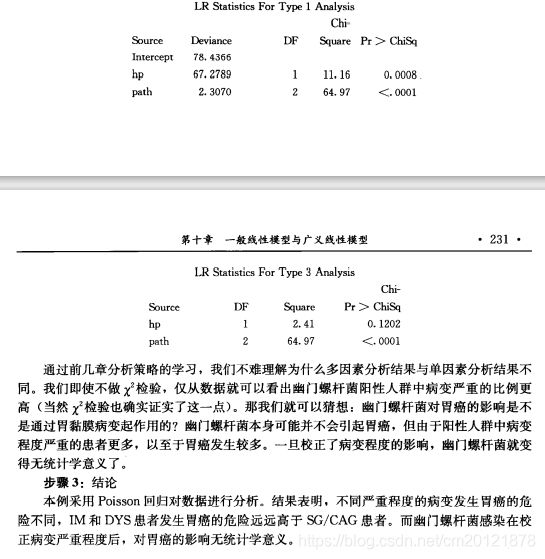
方差分析、简单线性回归、多重线性回归、协方差分析
proc glm<选项>;
class 分类变量;
model 因变量=自变量;
广义线性模型
二项分布、poisson分布、负二项分布
proc genmod<选项>;
class 分类变量;
model 因变量=自变量;
weight 变量;

sas实现
协方差分析
poisson回归分析
第十章 多水平数据的SAS分析
多水平数据常用统计方法
多水平模型
proc mixed 选项;
class 变量;
model 因变量=自变量/选项;
random 随机效应/选项;
广义估计方程
proc genmod<选项>;
class 分类变量;
model 因变量=自变量;
repeated subject=水平2变量/<选项>;
weigh 变量;

分析思路
定量资料的多水平数据分析
步骤1:确定分析方法
步骤2:空模型拟合–只包含截距项
步骤3:在模型中加入解释变量
步骤4:拟合随机斜率模型
步骤5:在模型中加入交互项
步骤6:结论