Deep drug-target binding affinity prediction with multiple attention blocks论文解读(一)2021SC@SDUSC
2021SC@SDUSC
论文解读:
摘要:
DTI预测背景:
药物靶间相互作用(DTI)预测由于其在药物发现过程中的重要地位而受到越来越多的关注。许多研究引入计算模型,将DTI预测作为回归任务,直接预测药物靶对的结合亲和性。
现有研究的缺陷与作者提出的模型:
然而,现有的研究在编码药物化合物时忽略了原子之间的本质相关性,仅仅通过连接来模拟药物靶对的相互作用。基于这些观察,在本研究中,我们提出了一个端到端的模型与多个注意块来预测药物靶对的结合亲和性得分。
作者模型的效果:
我们提出的模型提供了通过关系感知自我注意块编码原子之间的相关性和通过多头注意块建模药物表征和目标表征的相互作用的能力。在两个基准数据集KIBA和DAVIS上的DTI预测实验结果表明,该方法利用了关系感知自注意块编码的相关信息和多头注意块提取的交互信息,优于现有方法。
最大相对位置长度的影响实验:
作者对最大相对位置长度的影响进行实验,得出最大相对位置长度k∈{3,5}的最佳值。
利用作者的模型进行冠状病毒与药物的结合亲和力预测:
此外,我们应用我们的模型预测2019冠状病毒病(COVID-19)相关基因组序列和3137个fda批准的药物的结合亲和力。
介绍:
药物作用
药物通过与靶蛋白相互作用来激活或抑制靶蛋白的生物学过程。因此,识别新的药物靶标相互作用(DTIs)是药物发现领域的一个重要步骤,就像药物再利用一样。然而,过渡性的昂贵实验限制了识别新产品的过程。
研究分类
部分的研究将DTI预测任务视为一个二分类问题。他们关注DTI的存在,而其他一些研究将其作为回归任务,直接预测结合亲和力评分。在这里,结合亲和力分数描述了药物靶对相互作用的强度。在本研究中,我们主要研究药物靶点结合亲和力的预测。(后者)
DeepDTA:
近年来,深度学习方法被用于DTI预测。DeepDTA提出了一种卷积神经网络
基于神经网络的药物表征学习模型、目标表征学习模型及其相互作用预测。
论文中论述说,该模型已经在当前取得了很好的效果。
作者对现有方法的局限性分析:
针对药物化合物SMILES:
忽略了原子之间的相关性,包括原子相对位置信息以及原子多样性。
作者使用这样一个smiles序列来进行说明
针对药靶相互作用interaction:
现有的方法大多仅仅是简单地将药物与靶标的表示连接起来,从而对药物与靶标之间的相互作用进行建模。
笔者提出的模型:
基于以上研究,作者提出了这样一个模型,它是基于相对自注意力与多头注意力的。
relative self-attention可以编码药物化合物中原子间的相互关系信息。
multi-head编码了药物分子与蛋白质序列的相互作用信息。
关键词解释:
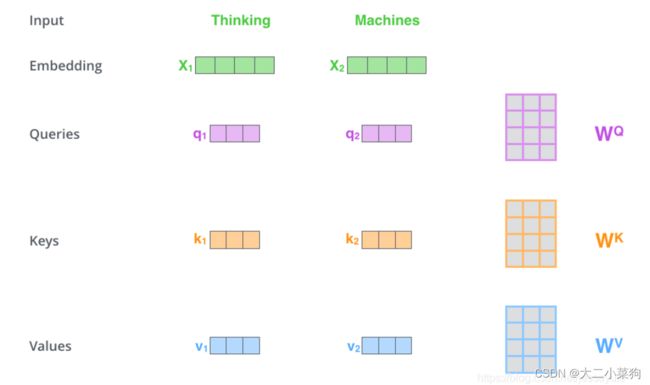
Multi-Head Attention
作者应该是想利用多头的机制来发掘药物化合物与蛋白质在不同位置在不同子空间下的信息,同时此设计出于并行计算的考虑,从而提高模型预测效果以及计算速度。
关于多头注意力机制的博客:https://blog.csdn.net/black_shuang/article/details/95384597
注意力机制
在NLP领域多用,作用是计算文本信息之间的相似度,在看上去没有任何关联的两个文本之间计算它们的相互联系。
计算使用矩阵之间的点乘 “·”,而非叉乘“x”,原因是,当两向量越接近时相似度应该是越大的。
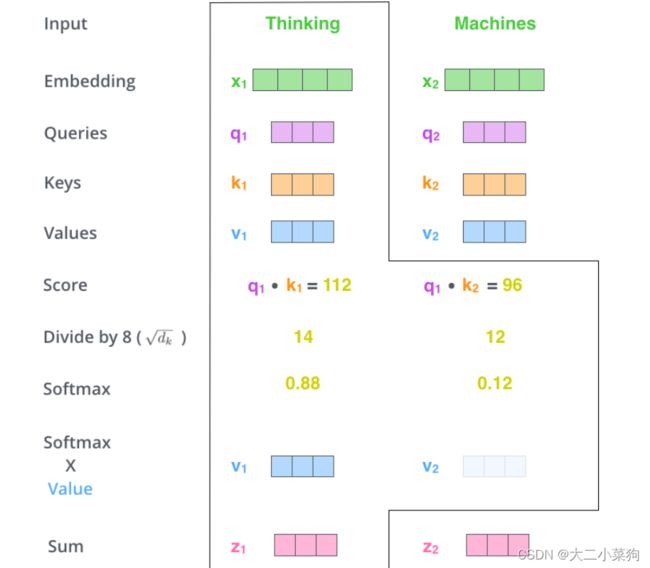
自注意力机制
计算同一文本自己与自己之间的相似度信息,从而得到上下文的联系信息,此处是为了得到smiles序列中原子之间的相对关系信息,从而提高对药物化合物的特征提取效果。
有个疑问(为啥蛋白质序列不用自注意力?是不是因为太长了,计算起来非常耗时间)
fasta长度
smiles长度
relation-aware self-attention
基于自注意力机制,利用定义好的原子关系字典,在计算attention时加上原子关系Wr的影响从而加强原子之间的相对关系信息。
池化
CSDN链接:https://blog.csdn.net/weixin_43336281/article/details/102149468