数据挖掘-实战记录(二)乳腺癌数据聚类实验及其分析报告
目录
一、数据探索
1.数据来源
2.数据描述
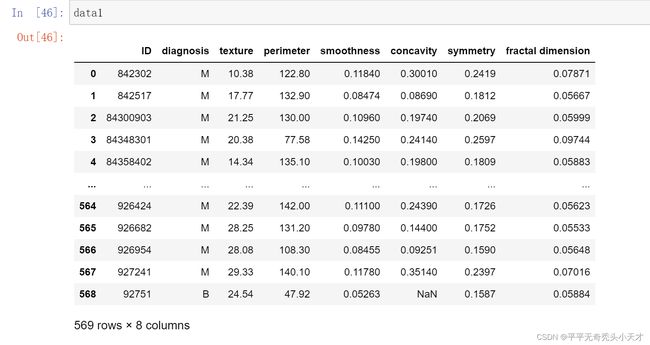
3.查看数据
5.研究各特征相互关系
二、数据预处理
1.处理缺失值
a)查看缺失值
b)缺失值处理
c)查看处理后的结果
2.查看异常值
3.处理相关值
三、数据分析(聚类)
1.k-means
a)k值评估
b)查看聚类效果
2.k-medoids
a)k-中点聚类
b)聚类可视化
3.层次聚类
a)层次聚类
4.SOM
a)聚类准备
b)聚类可视化
四、实验结果对比
五、参考资料
k-means
k-medoids
层次聚类
som
六、源码
一、数据探索
1.数据来源
http://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/
2.数据描述
该数据为乳腺癌诊断、癌变细胞特征值统计数据,初步推测可以根据该数据集聚类细胞癌变的特征属性
![]()
以下用表格形式描述数据各属性内容
| 1 |
ID |
ID号 |
|
| 2 |
Diagnosis |
诊断 |
|
| 3 |
radius |
半径 |
|
| 4 |
texture |
纹理 |
|
| 5 |
perimete |
周长 |
|
| 6 |
area |
面积 |
|
| 7 |
细胞一 |
smoothness |
平滑度 |
| 8 |
compactness |
紧密度 |
|
| 9 |
concavity |
凹度 |
|
| 10 |
concave points |
凹点 |
|
| 11 |
symmetry |
对称性 |
|
| 12 |
fractal dimension |
分形维数 |
|
| 13 |
radius |
半径 |
|
| 14 |
texture |
纹理 |
|
| 15 |
perimete |
周长 |
|
| 16 |
area |
面积 |
|
| 17 |
细胞二 |
smoothness |
平滑度 |
| 18 |
compactness |
紧密度 |
|
| 19 |
concavity |
凹度 |
|
| 20 |
concave points |
凹点 |
|
| 21 |
symmetry |
对称性 |
|
| 22 |
fractal dimension |
分形维数 |
|
| 23 |
radius |
半径 |
|
| 24 |
texture |
纹理 |
|
| 25 |
perimete |
周长 |
|
| 26 |
area |
面积 |
|
| 27 |
细胞三 |
smoothness |
平滑度 |
| 28 |
compactness |
紧密度 |
|
| 29 |
concavity |
凹度 |
|
| 30 |
concave points |
凹点 |
|
| 31 |
symmetry |
对称性 |
|
| 32 |
fractal dimension |
分形维数
|
|
3.查看数据
查看数据以及查看数据
以上结果为查看数据集中各数据类型
4.数据描述性分析
查看数据的最大值,最小值,四分之一值,均值,标准差
由上述结果不难看出,存在无效零值,因此需要进行数据预处理
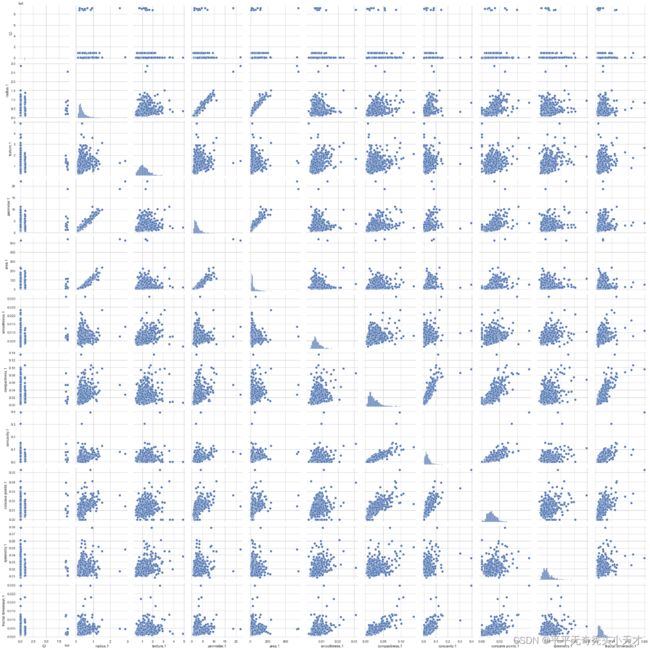
5.研究各特征相互关系
将数据集中三个细胞的十个特征值分为三个数据集data1,data2,data3,分别研究三个子数据集的各个特征值相互关系
特征矩阵
Data1特征矩阵:
Data2特征矩阵:
Data3特征矩阵:
特征相关系数图
总特征相关系数图
当数据集中的所有数据绘成一个特征相关系数,图片较为复杂以及影响实验观察,因此根据数据集本身是三个细胞的十个特征属性,分为三个子数据集然后分别对三个子数据集进行特征相关系数绘图,方便观察以及得出结论,且不影响最终结论。
Data1数据集中各个数据特征值相关系数图
Data1数据集中半径与周长、面积是大于0.90严格相关的,凹度凹点以及紧密度也是相关系数大于0.70的相关。
Data2数据集中各个数据特征值相关系数图
Data2数据集中半径与周长、面积是大于0.90严格相关的,凹度凹点以及紧密度也是相关系数大于0.70的相关。
Data3数据集中各个数据特征值相关系数图
Data3数据集中半径与周长、面积是大于0.90的严格相关的,凹度凹点以及紧密度也是相关系数大于0.70的相关。
综上所述,可以在数据预处理的时候将半径、周长、面积删去其中两列,不会影响数据集聚类结果,将凹度凹点以及紧密度删去其中两项,在一定程度上也不会影响数据集聚类结果。
二、数据预处理
1.处理缺失值
a)查看缺失值
缺失值如下图结果所示,统计的无意义零值(缺失值)wei 13个,在总数据集569条中为少数。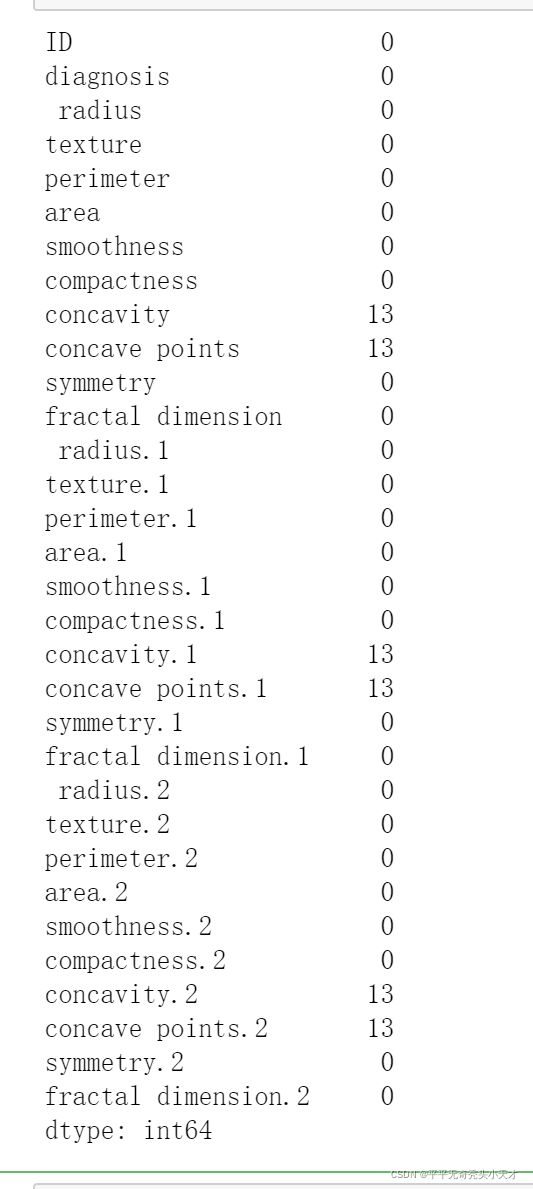
b)缺失值处理
由于缺失值仅有13条,因此删除对应缺失值所在行不会大幅度影响结果聚类,因此在本次实验中采取删除缺失值所在行的缺失值处理方法
标记缺失值后删除缺失值所对应行。
c)查看处理后的结果
用查看缺失值的方式查看
2.查看异常值
查看异常值
尝试着查看数据集data_new的异常值,由于总属性种类过多,对分析结果造成较大影响,因此还是将数据依照三个细胞分为三个子数据集查看异常值
Data1异常值查看
异常值结果如下图所示
Data2异常值查看
异常值结果如下图所示
Data3异常值查看
异常值结果如下图所示
3.处理相关值
由特征值相互关系中分析出:半径、周长、面积删去其中两列,不会影响数据集聚类结果,将凹度凹点以及紧密度删去其中两项,在一定程度上也不会影响数据集聚类结果,因此在处理相关值时删除半径、面积,凹点、紧密度这四个特征值
结果如下图所示
将处理好的data_new数据依次分给子数据集
聚类时时,由于ID与判断是否得病两列会影响聚类,因此把两列特征值删去
三、数据分析(聚类)
1.k-means
a)k值评估
数据集评估
肘部法则
人工选择k值存在偶然性以及误差,因此查阅资料可知,通过肘部发展迭代K值最终在快速下降趋于平缓下降的转折点选择出聚类的最好情况。
(kmeans聚类分析——Python实现_thisissally的博客-CSDN博客)
由图可以看出在2,3类都有可能成为聚类情况,因此再对2,3类用系数评估
当k=2时聚类
![]()
显示中心点
当k=3时
查看中心点
![]()
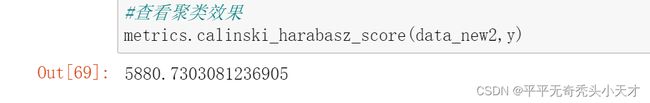
b)查看聚类效果
聚类效果好坏的判断有三种方式,本实验选用Calinski-Harabasz(CH)方式判定聚类效果。
K=2
K=3
输出聚类结果
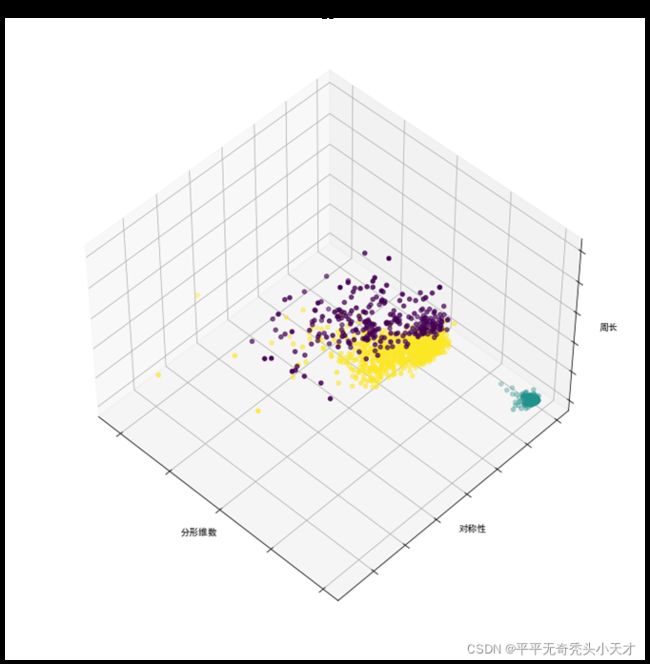
c)聚类可视化
可视化三维图时data_new1特征值数据的横向分布不利于绘图,因此将分散的属性值结合为一个,因此转换为data_new2数据做聚类可视化分析。
2.k-medoids
a)k-中点聚类
直接使用data_new2数据框进行k-中点聚类
下载pyclust库,使用pyclust库下的k-Medoids板块
聚类结果如下,
b)聚类可视化
分别为二类、三类、四类、五类的效果,由结果不难看出,二类最为合适,彻底聚类成功,三类效果次于两类,有略微的交杂。四类五类结果明显不如前者。
3.层次聚类
a)层次聚类
先进行sklearn的层次聚类
聚类结果如下
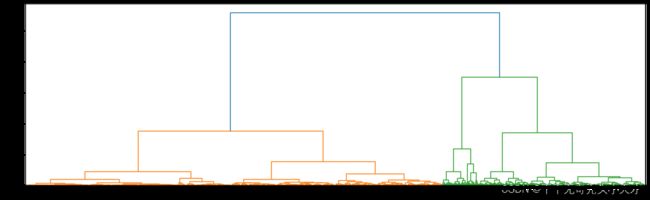
b)聚类可视化
结果如下
明显聚类为三类更好的描述数据集的聚类情况。
4.SOM
a)聚类准备
下载Minisom库
开始聚类
查看聚类结果
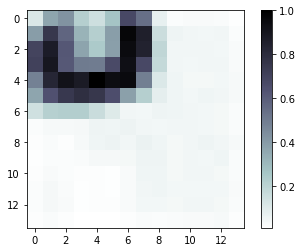
b)聚类可视化
热力图:
散点图
综上分为三类
合并最终聚类结果
四、实验结果对比
在各种聚类方式中,k-medoids需要进行大量的机器运算,结果出来的也十分缓慢,在实际研究中不建议采用,尤其当实验数据较大时,严重耽误时间导致效率过低,在运行集中聚类方式时,更建议采用k-means算法,该算法较为完善且在网络上有大量参考文献与资料,可视化也是多种多样的,其次就是SOM算法,运行速度较快,结果较清晰明显。
层次聚类分析与k-medoids在实验中的运行速度以及运行效果并没有另外两种好。
五、参考资料
k-means
- k-means+python︱scikit-learn中的KMeans聚类实现( + MiniBatchKMeans) - 知识天地 - 博客园
- kmeans聚类分析——Python实现_thisissally的博客-CSDN博客
k-medoids
- k-medoid(k中心点)聚类算法Python实现_Trident_lin的博客-CSDN博客_k中心点聚类算法
层次聚类
- (数据科学学习手札13)K-medoids聚类算法原理简介&Python与R的实现 - 费弗里 - 博客园
- 【Python-ML】SKlearn库层次聚类凝聚AgglomerativeClustering模型_医疗影像检索-CSDN博客_python sklearn 层次聚类
- 机器学习之层次聚类(hierarchical clustering)_Cherish0719的博客-CSDN博客_机器学习层次聚类
- 聚类热图分类注释_Python可视化matplotlib&seborn15-聚类热图clustermap(建议收藏)..._伥鬼的博客-CSDN博客
som
- https://github.com/JustGlowing/minisom
- 自组织神经网络(SOM)的Python第三方库minisom聚类功能实现_应无所住而生其心-CSDN博客_minisom python
- SOM(自组织映射神经网络)——案例篇 - 知乎
六、源码
# 导入相关包
import numpy as np
import pandas as pd
from numpy import nan
#%matplotlib inline
import matplotlib.pyplot as plt
plt.style.use('seaborn')
import seaborn as sns
sns.set_style("whitegrid")
#k-means
from sklearn.cluster import KMeans
from sklearn import metrics
from mpl_toolkits.mplot3d import Axes3D
plt.rcParams['font.sans-serif']=['SimHei'] #用来正常显示中文标签
plt.rcParams['axes.unicode_minus']=False #用来正常显示负号
#k-medoid
from matplotlib import pyplot
from pyclust import KMedoids
from sklearn.manifold import TSNE
#层次聚类
from scipy.spatial.distance import pdist,squareform
from scipy.cluster.hierarchy import linkage
from scipy.cluster.hierarchy import dendrogram
from sklearn.cluster import AgglomerativeClustering
plt.rcParams['font.sans-serif']=['SimHei'] #用来正常显示中文标签
plt.rcParams['axes.unicode_minus']=False #用来正常显示负号
#SOM
from sklearn.model_selection import train_test_split
from minisom import MiniSom
import math
# 导入数据集
#查看数据
data = pd.read_csv('D:\数据挖掘\实验二\wdbc.csv')
data.info()
print(data.describe())
#%matplotlib inline
import matplotlib.pyplot as plt
plt.style.use('seaborn')
import seaborn as sns
sns.set_style("whitegrid")
#数据探索
l1 = [' radius','texture','perimeter','area','smoothness','compactness','concavity','concave points','symmetry','fractal dimension']
l2 = [' radius.1','texture.1','perimeter.1','area.1','smoothness.1','compactness.1','concavity.1','concave points.1','symmetry.1','fractal dimension.1']
l3 = [' radius.2','texture.2','perimeter.2','area.2','smoothness.2','compactness.2','concavity.2','concave points.2','symmetry.2','fractal dimension.2']
data1 = data[['ID','diagnosis',' radius','texture','perimeter','area','smoothness','compactness','concavity','concave points','symmetry','fractal dimension']]
data2 = data[['ID','diagnosis',' radius.1','texture.1','perimeter.1','area.1','smoothness.1','compactness.1','concavity.1','concave points.1','symmetry.1','fractal dimension.1']]
data3 = data[['ID','diagnosis',' radius.2','texture.2','perimeter.2','area.2','smoothness.2','compactness.2','concavity.2','concave points.2','symmetry.2','fractal dimension.2']]
#各特征值与结果的关系
# 设置颜色主题
antV = ['#1890FF', '#2FC25B', '#FACC14', '#223273', '#8543E0', '#13C2C2', '#3436c7', '#F04864']
#生成各特征之间的关系矩阵图
g = sns.pairplot(data=data1, palette=antV)
f = sns.pairplot(data=data2, palette=antV)
k = sns.pairplot(data=data3, palette=antV)
# 查看特征间的相关性
#总特征相关系数图
pearson_mat = data.corr(method='spearman')
plt.figure(figsize=(15,15))
ax = sns.heatmap(pearson_mat,square=True,annot=True,cmap='YlGnBu')
bottom, top = ax.get_ylim()
ax.set_ylim(bottom + 0.5, top - 0.5)
plt.show()
#data1
pearson_mat = data1.corr(method='spearman')
plt.figure(figsize=(15,15))
ax = sns.heatmap(pearson_mat,square=True,annot=True,cmap='Purples')
bottom, top = ax.get_ylim()
ax.set_ylim(bottom + 0.5, top - 0.5)
plt.show()
#data2
pearson_mat = data2.corr(method='spearman')
plt.figure(figsize=(15,15))
ax = sns.heatmap(pearson_mat,square=True,annot=True,cmap='OrRd')
bottom, top = ax.get_ylim()
ax.set_ylim(bottom + 0.5, top - 0.5)
plt.show()
#data3
pearson_mat = data3.corr(method='spearman')
plt.figure(figsize=(15,15))
ax = sns.heatmap(pearson_mat,square=True,annot=True,cmap='Greens')
bottom, top = ax.get_ylim()
ax.set_ylim(bottom + 0.5, top - 0.5)
plt.show()
#数据预处理
#缺失值处理
# 按列统计零的值
num_missing = (data == 0).astype(int).sum(axis=0)
# 输出结果
print(num_missing)
#标记缺失值
data = data.replace(0, nan)
#处理缺失值少的特征值删除这两列中缺失值对应的行数
data_new = data.dropna(axis=0,subset = ["concavity", "concave points"])
num_missing_new = (data_new == 0).astype(int).sum(axis=0)
print(num_missing_new)
#查看异常值
#data1
numeric_columns = []
object_columns = []
for c in data1.columns[0:-1]:
if data1[c].dtype == 'object':
object_columns.append(c)
else:
numeric_columns.append(c)
fig = plt.figure(figsize=(20,20))
for i,col in enumerate(numeric_columns):
ax = fig.add_subplot(5,3,i+1)
sns.boxplot(data1[col],orient='v',ax=ax)
plt.xlabel(col)
plt.show()
#data2
numeric_columns = []
object_columns = []
for c in data2.columns[0:-1]:
if data2[c].dtype == 'object':
object_columns.append(c)
else:
numeric_columns.append(c)
fig = plt.figure(figsize=(20,20))
for i,col in enumerate(numeric_columns):
ax = fig.add_subplot(5,3,i+1)
sns.boxplot(data2[col],orient='v',ax=ax)
plt.xlabel(col)
plt.show()
#data3
numeric_columns = []
object_columns = []
for c in data3.columns[0:-1]:
if data3[c].dtype == 'object':
object_columns.append(c)
else:
numeric_columns.append(c)
fig = plt.figure(figsize=(20,20))
for i,col in enumerate(numeric_columns):
ax = fig.add_subplot(5,3,i+1)
sns.boxplot(data3[col],orient='v',ax=ax)
plt.xlabel(col)
plt.show()
#处理相关值
#删除特征值
data_new0 = data_new.drop([' radius','area','compactness','concave points',' radius.1','area.1','compactness.1','concave points.1',' radius.2','area.2','compactness.2','concave points.2'],axis=1,inplace=True)
data1 = data[['ID','diagnosis','texture','perimeter','smoothness','concavity','symmetry','fractal dimension']]
data2 = data[['ID','diagnosis','texture.1','perimeter.1','smoothness.1','concavity.1','symmetry.1','fractal dimension.1']]
data3 = data[['ID','diagnosis','texture.2','perimeter.2','smoothness.2','concavity.2','symmetry.2','fractal dimension.2']]
#k-means
#将ID,diagnosis两列与数据类型不合的特征值删去
#可视化
data_new1 = data_new.drop(['ID','diagnosis'],axis=1)
#将处理好的数据写入data_new2,所有聚类都使用data_new2数据集
data_new2 = pd.read_csv('D:\数据挖掘\实验二\data_new2.csv')
#寻找合适的K值
sse = []
for i in range(1,11): # 循环使用不同k测试结果
kmeans = KMeans(n_clusters = i, init = 'k-means++', random_state = 42)
kmeans.fit(data_new2)
sse.append(kmeans.inertia_) # kmeans.inertia_是每类数据到其中心点的距离之和。值越小,聚类越好。类别越多,k越大,值越小。
plt.plot(range(1,11), sse)
plt.title('The Elbow Method')
plt.xlabel('Number of clusters')
plt.ylabel('SSE')
plt.show()
#k值设为2
estimator = KMeans(n_clusters=2)#构造聚类器
estimator.fit(data_new2)#聚类
y = estimator.fit_predict(data_new2)
label_pred = estimator.labels_ #获取聚类标签
centroids = estimator.cluster_centers_ #获取聚类中心
inertia = estimator.inertia_ # 获取聚类准则的总和
#查看聚类效果
metrics.calinski_harabasz_score(data_new2,y)
#k值设为3
estimator = KMeans(n_clusters=3)#构造聚类器
estimator.fit(data_new2)#聚类
y = estimator.fit_predict(data_new2)
label_pred = estimator.labels_ #获取聚类标签
centroids = estimator.cluster_centers_ #获取聚类中心
inertia = estimator.inertia_ # 获取聚类准则的总和
metrics.calinski_harabasz_score(data_new2,y)
#结果
#输出原数据及类别
e=pd.concat([data_new2,pd.Series(label_pred,index=data_new2.index)],axis=1)
e.columns=list(data_new2.columns)+['result']
#可视化
#雷达图
'''
未完成
labers = estimator.labels_.tolist()
newdata = pd.DataFrame(labers,index = [labers])
columns = ['rank', 'title', 'cluster', 'genre']
newdata [0].value_counts()
'''
#3D图
labels = estimator.labels_
fig = plt.figure(1, figsize=(10, 10))
ax = Axes3D(fig, rect=[0, 0, .95, 1], elev=48, azim=134)
ax.scatter(data_new2.values[:, 1], data_new2.values[:, 0],data_new2.values[:, 3], c= labels.astype(np.float))
ax.w_xaxis.set_ticklabels([])
ax.w_yaxis.set_ticklabels([])
ax.w_zaxis.set_ticklabels([])
ax.set_xlabel('凹度')
ax.set_ylabel('平滑度')
ax.set_zlabel('纹理')
ax.set_title("3类")
ax.dist = 12
plt.show()
#k- medoids聚类
#准备可视化需要的降维数据
data_TSNE = TSNE(learning_rate=10).fit_transform(data_new2)
data_TSNE
#对不同的k进行试探性K-medoids聚类并可视化
data_new2 = pd.read_csv('D:\数据挖掘\实验二\data_new2.csv')
plt.figure(figsize=(12,8))
for i in range(2,6):
k = KMedoids(n_clusters=i,distance='euclidean',max_iter=1000).fit_predict(data_new2.values)
colors = ([['red','blue','black','yellow','green'][i] for i in k])
plt.subplot(219+i)
plt.scatter(data_TSNE[:,0],data_TSNE[:,1],c=colors,s=10)
plt.title('K-medoids Resul of '.format(str(i)))
#结果
#输出原数据及类别
k = KMedoids(n_clusters=3,distance='euclidean',max_iter=1000).fit_predict(data_new2.values)
i=pd.concat([data_new2,pd.Series(k,index=data_new2.index)],axis=1)
i.columns=list(data_new2.columns)+['result']
i
#层次聚类
#标准化
data_new2 = pd.read_csv('D:\数据挖掘\实验二\data_new2.csv')
a=round(
(data_new2-data_new2.min())/(data_new2.min()),
3)
k=3
model=AgglomerativeClustering(n_clusters=k,linkage='ward')
model.fit(a)
#结果
#输出原数据及类别
r=pd.concat([data_new2,pd.Series(model.labels_,index=data_new2.index)],axis=1)
r.columns=list(data_new2.columns)+['result']
#聚类可视化
plt.figure(figsize=(20,6))
Z = linkage(a, method='ward', metric='euclidean')
p = dendrogram(Z)
plt.show()
'''
#未完成
style=['ro-','go-','bo-']
xlabels= ['texture','perimeter','smoothness','concavity','symmetry','fractal dimension']
for i in range(k): #注意作图、做出不同样式
plt.figure()
tmp=r[r[u'result']==i].iloc[:,:4] # 提取每一类
for j in range(len(tmp)):
plt.plot(range(1,5),tmp.iloc[j],style[i])
plt.xticks(range(1,5),xlabels,rotation=20) #坐标标签
plt.subplots_adjust(bottom=0.15) # 调整底部
'''
#SOM
data = data_new2 = pd.read_csv('D:\数据挖掘\实验二\data_new2.csv')
# 数据规范化
data = (data - np.mean(data, axis=0)) / np.std(data, axis=0)
data = data.values
# 初始化和训练
som_shape = (1, 3)
som = MiniSom(som_shape[0], som_shape[1], data.shape[1], sigma=.5, learning_rate=.5,
neighborhood_function='gaussian', random_seed=10)
som.train_batch(data, 500, verbose=True)
# 每个神经元代表一个簇
winner_coordinates = np.array([som.winner(x) for x in data]).T
#利用np.ravel_多重指数,我们将二维
#一维索引的坐标
cluster_index = np.ravel_multi_index(winner_coordinates, som_shape)
#结果
#输出原数据及类别
o=pd.concat([data_new2,pd.Series(cluster_index,index=data_new2.index)],axis=1)
o.columns=list(data_new2.columns)+['result']
o
# #使用data的前2个维度对簇进行一维索引绘制的坐标
for c in np.unique(cluster_index):
plt.scatter(data[cluster_index == c, 0],
data[cluster_index == c, 1], label='cluster='+str(c), alpha=.7)
#绘制中心点
for centroid in som.get_weights():
plt.scatter(centroid[:, 0], centroid[:, 1], marker='x',
s=30, linewidths=35, color='k', label='centroid')
plt.legend()