SPM一阶水平分析
在进行一阶水平分析时要保证你的数据是经过预处理的。
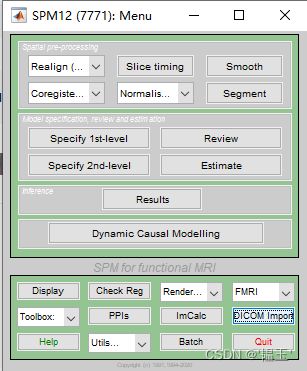
点击Specify 1st-level
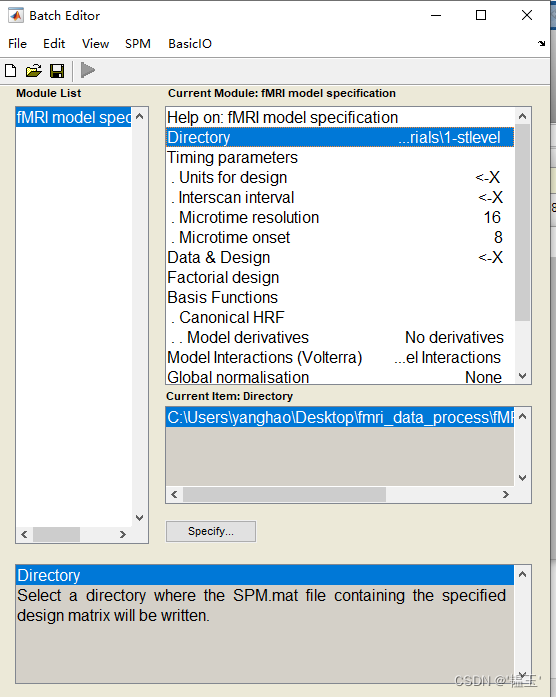
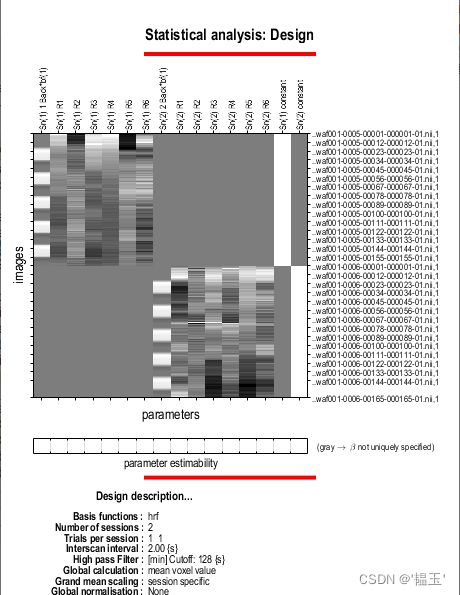
Directory是选择输出文件夹 ,units for design有seconds和scans两个选项,这里选择scans,因为scans比较简单。
interscan interval: 就是TR,Microtime resolution:是切片数量(slice number),MIcroctime oneset:参考切片(reference slice)
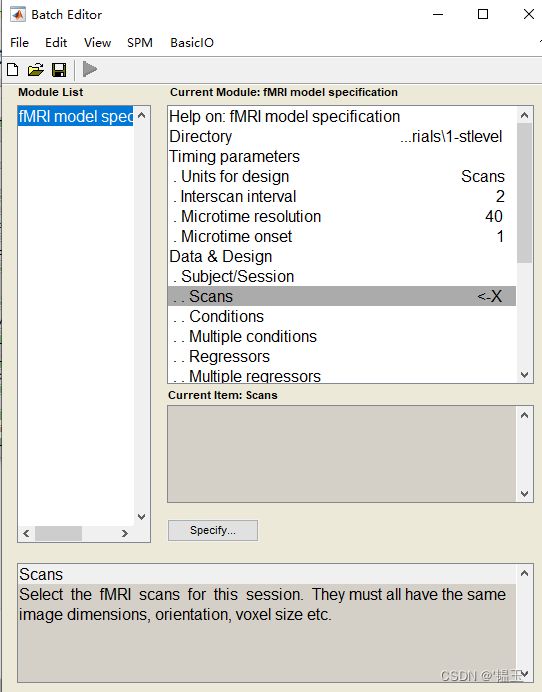
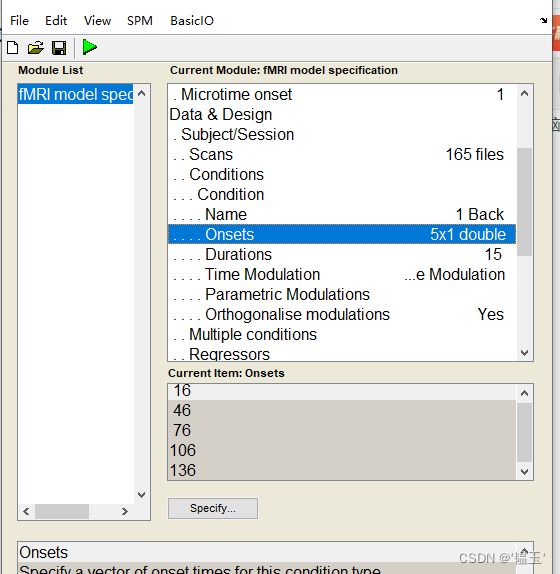
Data Design:双击添加数据,
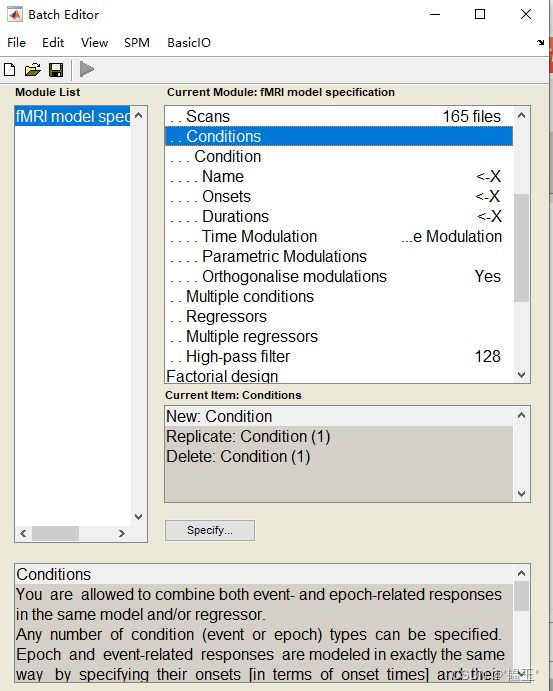
conditions:有几组对比条件就添加几个,会作为GLM模型的回归因子(![]() )
)
Scans进行分析的选择fmri文件,双击conditions创建一个condition
name:起一个名字
Onesets:每个刺激块开始的时间
Durations :每个刺激块持续的时间
Mutiple conditions:可以添加经过头动矫正时获得的rp开头的txt文件。
以我的数据来举例说明:
一个run有5个block,每个block持续30s,TR为2s,所以每个block有15个scans,每个刺激快之后是30s的休息,第一个刺激快的开始时间切片为16,总共有165个全脑,故Onesets填16:30:165
Duration,为15
完成之后,在menu窗口:点击Estimate,进行一阶水平分析
选择上一步生成的SPM.mat文件。 点击RUN
完成之后,在menu窗口点击Results,和上一步中一样选择,SPM.mat文件,出现如下窗口,
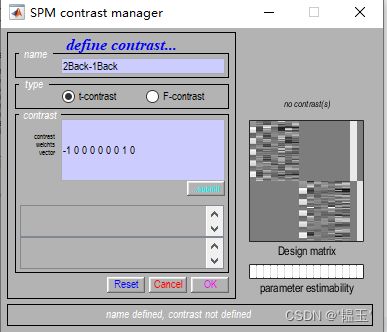
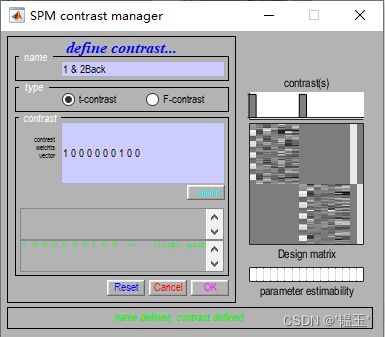
然后点击Define new contrast,定义新的对比分析
name:输入自定义对比分析的名字,比如我输入的是task-rest,即任务态和静息态的对比
type:选择默认的t检验
contrast :输入需要对比的![]() 因子系数,因为我只定义了一个condition,所以只在第一个
因子系数,因为我只定义了一个condition,所以只在第一个![]() 因子输入1,其他头动因子为零,这样就是任务态和静息态的对比。当你有多个conditions时,可以使用不同的系数,来比较不同条件的对比。比如你有两个条件,positive,negetive,和6个头动因子,总共有8个回归因子,系数可以定义为1 -1 0 0 0 0 0 0 ,即两个条件间的对比
因子输入1,其他头动因子为零,这样就是任务态和静息态的对比。当你有多个conditions时,可以使用不同的系数,来比较不同条件的对比。比如你有两个条件,positive,negetive,和6个头动因子,总共有8个回归因子,系数可以定义为1 -1 0 0 0 0 0 0 ,即两个条件间的对比
然后点击OK
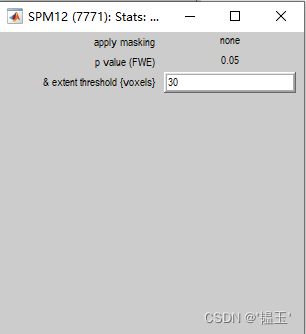
是否使用mask,这里选择none
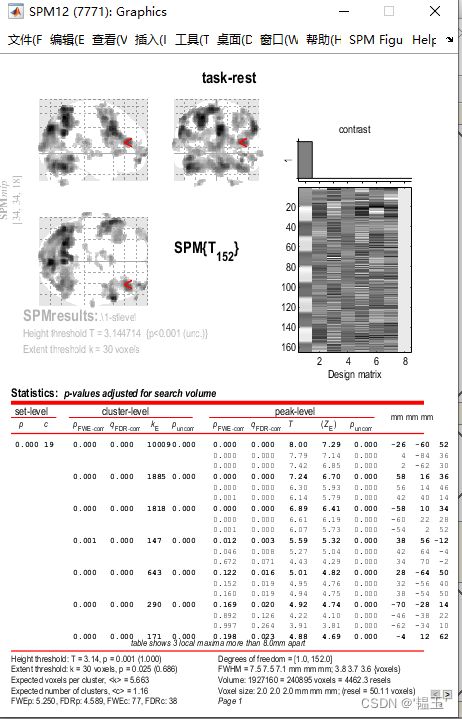
FWE是比较严格条件,这里选择FWE,p值为0.05, voxels大小的阈值为30,避免噪声影响。
这里的体素大小太了,以为设置的条件太严格,把FWE去掉,选择p值为0.001
可以发现体素的集合变大了
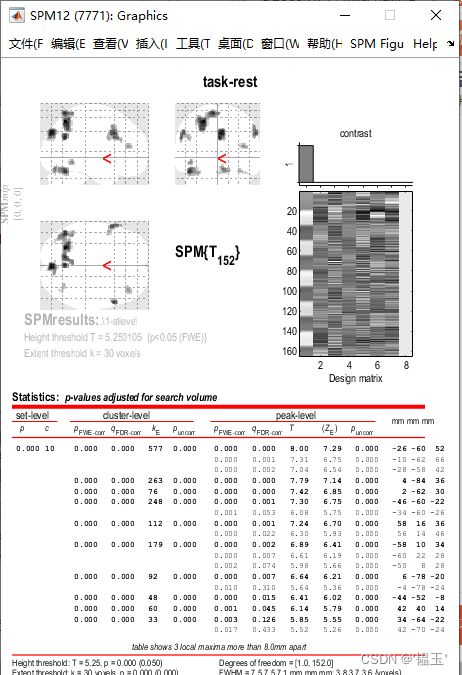
在这个页面,可以看到t值为5.36
选择overlays-->sections-->你的T1结构像,可以叠加到T1结构像上去看
定义两个conditions的方法:
第一完成第一个seesion,之后点击replicateSession(1),得到第二个Session,需要更改第二个session中的Scans,conditions中的name,multiple regressor 注意对应相应的文件
点击运行之后会有得到下图:左上一个为1 Back,右下为2 Back
之后的操作与上面一致。
定义contrasts,会有些不同, 不需要写完所有的0,SPM会自动补齐0
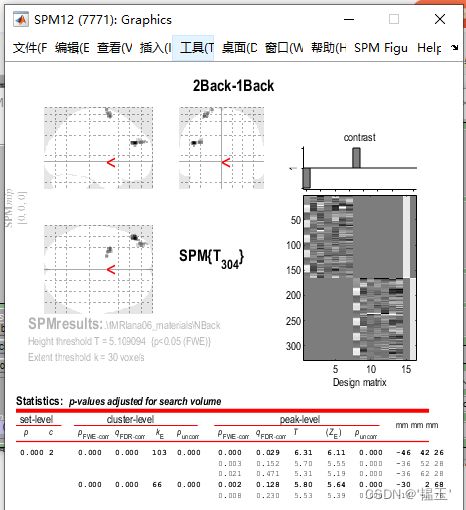
这里定义的是2Back与1Back的对比
还可以定义1&2Back ,这是任务态与静息态的对比
首先来看第一个对比:选择FWE,p: 0.05,voxels: 30
会发现只有两个很小的区域
再看第二个contrast:切换到第二个contrast
结果图: