Python实现K-Means聚类(案例:用户分类)
目录
K-Means聚类算法介绍
K-Means聚类算法基础原理
K-Means聚类算法实现流程
开始做一个简单的聚类
数据导入
数据探索
开始聚类
查看输出结果
聚类质心
K-Means聚类算法的评估指标
真实标签已知
真实标签未知
实用案例:基于轮廓系数来选择最佳的n_clusters
绘制轮廓系数分布图
结果对比
优化方案选择
K-Means聚类算法介绍
K-Means又称为K均值聚类算法,属于聚类算法中的一种,而聚类算法在机器学习算法中属于无监督学习,在业务中常常会结合实际需求与业务逻辑理解来完成建模;
无监督学习:训练时只需要特征矩阵X,不需要标签;
K-Means聚类算法基础原理
K-Means聚类算法是聚类算法家族中的典型代表,同时也是最简单的算法,接下来为大家简单地介绍聚类算法基本原理:
将一组存在N个样本的特征矩阵X划分为K个无交集的簇,每一个簇中含有多个数据,每一个数据代表着一个样本,在同一个簇中的数据即被算法认为是同一类;
- N:假设为样本数量;
- K:假设为聚类簇的数量;
- 簇:类似于集合,也可以通俗地理解成一个小组,不同小组等于不同分类;
而一个簇中的所有数据的均值,被称为这个簇的质心,质心的维度与特征矩阵X的维度相同,如特征矩阵X是三维数据集,质心也就是一个三维的坐标,如此类推至更高维度;
K-Means聚类算法实现流程
步骤一:随机在N个样本中抽取K个作为初始的质心;
步骤二:开始遍历除开质心外的所有样本点,将其分配至距离它们最近的质心,每一个质心以及被分配至其下的样本点视为一个簇(或者说一个分类),这样便完成了一次聚类;
步骤三:对于每一个簇,重新计算簇内所有样本点的平均值,取结果为新的质心;
步骤四:比对旧的质心与新的质心是否再发生变化,若发生变化,按照新的质心从步骤二开始重复,若没发生变化,聚类完成;
关键要点:不断地为样本点寻找质心,然后更新质心,直至质心不再变化;
开始做一个简单的聚类
环境说明:本文实际案例中使用Jupyter环境下运行(安装与使用可自行百度);
数据导入
做数据分析前,首先第一步是导入数据,可以利用pandas内的read_csv函数来导入数据;
首先,导入所需要用到的类,并使用read_csv函数导入案例数据:
import numpy as np
import pandas as pd
data = pd.read_csv(r'D:\Machine_learning\KMeans\client_data.csv')
# 使用pandas中的read_csv函数导入数据集后,默认格式为DataFrame
# 直接查看当前数据集长什么样子
data.head()数据打开后会发现大概长这样:
交易额 成交单量 最近交易时间
0 76584.92 294 64
1 94581.00 232 1
2 51037.60 133 1
3 43836.00 98 1
4 88032.00 95 2
# 若表头项为中文时,可能出现乱码情况,请自行百度解决,或直接修改为英文;数据探索
先探索数据类型:
# 探索数据类型
data.info()
# 输出结果:
RangeIndex: 8011 entries, 0 to 8010
Data columns (total 3 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 交易额 8011 non-null float64
1 成交单量 8011 non-null int64
2 最近交易时间 8011 non-null int64
dtypes: float64(1), int64(2)
memory usage: 187.9 KB - 共8011个数据样本,3个维度列(2个整数、1个浮点数),且无缺失数据;
- 数据背景:从三个维度获取所有用户交易180天内交易数据(数据获取、清洗规则在此不作详细说明);
- 第一列:索引(read_csv函数导入数据时会自动生成索引,若数据集本身自带索引,可设置参数index_=0,代表数据集中第一列为索引);
- 第二列:180天内交易额,浮点数;
- 第三列:180天内成交单量,整数;
- 第四列:最近成交订单的日期与当前日期差,整数(180内无数据按照180运算);
由于sklearn中K-Means聚类算法仅支持二维数组运算,所以要先将数据集转化为二维数组:
data = np.array(data,type(float))
# 查看数据集
data
——————————————————————————————————————————————————
# 输出结果:
array([[76584.92, 294.0, 64.0],
[94581.0, 232.0, 1.0],
[51037.6, 133.0, 1.0],
...,
[0.0, 0.0, 180.0],
[0.0, 0.0, 180.0],
[0.0, 0.0, 180.0]], dtype=object)查看数组结构:
# 查看数组结构
data.shape
——————————————————————————————————————————————————
# 输出结果:
(8011, 3)
开始聚类
数据集导入完成后,现在调用sklearn完成简单的聚类:
from sklearn.cluster import KMeans
X = data
# 实例化K-Means算法模型,先使用5个簇尝试聚类
cluster = KMeans(n_clusters=5, random_state=0)
# 使用数据集X进行训练
cluster = cluster.fit(X)
# 调用属性labels_,查看聚类结果
cluster.labels_
——————————————————————————————————————————————————
# 输出结果:
array([4, 4, 1, ..., 0, 0, 0])- 参数n_clusters:
设定聚类的目标簇数量,本次聚类先用5个簇尝试; - 参数random_state:
设定随机数种子,若不设定则每次聚类时都会使用不同的随机质心; - 接口fit():
使用数据集对模型进行训练; - 属性labels_:
查看训练后,每一样本的预测分类结果;
查看输出结果
查看输出结果的数组结构:
# 查看预测结果的数据结构
cluster.labels_.shape
——————————————————————————————————————————————————
# 输出结果:
(8011,)分类结果的数组结构为(8011,),刚好对应着8011个样本的预测分类结果;
再次确认目标分类结果只有5类,可以使用numpy中的unique()函数实现:
# 查看数组中存在的类别(对一维数组去重)
np.unique(cluster.labels_)
————————————————————————————————————————
# 输出结果:
array([0, 1, 2, 3, 4])输出结果0~4中分别代表着5个不同的分类;
查看预测结果中每一分类的数量:
# 查看每一分类结果的数量
pd.value_counts(cluster.labels_)
——————————————————————————————————————————
0 7068
2 688
4 198
1 38
3 19
dtype: int64分类为0的数据占比较大(约88%),这部分数据数据实际行业应用中的长尾数据,这类用户对平台几乎没有任何价值贡献;
聚类质心
聚类质心代表每一个分类簇的中心,某种意义上讲,质心坐标可以代表着这一个簇的普遍特征,质心可以通过调用属性cluster_centers_来查看:
# 查看质心
cluster.cluster_centers_
——————————————————————————————————————————————————————————
# 输出结果:
array([[3.40713759e+02, 7.43350311e-01, 1.48025750e+02],
[4.30125087e+04, 4.70000000e+01, 2.03947368e+01],
[6.06497324e+03, 9.37354651e+00, 3.55159884e+01],
[7.57037853e+04, 7.84736842e+01, 1.52631579e+01],
[1.80933537e+04, 2.34040404e+01, 1.49444444e+01]])输出结果中分别对应着0~4五种分类的普遍数据特征;
K-Means聚类算法的评估指标
当我们完成聚类建模后,怎么知道聚类的效果好不好,这时我们便需要「评估指标」来评价模型的优劣,并根据此来调整参数;
对于聚类算法的评估指标,从大方向上区分为两种:真实标签已知与真实标签未知;
真实标签已知
即我们对于每一个样本的标签Y都是已知的,但是这种情况在实际的业务中几乎是不存在的,若标签已知,使用分类算法(如随机森林、SVM等)在各个方面来说都会更加合适;
- 调整兰德系数:
在sklearn中的类为sklearn.metrics.adjusted_rand_score(y_true, y_pred)
y_true:代表测试集中一个样本的真实标签;
y_pred:使用测试集中样本调用预测接口的预测结果(上文中使用的cluster.labels_);
调整兰德系数的取值在[-1,1]:数值越接近1越好,大于0时聚类效果较为优秀,小于0时代表簇内差异巨大甚至相互独立,模型几乎不可用;
由于案例数据集中真实标签是未知的,故不在此展示;
真实标签未知
即我们对每一个样本的标签Y都是未知的,我们事先不知道每一个样本是属于什么分类,这种情况才是符合我们实际业务中真实使用聚类算法的场景;
- 轮廓系数系数:
在sklearn中的类为:
返回轮廓系数的均值:sklearn.metrics.silhouette_score(X, y_pred);
返回数据集中每个样本自身的轮廓系数:sklearn.metrics.silhouette_sample(X, y_pred);
轮廓系数的取值在(-1,1):
对于某一样本点来说,当值越接近1时就代表自身与所在的簇中其他样本越相似,并且与其他簇中的样本不相似,而当值越接近-1时则代表与上述内容相反;综述,轮廓系数越接近1越好,负数则表示聚类效果非常差;
那接下来看看轮廓系数在刚才的聚类中效果如何:
# 导入轮廓系数所需要的库
from sklearn.metrics import silhouette_score
from sklearn.metrics import silhouette_samples
# 查看轮廓系数均值
silhouette_score(X,cluster.labels_)
——————————————————————————————————————————————————
# 输出结果
0.8398497410297728
——————————————————————————————————————————————————
# 查看每一样本轮廓系数
silhouette_samples(X,cluster.labels_)
——————————————————————————————————————————————————
# 输出结果
array([0.94301872, 0.94301872, 0.94301872, ..., 0.64706719, 0.60820687,
0.58272791])
——————————————————————————————————————————————————
# 查看样本轮廓系数结果的数组结构
silhouette_samples(X,cluster.labels_).shape
——————————————————————————————————————————————————
# 输出结果
(8011,)本次聚类的轮廓系数为0.84,表示聚类效果良好;
样本轮廓系数的数据结构可以看出:数组中每一个输出结果对应着每一个样本的轮廓系数,共8011个;
- 卡林斯基-哈拉巴斯指数:
sklearn中的类:sklearn.metrics.calinski_haabasz_score (X, y_pred);
卡林斯基-哈拉巴斯指数的数值无上限,且对于模型效果来说越高越好,而由于无上限的特性,导致只能用作对比,而无法快速知晓模型效果是否好;
可以看看轮廓系数在刚才的聚类中效果如何:
# 调用所需要的类
from sklearn.metrics import calinski_harabasz_score
calinski_harabasz_score(X,cluster.labels_)
———————————————————————————————————————————————————————
# 输出结果
31777.971149699857输出的结果为31778,那究竟效果如何?因为没有对照组,所以无法得知,如果有兴趣的小伙伴可以在调整参数的时候使用对照组试试效果;
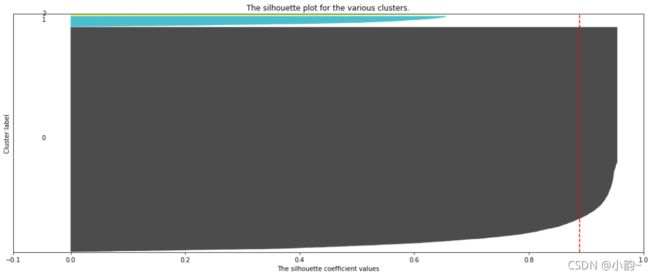
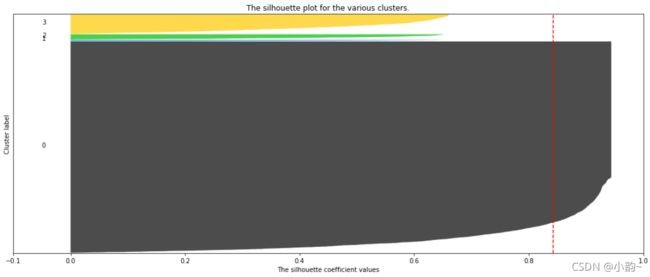
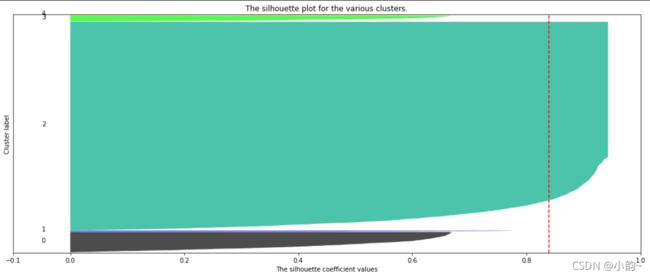
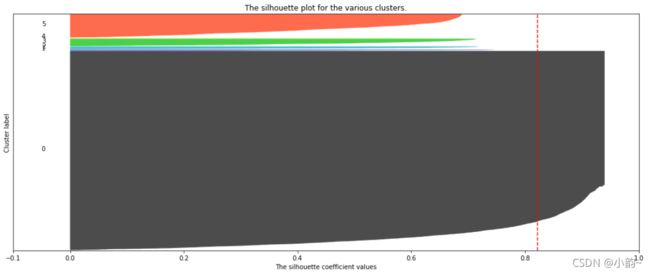
实用案例:基于轮廓系数来选择最佳的n_clusters
需要绘制轮廓系数分布图,先导入所需用到的库:
import matplotlib.pyplot as plt
import matplotlib.cm as cm绘制轮廓系数分布图
使用for循环分别对2~8个簇的情况画出轮廓系数分布图:
for n_clusters in [2,3,4,5,6,7,8]:
n_clusters = n_clusters
# 设置画布
fig, ax1 = plt.subplots(1)
# 设置画布尺寸
fig.set_size_inches(18, 7)
# 设置画布X轴
ax1.set_xlim([-0.1, 1])
# 设置画布Y轴:X.shape[0]代表着柱状的宽度,(n_clusters + 1) * 10代表着柱与柱之间的间隔
ax1.set_ylim([0, X.shape[0] + (n_clusters + 1) * 10])
# 模型实例化
clusterer = KMeans(n_clusters=n_clusters, random_state=100)
# 开始训练模型
clusterer = clusterer.fit(X)
# 提取训练结果中的预测标签
cluster_labels = clusterer.labels_
# 提取训练结果中的轮廓系数均值
silhouette_avg = silhouette_score(X, cluster_labels)
# 打印出当前的簇数与轮廓系数均值
print("簇数为", n_clusters,
",轮廓系数均值为", silhouette_avg)
# 提取每一个样本的轮廓系数
sample_silhouette_values = silhouette_samples(X, cluster_labels)
# 设置Y轴的起始坐标
y_lower = 10
# 添加一个循环,把每一个样本的轮廓系数画在图中
for i in range(n_clusters):
# 提取第i个簇下的所有样本轮廓系数
ith_cluster_silhouette_values = sample_silhouette_values[cluster_labels == i]
# 对样本的轮廓系数进行排序(降序)
ith_cluster_silhouette_values.sort()
# 设置当前簇的柱状宽度(使用样本数量)以便于设置下一个簇的起始坐标
size_cluster_i = ith_cluster_silhouette_values.shape[0]
# 设置Y轴第i个簇的起始坐标
y_upper = y_lower + size_cluster_i
# 设置颜色
color = cm.nipy_spectral(float(i)/n_clusters)
# 画图
ax1.fill_betweenx(np.arange(y_lower, y_upper)
,ith_cluster_silhouette_values
,facecolor=color
,alpha=0.7
)
ax1.text(-0.05
, y_lower + 0.5 * size_cluster_i
, str(i))
y_lower = y_upper + 10
# 设置图的标题
ax1.set_title("The silhouette plot for the various clusters.")
ax1.set_xlabel("The silhouette coefficient values")
ax1.set_ylabel("Cluster label")
# 添加轮廓系数均值线,使用虚线
ax1.axvline(x=silhouette_avg, color="red", linestyle="--")
ax1.set_yticks([])
ax1.set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
plt.show()结果对比
输出结果:
从本次的输出结果中可知,当簇数量为2时,会存在最大的轮廓系数均值,是否簇数量为2就是最佳的参数呢?
答案必须是否定的,我们可以通过轮廓系数分部图看到,基本上每一个图内都会有一片面积很大的块,这就是长尾数据带来的,因为他们基本都集中在一个点上,所以导致整体轮廓系数均值“被平均”得很大,这样的状况也是很多实际业务数据中常常会碰到的;
优化方案选择
既然由于长尾数据对轮廓系数带来较大偏差,那咱们的思路可以把长尾数据剔除掉,仅计算非长尾数据(数据分析需要在不同的具体场景下有不同的思路,以下仅是一种思路举例);
- 当簇数量为3时:
# 实例化,训练模型
n_clusters = 3
clusterer = KMeans(n_clusters=n_clusters, random_state=100)
clusterer = clusterer.fit(X)
# 查看训练结果
pd.value_counts(clusterer.labels_)
————————————————————————————————————————————————————————————
# 输出结果
0 7599
1 362
2 50
dtype: int64长尾数据所在的簇为0,计算非长尾数据的轮廓系数均值:
cluster_labels = clusterer.labels_
print(np.average(silhouette_samples(X, cluster_labels)[cluster_labels != 0]))
——————————————————————————————————————————————————————————————————————————————
# 输出结果
0.4909204497858037- 当簇数量为4时:
# 实例化,训练,并查看结果分布
n_clusters = 4
clusterer = KMeans(n_clusters=n_clusters, random_state=100)
clusterer = clusterer.fit(X)
pd.value_counts(clusterer.labels_)
————————————————————————————————————————————————————————————
# 输出结果
0 7125
3 663
2 179
1 44
dtype: int64
————————————————————————————————————————————————————————————
# 计算非长尾数据的轮廓系数均值
cluster_labels = clusterer.labels_
print(np.average(silhouette_samples(X, cluster_labels)[cluster_labels != 0]))
————————————————————————————————————————————————————————————
# 输出结果
0.4766824917258095- 当簇数量为5时:
# 实例化,训练,并查看结果分布
n_clusters = 5
clusterer = KMeans(n_clusters=n_clusters, random_state=100)
clusterer = clusterer.fit(X)
pd.value_counts(clusterer.labels_)
————————————————————————————————————————————————————————————
# 输出结果
2 7065
0 691
3 198
1 38
4 19
dtype: int64
————————————————————————————————————————————————————————————
# 计算非长尾数据的轮廓系数均值
cluster_labels = clusterer.labels_
print(np.average(silhouette_samples(X, cluster_labels)[cluster_labels != 2]))
————————————————————————————————————————————————————————————
# 输出结果
0.49228555254491085- 当簇数量为6时:
# 实例化,训练,并查看结果分布
n_clusters = 6
clusterer = KMeans(n_clusters=n_clusters, random_state=100)
clusterer = clusterer.fit(X)
pd.value_counts(clusterer.labels_)
————————————————————————————————————————————————————————————
# 输出结果
0 6806
5 799
3 252
2 99
1 36
4 19
dtype: int64
————————————————————————————————————————————————————————————
# 计算非长尾数据的轮廓系数均值
cluster_labels = clusterer.labels_
print(np.average(silhouette_samples(X, cluster_labels)[cluster_labels != 0]))
————————————————————————————————————————————————————————————
# 输出结果
0.5043196493336838- 当簇数量为7时:
# 实例化,训练,并查看结果分布
n_clusters = 7
clusterer = KMeans(n_clusters=n_clusters, random_state=100)
clusterer = clusterer.fit(X)
pd.value_counts(clusterer.labels_)
————————————————————————————————————————————————————————————
# 输出结果
0 6374
5 931
6 387
2 188
1 76
4 36
3 19
dtype: int64
————————————————————————————————————————————————————————————
# 计算非长尾数据的轮廓系数均值
cluster_labels = clusterer.labels_
print(np.average(silhouette_samples(X, cluster_labels)[cluster_labels != 0]))
————————————————————————————————————————————————————————————
# 输出结果
0.501667625921486- 当簇数量为8时:
# 实例化,训练,并查看结果分布
n_clusters = 8
clusterer = KMeans(n_clusters=n_clusters, random_state=100)
clusterer = clusterer.fit(X)
pd.value_counts(clusterer.labels_)
————————————————————————————————————————————————————————————
# 输出结果
0 6411
5 927
4 372
2 172
6 74
1 32
7 13
3 10
dtype: int64
————————————————————————————————————————————————————————————
# 计算非长尾数据的轮廓系数均值
cluster_labels = clusterer.labels_
print(np.average(silhouette_samples(X, cluster_labels)[cluster_labels != 0]))
————————————————————————————————————————————————————————————
# 输出结果
0.4974116370311323对比上述结果,当n_clusters=6时,轮廓系数均值存在最大值0.5043;
这时查看质心的坐标:
# 设置参数n_clusters=6再次训练模型
n_clusters = 6
clusterer = KMeans(n_clusters=n_clusters, random_state=100)
clusterer = clusterer.fit(X)
# 使用属性cluster_centers_查看质心坐标
clusterer.cluster_centers_
——————————————————————————————————————————————————————————————
# 输出结果
array([[2.50559675e+02, 5.95944755e-01, 1.51620923e+02],
[4.36372269e+04, 4.83333333e+01, 2.06111111e+01],
[2.23257222e+04, 2.73232323e+01, 1.72020202e+01],
[1.15493973e+04, 1.65515873e+01, 1.95833333e+01],
[7.57037853e+04, 7.84736842e+01, 1.52631579e+01],
[4.25642288e+03, 6.82227785e+00, 4.39336671e+01]])
——————————————————————————————————————————————————————————————
# 查看聚类结果分布
pd.value_counts(clusterer.labels_)
——————————————————————————————————————————————————————————————
# 输出结果
0 6806
5 799
3 252
2 99
1 36
4 19
dtype: int64
——————————————————————————————————————————————————————————————
# 聚类结果分布以百分比形式显示
pd.value_counts(clusterer.labels_,normalize=True)
——————————————————————————————————————————————————————————————
# 输出结果
0 0.849582
5 0.099738
3 0.031457
2 0.012358
1 0.004494
4 0.002372
dtype: float64从结果可得(数据结果为科学计数法),6个类别客户的画像特征分别对应着:
- 分类0——6806位——占比85%:
交易额:251元,平均单量:0.6单,最近交易时间:152天前; - 分类1——36位——占比0.4%:
交易额:43637元,平均单量:48单,最近交易时间:21天前; - 分类2——99位——占比1.2%:
交易额:22325元,平均单量:27单,最近交易时间:17天前; - 分类3——252位——占比3.1%:
交易额:11549元,平均单量:17单,最近交易时间:20天前; - 分类4——19位——占比0.2%:
交易额:75703元,平均单量:78单,最近交易时间:15天前; - 分类5——799位——占比10%:
交易额:4256元,平均单量:7单,最近交易时间:44天前;