16S多样性ASV分析-低氧区ODZ水层分析
第一节. 质控,实验设计,双端序列合并(在Qiime1中进行)
此部分内容来自
https://blog.csdn.net/woodcorpse/article/details/76903998
一、初步看下测序质量
首先,安装fastqc
先把原始数据做fastqc, 结果放进multiQC,看原始数据质量如何,有没有adapter
#首先安装好fastqc、运行
# 下载fastqc http://www.bioinformatics.babraham.ac.uk/projects/download.html#fastqc
wget http://www.bioinformatics.babraham.ac.uk/projects/fastqc/fastqc_v0.11.5.zip
# 解压fastqc
unzip fastqc_v0.11.5.zip
# 赋与fastqc可执行权限
chmod 755 FastQC/fastqc
# 运行
~/FastQC/fastqc -t 2 *.fq.gz
新建一个文件夹存放这些结果
#新建名为fastqcout的文件夹
mkdir fastqcout
#将所有此步产生的文件移入fastqcoutq文件夹
mv *fastqc.zip fastqcout
mv *fastqc.html fastqcout
下一步进行multiqc,将所有的fastqc结果合成到一个网页之中
#进入fastqc文件夹
cd fastq
#在当前文件夹中将所有文件进行multiqc
multiqc .
关于multiqc产生的结果如何评估,可以参见
https://www.jianshu.com/p/f83626fd1fa1
https://www.jianshu.com/p/85da4dcc6020
也可以单独看每一个fastqc的质控结果,详细参见
https://www.jianshu.com/p/85da4dcc6020
二、import data
接下来所有的的流程里要用到两个文件:①manifest(csv格式或者txt)文件;②sample-metadata(tsv格式)文件
输入格式为SequencesWithQuality(一般fastq格式都是SampleData[SequencesWithQuality])
首先在工作目录中,建立两个文件夹:sample_data和results。将刚才的两个文件分别放入两个文件夹:manifest→sample_data;sample-metadata→result
manifest中的第二列:absolute-filepath这一列要注意, P W D 是 目 前 绝 对 路 径 , 文 件 的 绝 对 路 径 需 要 写 为 PWD是目前绝对路径,文件的绝对路径需要写为 PWD是目前绝对路径,文件的绝对路径需要写为PWD/sample_data/xxxxxxxx.fq,qz。不然会报错
保证你在进行下一步的时候是在你的工作文件夹中(也就是包含sample_data和results两个文件夹的那个文件夹)。
在准备好文件夹、manifest等原料以后,可以进行数据的输入:
qiime tools import \
--type 'SampleData[PairedEndSequencesWithQuality]' \
--input-path sample_data/pe-33-manifest.csv \
--output-path results/paired-end-demux.qza \
--input-format PairedEndFastqManifestPhred33
这一步成功后,系统会显示:
Imported sample_data/pe-33-manifest.csv as PairedEndFastqManifestPhred33 to results/paired-end-demux.qza
此时生成的文件类型是qza格式,要想进行qza的可视化,需要将它转为qzv格式,输入命令行:
qiime demux summarize \
--i-data results/paired-end-demux.qza \
--o-visualization results/demux.qzv
查看.qzv结果
使用 https://view.qiime2.org 网址显示结果(把文件拖动到这个网页里)
三、去除非生物序列(去引物)
使用插件:q2-cutadapt
我的数据中包含引物(引物、测序接头、PCR间隔区等都是非生物序列),并且我的去噪过程选择的是deblur(不像dada2可以直接设置参数对序列进行trim),所以应该在后续操作之前首先删除这些序列。
但是如果正反向引物长度相同,可以在deblur那一步选择trim参数对前面固定长度进行截断。
#Removing non-biological sequences
qiime cutadapt trim-paired \
--i-demultiplexed-sequences paired-end-demux.qza \
--p-front-f ACTCCTACGGGAGGCAGCA \
--p-front-r GGACTACHVGGGTWTCTAAT \
--o-trimmed-sequences paired-end-trimmed-seqs.qza
输出文件:
去掉引物的双端序列:paired-end-trimmed-seqs.qza
#对去引物的双端序列进行可视化
qiime demux summarize \
--i-data paired-end-trimmed-seqs.qza \
--o-visualization paired-end-trimmed-seqs.qzv
输出文件:
去引物的双端序列可视化统计结果:paired-end-trimmed-seqs.qza
四、序列双端合并join-paired
在双端合并之前,先要对单端的序列进行质控,用vsearch join-pairs命令(allow 3 mismatches for at least 10 bp overlap)
time qiime vsearch join-pairs \
--i-demultiplexed-seqs paired-end-trimmed-seqs.qza \
--p-truncqual 20 \
--p-minovlen 10 \
--p-maxdiffs 3 \
--o-joined-sequences demux-joined.qza
这一步输出的文件有:demux-joined.qza
五、查看合并序列的数据质量和摘要Viewing a summary of joined data with read quality
#获得拼接数据的可视化结果
qiime demux summarize \
--i-data demux-joined.qza \
--o-visualization demux-joined.qzv
输出可视化对象:
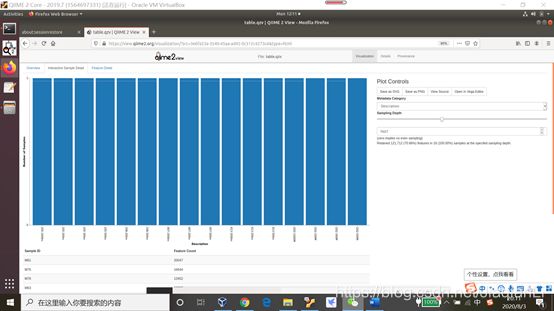
demux-joined.qzv(看一下这个文件里的mean或者total sequences number)
这份摘要报告对于确定成功合并序列大约有多长特别有用**(当我们用deblur去噪时,我们会回到这个问题上)**。
在这个可视化中查看质量图时,如果您将鼠标悬停在一个特定的位置上,将看到有多少个序列至少有那么长(为计算序列质量而采样的序列数量统计)。记下最高的序列位置,其中大部分(比如,>99%)的序列至少有那么长。
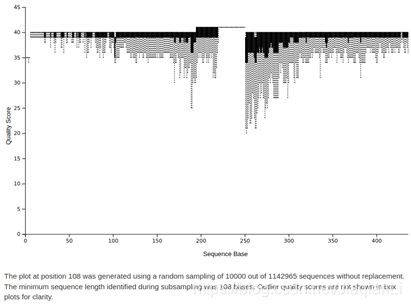
例如,当将鼠标悬停在可视化箱线图中的一个黑箱体上时(该黑箱体是由比本教程中使用的数据集更大的数据集生成的),可以看到1142965个序列中有10000个用于估计该位置的质量分数分布。
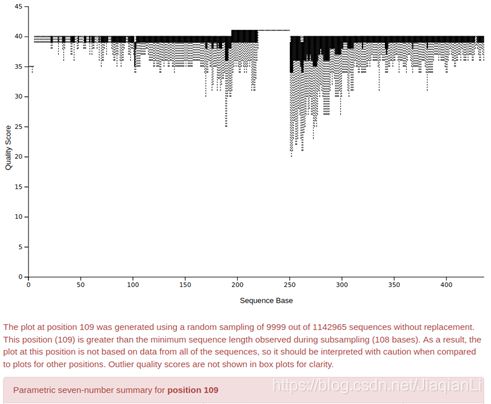
此处如果鼠标悬停的位置显示红色方框,说明一些序列没有这么长,例如在悬停在109这个位置时,只有小于9999个序列用于估计该位置的质量分数分布。
基于对这些图的比较,将注意到我的大多数序列至少有108个碱基长。
六、序列质控
接下来将使用质量过滤器quality-filter q-score-joined对序列进行质量控制。
此方法与质量过滤 quality-filter q-score 相同,只是它仅对合并的序列进行操作。此方法的参数尚未在双端合并的数据上进行广泛的基准测试,因此我们建议尝试使用不同的参数设置。
注意,此处的–p-min-quality参数,和合并序列时的truncqual参数任选一个设置即可,具体看哪种方式可以保留更多的序列。
qiime quality-filter q-score-joined \
--i-demux demux-joined.qza \
--p-min-quality 20 \
--o-filtered-sequences demux-joined-filtered.qza \
--o-filter-stats demux-joined-filter-stats.qza
输出对象:
统计结果:demux-joined-filter-stats.qza。
数据过滤后结果:demux-joined-filtered.qza。
#使用metadata tabulate命令对统计结果进行可视化
qiime metadata tabulate \
--m-input-file demux-joined-filter-stats.qza \
--o-visualization demux-joined-filter-stats.qzv
输出结果:
可视化的统计结果:demux-joined-filter-stats.qzv
在这个阶段,我选择继续使用Deblur进行额外的质控(或者您也可以进行序列去冗余,并选择使用q2-vsearch将它们聚类到OTU中,但此处我选择ASV,所以没有给聚类到OTU)
Deblur
time qiime deblur denoise-16S \
--i-demultiplexed-seqs demux-joined-filtered.qza \
--p-trim-length 400 \
--p-sample-stats \
--p-jobs-to-start 20 \
--o-representative-sequences rep-seqs.qza \
--o-table table.qza \
--o-stats deblur-stats.qza
#将此步产生的统计过程可视化
qiime deblur visualize-stats\
--i-deblur-stats deblur-stats.qza \
--o-visualization deblur-stats.qzv
输出结果:
代表序列:rep-seqs.qza。
统计过程:deblur-stats.qza。
特征表:table.qza。
可视化统计过程:deblur-stats.qzv
这一步output为feature table,代表序列及每个sample的summary。这里feature table类似于之前我们用uparse算法聚类(把有97%相似性的序列聚集在一起成一个OTU,达到去重和聚类的目的)出OTU table类似,但是不论是DADA2还是Deblur, 都可以理解为用100%相似程度来聚类“OTU”,又称为sequence variants,或者sOTU,在qiime2的文档中,用ASV进行统一这些各种的叫法。
为什么用ASV?
1、它比用97%相似程度聚类的“OTU”更加精准,在后续的alpha diversity及物种注释中会更加准确,因为传统意义上的OTU的97%的相似程度是在genus水平上大家认可的,但如果需要到species甚至strain水平的话,使用传统的OTU是很不靠谱的。
2、ASV相比于OTU最大的好处就是可以随时合并不同的数据跑出来的代表序列。因为使用的是100%的identidy,所以一样的序列就是一样的物种,不会受到不同数据、测序平台、文库建立、处理方法等等的误差影响。只要是qiime2上跑出来的数据,即使是不同时间不同人的结果,也可以直接合并,甚至不需要接触到原始的测序数据,也大大方便了大数据之间的整合。所以现在也越来越多的人倡导使用ASV以避免数据不可合并和难以合并的缺点。
作者:GPZ_Lab 链接:https://www.jianshu.com/p/78b5f27d97e2
查看Deblur特征表
总结q2-deblur生成的功能表,这个表和相应的代表序列现在可以用同样的方法和可视化工具来分析,这些方法和可视化工具将用于单端序列数据。
qiime feature-table summarize \
--i-table table.qza \
--o-visualization table.qzv \
--m-sample-metadata-file sample-metadata.tsv
输出对象:
可视化特征表:table.qzv。
样品稀释(抽平)
要比较物种多样性就需要让样品们的采样深度统一在一个水平上,这一步就需要用到稀释,选择的采样深度太低会导致有些样品被丢掉,选择的测序深度太高会导致低于该测序深度的ASV被丢弃,
qiime feature-table rarefy \
--i-table table.qza \
--p-sampling-depth 4511 \
--o-rarefied-table table-rarefied.qza
输出结果:
稀释好的ASV table:table-rarefied.qza。
七、构建进化树用于多样性分析
QIIME 2支持几种系统发育多样性度量方法,包括Faith’s Phylogenetic
Diversity、weighted和unweighted
UniFrac。除了每个样本的特征计数(即QIIME2对象FeatureTable[Frequency])之外,这些度量还需要将特征彼此关联结合有根进化树。此信息将存储在一个QIIME2对象的有根系统发育对象Phylogeny[Rooted]中。为了生成系统发育树,我们将使用q2-phylogeny插件中的align-to-tree-mafft-fasttree工作流程。首先,工作流程使用mafft程序执行对FeatureData[Sequence]中的序列进行多序列比对,以创建QIIME2对象FeatureData[AlignedSequence]。接下来,流程屏蔽(mask或过滤)对齐的的高度可变区(高变区),这些位置通常被认为会增加系统发育树的噪声。随后,流程应用FastTree基于过滤后的比对结果生成系统发育树。FastTree程序创建的是一个无根树,因此在本节的最后一步中,应用根中点法将树的根放置在无根树中最长端到端距离的中点,从而形成有根树。
http://blog.sciencenet.cn/blog-3334560-1229017.html
rep-seqs.qzv其实就是每个sOTU的序列内容,点击其序列连接可以直接blast,十分方便。rooted tree在后续的alpha diversity中可能会用到(如果需要计算PD value)。
链接:https://www.jianshu.com/p/78b5f27d97e2
time qiime phylogeny align-to-tree-mafft-fasttree \
--i-sequences rep-seqs.qza \
--o-alignment aligned-rep-seqs.qza \
--o-masked-alignment masked-aligned-rep-seqs.qza \
--o-tree unrooted-tree.qza \
--o-rooted-tree rooted-tree.qza
输出结果文件:
多序列比对结果:aligned-rep-seqs.qza
过滤去除高变区后的多序列比对结果:masked-aligned-rep-seqs.qza
有根树,用于多样性分析:rooted-tree.qza
无根树:unrooted-tree.qza
八、Alpha和beta多样性分析
Alpha and beta diversity analysis
QIIME 2的多样性分析使用q2-diversity插件,该插件支持计算α和β多样性指数、并应用相关的统计检验以及生成交互式可视化图表。我们将首先应用core-metrics-phylogenetic方法,该方法将FeatureTableFrequency抽平到用户指定的测序深度,然后计算几种常用的α和β多样性指数,并使用Emperor为每个β多样性指数生成主坐标分析(PCoA)图。默认情况下计算的方法有:
一、α多样性 :
①香农(Shannon’s)多样性指数:群落丰富度的定量度量,即包括丰富度richness和均匀度evenness两个层面)
②可观测的OTU (Observed OTUs,群落丰富度的定性度量,只包括丰富度)
③Faith’s系统发育多样性(包含特征之间的系统发育关系的群落丰富度的定性度量)
④均匀度Evenness(或 Pielou’s均匀度;群落均匀度的度量)
二、β多样性
①Jaccard距离(群落差异的定性度量,即只考虑种类,不考虑丰度)
②Bray-Curtis距离(群落差异的定量度量,较常用)
③非加权UniFrac距离(包含特征之间的系统发育关系的群落差异定性度量)
④加权UniFrac距离(包含特征之间的系统发育关系的群落差异定量度量)需要提供给这个脚本的一个重要参数是–p-sampling-depth,它是指定重采样(即稀疏/稀疏rarefaction)深度。因为大多数多样指数对不同样本的不同测序深度敏感,所以这个脚本将随机地将每个样本的测序量重新采样至该参数值。例如,提供–p-sampling-depth 500,则此步骤将对每个样本中的计数进行无放回抽样,从而使得结果表中的每个样本的总计数为500。如果任何样本的总计数小于该值,那么这些样本将从多样性分析中删除。选择这个值很棘手。我们建议你通过查看上面创建的表table.qzv文件中呈现的信息并选择一个尽可能高的值(因此每个样本保留更多的序列)同时尽可能少地排除样本来进行选择。
http://blog.sciencenet.cn/blog-3334560-1229017.html
#计算核心多样性
qiime diversity core-metrics-phylogenetic \
--i-phylogeny rooted-tree.qza \
--i-table table.qza \
--p-sampling-depth 4511 \
--m-metadata-file sample-metadata.tsv \
--output-dir core-metrics-results
输出结果:有13个数据结果和
- core-metrics-results/faith_pd_vector.qza: Alpha多样性考虑进化的faith指数
- core-metrics-results/unweighted_unifrac_distance_matrix.qza: 无权重unifrac距离矩阵
- core-metrics-results/bray_curtis_pcoa_results.qza: 基于Bray-Curtis距离PCoA的结果。
- core-metrics-results/shannon_vector.qza: Alpha多样性香农指数
- core-metrics-results/rarefied_table.qza: 等量重采样后的特征表
- core-metrics-results/weighted_unifrac_distance_matrix.qza: 有权重的unifrac距离矩阵
- core-metrics-results/jaccard_pcoa_results.qza: jaccard距离PCoA结果
- core-metrics-results/observed_otus_vector.qza: Alpha多样性observed otus指数
- core-metrics-results/weighted_unifrac_pcoa_results.qza:基于有权重的unifrac距离的PCoA结果
- core-metrics-results/jaccard_distance_matrix.qza: jaccard距离矩阵
- core-metrics-results/evenness_vector.qza: Alpha多样性均匀度指数
- core-metrics-results/bray_curtis_distance_matrix.qza: Bray-Curtis距离矩阵
- core-metrics-results/unweighted_unifrac_pcoa_results.qza: 无权重的unifrac距离的PCoA结果
以及4个可视化结果:
- core-metrics-results/unweighted_unifrac_emperor.qzv:无权重的unifrac距离PCoA结果采用emperor可视化
- core-metrics-results/jaccard_emperor.qzv:jaccard距离PCoA结果采用emperor可视化
- core-metrics-results/bray_curtis_emperor.qzv: Bray-Curtis距离PCoA结果采用emperor可视化
- core-metrics-results/weighted_unifrac_emperor.qzv: 有权重的unifrac距离PCoA结果采用emperor可视化
在计算多样性度量之后,可以开始基于样本元数据的分组信息或属性值背景探索样本的微生物组成差异。
首先测试分类元数据列和alpha多样性数据之间的关系。接下来为Faith系统发育多样性(群体丰富度的度量)和Evenness均匀度进行可视化操作。
1.Alpha多样性组间显著性分析和可视化
#Faith系统发育多样性可视化
qiime diversity alpha-group-significance \
--i-alpha-diversity core-metrics-results/faith_pd_vector.qza \
--m-metadata-file sample-metadata.tsv \
--o-visualization core-metrics-results/faith-pd-group-significance.qzv
#Evenness 均匀度可视化
qiime diversity alpha-group-significance \
--i-alpha-diversity core-metrics-results/evenness_vector.qza \
--m-metadata-file sample-metadata.tsv \
--o-visualization core-metrics-results/evenness-group-significance.qzv
输出可视化结果:
1.core-metrics-results/faith-pd-group-significance.qzv
2.core-metrics-results/evenness-group-significance.qzv
从上面两个可视化结果可以看出哪些分类样本元数据列与微生物群落丰富度的差异密切相关,以及这些差异在统计学是否具有有显著性。图中可按Column选择分类方法,查看不同分组下箱线图间的分布与差别。图形下面的表格,详细详述了组间比较的显著性和假阳性率统计。
2.beta多样性
我这一步当中,每一个站位都是unique的,所以无法进行下述分析。如果想进行下述分析,需要把samples按照想要研究的目的分组。(例如1xx断面、2xx断面等,一个组里需要包含几个不同的samples),所以下面的方法可供下次有目的性的研究参考。
接下来,使用PERMANOVA方法beta-group-significance分析分类型元数据的样本组间差异。以下命令将测试一组样本之间的距离,是否比来自其他组的样本彼此更相似。
如果用这个命令的–p-pairwise参数,它将执行成对检验,结果将允许我们确定哪对特定组彼此不同是否显著不同。这个命令运行起来可能很慢,尤其是当使用–p-pairwise参数,因为它是基于置换检验的。因此,我们将在元数据的特定列上运行该命令(当然,在本研究中只有一个按照站位分的特定列),而不是在其适用的所有元数据列上运行该命令。
这里,我们将使用description元数据列将此应用到未加权的UniFrac距离,如下所示。
time qiime diversity beta-group-significance \
--i-distance-matrix core-metrics-results/unweighted_unifrac_distance_matrix.qza \
--m-metadata-file sample-metadata.tsv \
--m-metadata-column Description \
--o-visualization core-metrics-results/unweighted-unifrac-Description-significance.qzv \
--p-pairwise
输出可视化结果:
core-metrics-results/unweighted-unifrac-Description-significance.qzv
该部分的结果可以用于观察不同站位组内和组间差异显著性分析,采用箱线图+统计表呈现
同样,我们对于这个数据集所拥有的连续样本元数据中没有一个与样本组成相关,因此这里我们不会测试这些关联。如果对执行这些测试感兴趣,那么可以使用qiime metadata distance-matrix结合qiime diversity mantel和qiime diversity bioenv命令组合使用。
3.Emperor研究PCoA
最后,在样本元数据分组间探索微生物群落组成差异的流行方法是排序。可以使用Emperor工具在元数据下探索主坐标分析(PCoA)绘图。虽然我们的core-metrics-phylogenetic命令已经生成了一些Emperor图,但我们希望传递一个可选的参数–p-custom-axes,这对于探索时间序列数据非常有用。
采于core-metrics-phylogeny的PCoA结果也是一样的,这使得很容易与Emperor生成新的可视化。
采用未加权的UniFrac和Bray-Curtis的PCoA结果生成Emperor图,以便所得到的图将包含主坐标1、主坐标2和实验开始以来的天数(days since the experiment start)的轴,我们将使用最后一个轴来探索这些样本是如何随时间变化的(有时间序列的时候会有这个轴,但是本研究中没有时间序列,所以没有第三个轴,如果有关于时间序列的研究,可以参考) 。
#三维分析图,但由于没有时间序列,故删去第三个参数,生成二维图
qiime emperor plot \
--i-pcoa core-metrics-results/bray_curtis_pcoa_results.qza \
--m-metadata-file sample-metadata.tsv \
--p-custom-axes Description \
--o-visualization core-metrics-results/bray-curtis-emperor-Description.qzv
qiime emperor plot \
--i-pcoa core-metrics-results/unweighted_unifrac_pcoa_results.qza \
--m-metadata-file sample-metadata.tsv \
--p-custom-axes Description \
--o-visualization core-metrics-results/unweighted-unifrac-emperor-Description.qzv
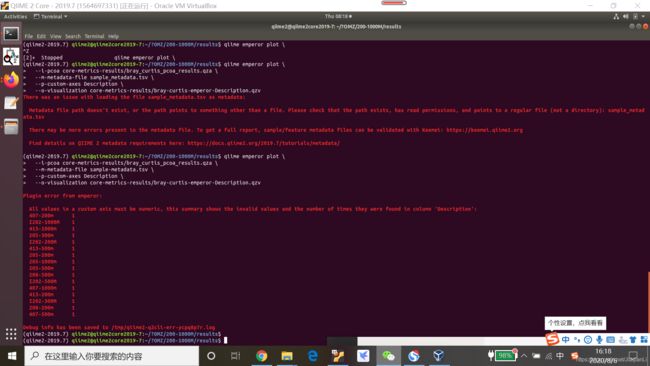 此处跟beta diversity一样,一个分组内必须由多个samples组成,才能进行emperor的排序分析,本研究的报错是由于,在description这个column下,每一个样本都是独特的,所以建议sample-metadata的样本元数据多设置几个column,便于组建的横向比较。
此处跟beta diversity一样,一个分组内必须由多个samples组成,才能进行emperor的排序分析,本研究的报错是由于,在description这个column下,每一个样本都是独特的,所以建议sample-metadata的样本元数据多设置几个column,便于组建的横向比较。
此处应该输出的可视化结果为基于不同距离的PCoA图,每一个点代表一个样本,相同颜色的点来自同一个分组,两点之间距离越近表明两者的群落构成差异越小。
PCA与PCoA的区别:
PCA(Principalcomponent analysis)主成分分析是一种研究数据相似性或差异性的可视化方法,采取降维的思想。PCA 可以找到距离矩阵中最主要的坐标,把复杂的数据用一系列的特征值和特征向量进行排序后,选择主要的前几位特征值,来表示样品之间的关系。PC后面的百分数表示对应特征向量对数据的解释量,此值越大越好;
PCoA(Principal Co-ordinates Analysis)主坐标分析,与PCA类似,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,它没有改变样本点之间的相互位置关系,只是改变了坐标系统。
在微生物分析中我们会基于beta多样性分析得到的距离矩阵,进行PCA和PCoA分析。PCA是基于样本的相似矩阵(如欧式距离)来寻找主成分,而PCoA是基于相异距离矩阵(欧式距离以外的其他距离,包括binary_jaccard ,bray_curtis ,unweighted_unifrac和weighted_unifrac距离)来寻找主坐标。
因此,如果样本数目比较多,而物种数目比较少,那肯定首选PCA;如果样本数目比较少,而物种数目比较多,那肯定首选PCoA。
九、Alpha稀疏曲线
稀疏曲线一般在微生物组研究中用于评估测序量或样本量的饱和情况。接下来,我们使用qiime diversity alpha-rarefaction可视化工具来探索α多样性与采样深度的关系。
该可视化工具在多个采样深度处计算一个或多个α多样性指数,范围介于1(可选地–p-min-depth控制)和最大采样深度–p-max-depth提供值之间,最大采样深度必须大于min_depth。在每个采样深度,将生成10个抽样表,并对表中的所有样本计算alpha多样性指数计算。迭代次数(在每个采样深度计算的稀疏表)可以通过–p-iterations来控制。在每个采样深度,将为每个样本绘制平均多样性值,如果提供样本元数据–m-metadata-file参数,则可以基于元数据对样本进行分组。
time qiime diversity alpha-rarefaction \
--i-table table.qza \
--i-phylogeny rooted-tree.qza \
--p-max-depth 10000 \
--m-metadata-file sample-metadata.tsv \
--o-visualization alpha-rarefaction.qzv
注意:提供的–p-max-depth参数的值应该通过查看上面创建的table.qzv文件中呈现的“每个样本的测序量”信息来确定。一般来说,选择一个在中位数附近的值似乎很好用。如果得到的稀疏图中的线看起来没有变平,那么你可能希望增加该值。如果由于大于最大采样深度而丢失了许多样本,则减少该值。
十、物种组成分析
qiime2中的物种注释功能,用户可以下载用于比对的数据库,并提供3种不同的方法(两种alignment-based的方法和一种基于naive-bayes的方法),自己train一个classifer用于物种分类,也可以下载并使用官方已经train好的(pre-trained)classifer。
不过GPZ_lab的教程中一般推荐的还是使用基于naive-bayes的方法。
并且它的实例是使用官方已经train好的classifergg-13-8-99-nb-classifier.qza(greengene),可以在官网上下载。
需要注意的是,常用的16s数据库有rdp、greengene、Silver,官网只可以下载后两个的。另外即使测序测的是V3V4,也可以选择16s全长的数据库进行训练,对结果并没有什么影响(naive-bayes based method)。
1.下载物种训练分类器
wget \
-O "silva-132-99-nb-classifier.qza" \
https://data.qiime2.org/2019.7/common/silva-132-99-nb-classifier.qza
2.物种注释和可视化
time qiime feature-classifier classify-sklearn \
--i-classifier silva-132-99-nb-classifier.qza \
--i-reads rep-seqs.qza \
--o-classification taxonomy.qza
qiime metadata tabulate \
--m-input-file taxonomy.qza \
--o-visualization taxonomy.qzv
输出结果:
①物种注释结果:taxonomy.qza
②分类器的训练结果:gg-13-8-99-515-806-nb-classifier.qza
可视化结果:
①物种注释可视化:taxonomy.qzv
接下来,可以用交互式条形图查看样本的分类组成。使用以下命令绘图堆叠柱状图,然后打开查看。
qiime taxa barplot \
--i-table table.qza \
--i-taxonomy taxonomy.qza \
--m-metadata-file sample-metadata.tsv \
--o-visualization taxa-bar-plots.qzv
输出结果:
①交互式物种组成堆叠柱状图:taxa-bar-plots.qzv
该文件是一个bar图,可以观察物种组成(优势物种),可以选择不同的物种levels进行绘制。