- 【数据结构】详解堆排序当中的topk问题(leetcode例题)
ylfxw
数据结构leetcode算法
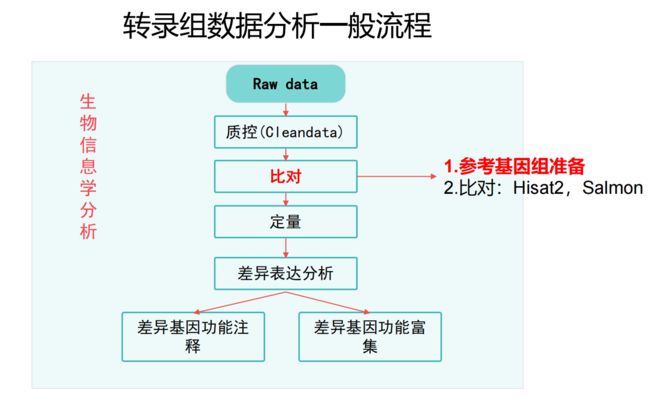
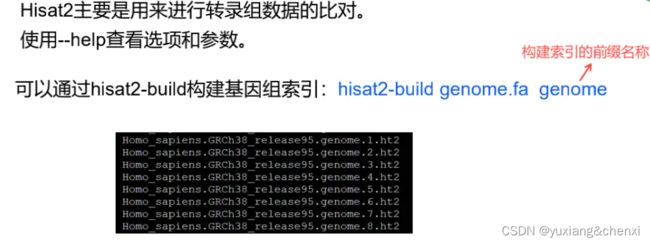
文章目录前言如何理解topk问题代码逻辑代码实现前言Leetcode相关题目:215.数组中的第K个最大元素如何理解topk问题**TopK问题是一个经典的问题,在计算机科学中,它的目标是在一组数据中找到前K个最大或最小的元素。**这个问题在许多场景下都很重要,比如搜索引擎的搜索结果排名、数据分析中的热门元素筛选等。.在最简单的形式中,给定一个数组(或列表)和一个整数K,TopK问题要求返回数组中
- 拼多多官方返利新动向,高省App引领购物省钱新趋势
古楼
电商行业的快速发展带来了无数的新趋势和新机遇,而拼多多官方返利的新趋势无疑是其中的一大亮点。高省App作为这一趋势的敏锐洞察者和积极参与者,致力于帮助用户精准把握这些新机遇。通过高省App,用户可以及时了解拼多多官方返利的最新政策和活动信息,从而做出更加明智的购物决策。同时,高省App还提供了专业的数据分析工具,帮助用户分析自己的消费行为和省钱效果,让省钱之路更加清晰和明确。我们在开始讲今天的文章
- 2023-04-12
王松奇
京心❤️达理想城店:王松奇2023年4月12日日精进落地真经严格就是爱,放纵既是害正能量语录每一颗螺丝都有标准每一颗螺丝都是标准产值目标:13万台次目标:80台油卡目标:13张今日体验今天开数据分析会台次少保养预存一定要盯紧中间10天要努力冲刺一下
- 小程序领域的营销推广策略
小程序开发2020
小程序ai
小程序领域的营销推广策略:从流量获取到生态运营的全链路解析关键词:小程序营销、用户增长策略、社交裂变、私域流量运营、数据分析驱动、场景化营销、全域流量整合摘要:本文系统解析小程序营销推广的核心策略体系,从微信生态底层逻辑出发,结合用户生命周期管理理论,构建包含「用户拉新-留存转化-裂变增长-数据迭代」的全链路运营框架。通过深度拆解社交裂变模型、场景化运营策略、私域流量沉淀方法及数据驱动决策体系,结
- 大学专业科普 | 计算智能、信息学与大数据
鸭鸭鸭进京赶烤
大数据
一、专业背景随着信息技术的飞速发展,数据的产生速度呈爆炸式增长,传统数据处理技术已经无法满足如此庞大的数据量和复杂的数据类型,大数据专业应运而生,旨在培养能够应对大数据挑战的专业人才。二、主要课程内容数学基础课程高等数学、概率论与数理统计、线性代数是大数据分析的核心数学基础,为数据处理、算法优化和模型构建提供必要的理论支持。计算机基础课程数据结构与算法、计算机网络、操作系统是大数据技术的重要支撑,
- 【源码交付】一站式自助数据分析解决方案(jvs-bi)
愤怒的小青春
java
简历咨询听说Java简历上写外卖,头条,商城项目没用,到底真的假的。不写这些还能写什么#简历中的项目经历要美团实习体验~❤️入职流程和体验入职先领工牌,电脑(可提前在网上申请入职电脑版本,技术岗应该是mac)还可以申请显24offer帮选个人情况:本硕末流211科班光大银行总行科技研发中心入职:总包24w最高:涨幅两三年普调一级,涨一级简历咨询听说项目写外卖,头条,商城项目没用。有一说一,真的没有
- 数据分析全攻略:从基础概念到实战应用的完整指南
SickeyLee
产品经理人工智能大数据信息可视化
数据分析全攻略:从基础概念到实战应用的完整指南数据分析已成为现代商业决策的核心驱动力,但很多人在面对数据时,常常陷入“不知道看什么、怎么分析、如何应用”的困境。本文将系统梳理数据分析的核心知识,从数据的本质到分析流程,从方法工具到实战指标,帮你搭建一套完整的数据分析思维框架,让数据真正为业务服务。一、数据是什么?不止于数字的“信息载体”提到数据,很多人会首先想到数字,但实际上数据的范畴远更广阔。数
- Python Pandas 实践学习笔记(1)
PythonPandas教程Pandas是一个开源的、BSD许可证的Python库,为Python编程语言提供高性能、易于使用的数据结构和数据分析工具。Python与Pandas在学术和商业领域都被广泛应用,包括金融、经济、统计学、分析等领域。在本教程中,我们将学习PythonPandas的各种特性以及如何在实践中使用它们。教程对象本教程适用于那些想要学习Pandas基础知识和各种函数的人。对于从
- 基于Paillier同态加密算法的金融数据安全共享机制研究【附数据】
金融数据分析与建模专家金融科研助手|论文指导|模型构建✨专业领域:金融数据处理与分析量化交易策略研究金融风险建模投资组合优化金融预测模型开发深度学习在金融中的应用擅长工具:Python/R/MATLAB量化分析机器学习模型构建金融时间序列分析蒙特卡洛模拟风险度量模型金融论文指导内容:金融数据挖掘与处理量化策略开发与回测投资组合构建与优化金融风险评估模型期刊论文✅具体问题可以私信或查看文章底部二维码
- 综合智能监测系统设计:有害气体实时检测与管理
黑泡尖子
本文还有配套的精品资源,点击获取简介:在工业化进程中,有害气体的排放对人类健康和环境构成威胁。设计一种智能监测系统,利用传感器技术、物联网和数据分析等,实时监控环境中有害气体的浓度,确保生产安全和环保。该系统涵盖硬件构建、软件开发和数据处理等环节,并提供高效准确的监测能力。系统集成了无线通信模块进行数据传输,具备数据预处理和分析能力,能够进行阈值设定与预警响应。用户界面友好,系统具有良好的集成性、
- 【Grafana】Prometheus指标可视化Grafana,手把手教你如何自定义图形
景天科技苑
grafanaprometheusprometheus可视化grafana自定义图形手撕grafana自定义监控图形
✨✨欢迎大家来到景天科技苑✨✨养成好习惯,先赞后看哦~作者简介:景天科技苑《头衔》:大厂架构师,华为云开发者社区专家博主,阿里云开发者社区专家博主,CSDN全栈领域优质创作者,掘金优秀博主,51CTO博客专家等。《博客》:Python全栈,前后端开发,小程序开发,人工智能,js逆向,App逆向,网络系统安全,数据分析,Django,fastapi,flask等框架,云原生k8s,Prometheu
- 高省app没有邀请码怎么注册?高省app总部邀请码是什么?
古楼
高省是正规平台吗?高省app是杭州长孚科技有限公司旗下的一款电商导购应用,为用户打造一个电商购物优惠平台,用户可以在这个App中领取主流商城的商品隐藏优惠券以及获得返利。基于第三方电商平台海量数据挖掘与分析,“高省”APP通过内容制作、分享等方式,为消费者打通吃喝玩乐购全场景全业态,让消费者省心省钱省时省力,为平台和品牌方导流创造收入,拓展了商家新的销售渠道。高省app逐渐构筑起了集各大主流电商平
- 时序数据库:数据库领域的未来之星
数据库管理艺术
数据库专家之路大数据AI人工智能MCP&AgentSQL实战数据库时序数据库ai
时序数据库:数据库领域的未来之星关键词:时序数据库、时间序列数据、物联网、大数据分析、数据库优化、TSDB、实时数据处理摘要:本文深入探讨了时序数据库(TimeSeriesDatabase,TSDB)这一新兴数据库技术。我们将从基本概念入手,分析时序数据库的核心原理和架构设计,详细讲解其特有的数据模型和存储机制。通过实际代码示例展示如何使用主流时序数据库处理时间序列数据,并探讨其在物联网、金融科技
- 基于Python的Twitter Card数据爬取与分析实战:从入门到精通
Python爬虫项目
pythontwitterdreamweaver自动化开发语言宽度优先爬虫
摘要本文详细介绍了如何使用Python最新技术栈构建一个高效的TwitterCard数据爬虫系统。我们将从TwitterCard的基本概念讲起,逐步深入到爬虫架构设计、反爬策略应对、数据解析与存储等核心环节。文章包含完整的代码实现,使用Playwright+Asyncio的高性能爬取方案,以及数据分析与可视化的实战案例。通过本文,读者将掌握大规模社交媒体数据采集的关键技术,并能够将这些技术应用于实
- 使用 Python 爬取网易云音乐歌单数据(完整教程)
Python爬虫项目
python开发语言githubselenium爬虫
一、引言随着在线音乐平台的普及,网易云音乐(NetEaseCloudMusic)凭借其个性化的推荐算法和丰富的用户互动,吸引了大量用户。网易云音乐的歌单中包含了丰富的音乐数据,包括歌曲名、歌手、专辑、播放量、评论数等信息。通过爬取这些数据,可以对音乐流行趋势进行分析,挖掘音乐推荐策略,甚至训练个性化推荐模型。本教程将使用Python构建一个爬虫,解析网易云音乐的歌单接口,获取歌曲数据并进行数据分析
- 全球气温逐年增高
CATTLECODE
人工智能
根据全球主要气候监测机构的权威数据,**全球气温确实在持续升高**,且呈现加速趋势。以下是关键事实和数据分析:一、科学共识与核心数据长期升温趋势(1880-2023):工业革命前(1850-1900)相比:全球平均气温上升约1.45°C2023年成为有记录以来最热年份(比19世纪基线高1.48°C)过去10年(2014-2023)是史上最热的十年(WMO数据)加速升温证据:二、权威机构数据验证机构
- 计算机专业大数据毕业设计-基于 Spark 的音乐数据分析项目(源码+LW+部署文档+全bao+远程调试+代码讲解等)
程序猿八哥
数据可视化计算机毕设spark大数据课程设计spark
博主介绍:✌️码农一枚,专注于大学生项目实战开发、讲解和毕业文撰写修改等。全栈领域优质创作者,博客之星、掘金/华为云/阿里云/InfoQ等平台优质作者、专注于Java、小程序技术领域和毕业项目实战✌️技术范围::小程序、SpringBoot、SSM、JSP、Vue、PHP、Java、python、爬虫、数据可视化、大数据、物联网、机器学习等设计与开发。主要内容:免费功能设计,开题报告、任务书、全b
- 如何使用爬虫简单的爬取一个网页的静态前端代码
什么是爬虫?Python爬虫是一种使用Python语言编写的程序,用于自动访问网页并提取所需信息。它通常用于网络数据抓取、数据挖掘和信息收集。Python爬虫可以模拟浏览器行为,向服务器发送请求并接收响应数据,然后解析这些数据以获取有用的信息。爬虫的基本原理(流程)发送请求:爬虫向目标网站的服务器发送HTTP请求(通常是GET请求)。获取响应:服务器返回网页的HTML内容。解析内容:爬虫解析HTM
- MySQL(141)如何处理重复数据问题?
辞暮尔尔-烟火年年
MySQLmysql数据库
处理重复数据问题是数据管理中的一个常见挑战。重复数据会影响数据库的性能、占用资源,并且可能导致数据分析结果的偏差。以下是处理重复数据问题的详细步骤以及结合代码的示例。一、识别重复数据首先,需要识别数据库中的重复数据。可以使用SQL查询来查找重复的数据。示例:假设我们有一个名为employees的表,其中包含以下字段:id、name和email。CREATETABLEemployees(idINTP
- 如何用优惠卷赚钱?优惠券群怎么才能做起来?
日常购物技巧呀
在数字化时代,社群经济日益繁荣,尤其是以优惠券为主题的社群,不仅能够帮助群成员省钱购物,还能为群主带来可观的收入。本文将详细解析如何建立和管理一个赚钱的优惠券群,包括群的建设、运营策略,以及如何通过专业数据分析来优化群的效果。目标读者包括宝妈、大学生、上班族和无业人员,这些群体通常对节省开支和额外收入有较高的需求。大家好,我是高省返利APP官方客服导师:童年,今日给大家推荐一款自用佣金高,还能做团
- 使用Python Scrapy打造个性化爬虫
使用PythonScrapy打造个性化爬虫——知识金字塔构建1.引入与连接:从“手动复制”到“自动化采集”的跨越你是否遇到过这样的场景?想整理1000条知乎优质回答做数据分析,却要逐条复制;想追踪某电商平台的商品价格波动,却要每天手动刷新页面……这些重复劳动,正是“个性化爬虫”的用武之地!与已有知识的连接:你可能用过requests+BeautifulSoup写过简单爬虫,但面对大规模数据、复杂反
- Python 数据插值:NumPy 实现多种插值方法
Python数据插值:用NumPy解锁缺失数据的秘密拼图关键词数据插值、NumPy、线性插值、多项式插值、缺失值处理、数据平滑、数值分析摘要在数据分析和科学计算中,我们经常遇到离散或缺失的观测数据——比如气象站每小时记录的温度值有缺失,或者实验中只采集了稀疏的采样点。这时候,数据插值(Interpolation)就像“数据修复师”,能根据已知点推断出未知点的数值,让离散数据变成连续的“故事”。本文
- Shell脚本-cut工具
咖啡の猫
chrome前端
一、前言在Linux/Unix系统中,cut是一个非常实用的文本处理命令,用于从文件或标准输入中提取特定列的内容。它特别适用于处理结构化文本数据,例如CSV文件、日志文件、配置文件等。无论是做数据分析、系统监控,还是编写自动化脚本,cut都是一个不可或缺的工具。本文将带你全面了解cut工具的使用方式,包括:✅cut的基本语法与常用参数✅如何按字符、字节、字段进行提取✅cut在Shell脚本中的实战
- 必看:购买黄金投资局中局曝光!伍戈碳中和云数据分析就是骗局!
不成功不收费
被骗案例:聊天软件讲股票,又忽悠股民搞碳中和项目,云数据分析项目等才后来让我们自己投钱怕我们不相信还请反诉警员给上课,给我画大饼,说每月发6千元的工资,所以放松警惕上当了,投了钱,我投的少,还特别照顾我单独做,达到他们的标准,他们明知我没有钱,每天照顾在两次,赚了一百多万,让我提款必须交税5%,我没钱无法交税,一分也取不出来,而且还有时侯规定,我只能自已做让账户回到我能交的起税的金额,但你要交钱时
- Excel处理控件Aspose.Cells指南:使用 Python 删除 Excel 中的重复行
CodeCraft Studio
文档管理控件pythonexcel开发语言
在Excel中删除重复行对于维护干净、准确和一致的数据集至关重要。它可以确保一致性,并有助于防止分析或报告中出现错误。重复数据会导致错误的分析和糟糕的决策。因此,识别和消除重复数据的能力对于软件开发人员、数据分析师和Excel用户来说是一项宝贵的技能。在本篇博文中,我们将向您展示如何使用Python以编程方式删除Excel工作表中的重复行。Python库用于删除Excel中的重复行Aspose.C
- Elasticsearch 聚合查询源码解读与架构方法论
北漂老男人
Elasticsearchelasticsearch架构大数据搜索引擎全文检索
Elasticsearch聚合查询源码解读与架构方法论01.引言Elasticsearch的聚合查询(Aggregation)是大规模分布式数据分析的核心能力。理解其源码结构与设计方法论,不仅有助于高效使用聚合,也能为自定义扩展、性能优化、集群运维等提供理论与实践基础。本文将从源码结构、核心模块、关键实现、行级注释与方法论出发,系统剖析聚合查询的底层原理。02.源码结构与核心模块2.1聚合相关源码
- python连接数据库的方法,Python 连接数据库的多种方法
AI MIU
python连接数据库的方法
JZGKCHINAPython是一种计算机程序设计语言,它是一种动态的、面向对象的脚本语言。它是一种跨平台的,可以运行在Windows,Mac和Linux/Unix系统上。在日常使用中需要对大量数据进行数据分析,那么就必然用到数据库,我们常用的数据库有SQLServer,MySQL,Oracle,DB2,SQLite,Hive,PostgreSQL,MongoDB还有其他常用的MicrosoftA
- Python领域制造业的Python应用
Python编程之道
Python编程之道python开发语言ai
Python在制造业中的应用:从自动化到智能制造关键词:Python、制造业、工业自动化、数据分析、机器学习、物联网、智能制造摘要:本文深入探讨Python编程语言在制造业中的广泛应用。从基础的自动化脚本到复杂的智能制造系统,Python凭借其丰富的库生态系统和易用性,正在重塑现代制造业。我们将分析Python在制造业中的核心应用场景,包括设备监控、质量控制、预测性维护和供应链优化等,并通过实际案
- 十种常用数据分析模型
耐思nice~
数据分析数据分析人工智能机器学习数学建模
1-线性回归(LinearRegression)场景:预测商品销售额优点:简单易用,结果易于解释缺点:假设线性关系,容易受到异常值影响概念:建立自变量和因变量之间线性关系的模型。公式:[y=b_0+b_1x_1+b_2x_2+...+b_nx_n]代码示例:importpandasaspdfromsklearn.linear_modelimportLinearRegressionfromsklea
- 【Pandas超实用经验汇总-数据建模分析】
Mr.小海
Python数据挖掘数据分析python
Pandas超实用经验汇总-数据分析前言基本方法1.读取文件2.查看数据3.修改、删除、替换数据等总结前言看见了很多教程虽然很全,但是很多技巧容易忘记且几乎用不上,读起来晦涩难懂,今天我给大家总结了Pandas的一些学习经验技巧,包含常见日常使用的pandas知识,以及一些技巧,这些技巧常见于数学建模,数据分析,数据挖掘比赛等。基本方法1.读取文件方法如下:importpandasaspd#正常写
- 集合框架
天子之骄
java数据结构集合框架
集合框架
集合框架可以理解为一个容器,该容器主要指映射(map)、集合(set)、数组(array)和列表(list)等抽象数据结构。
从本质上来说,Java集合框架的主要组成是用来操作对象的接口。不同接口描述不同的数据类型。
简单介绍:
Collection接口是最基本的接口,它定义了List和Set,List又定义了LinkLi
- Table Driven(表驱动)方法实例
bijian1013
javaenumTable Driven表驱动
实例一:
/**
* 驾驶人年龄段
* 保险行业,会对驾驶人的年龄做年龄段的区分判断
* 驾驶人年龄段:01-[18,25);02-[25,30);03-[30-35);04-[35,40);05-[40,45);06-[45,50);07-[50-55);08-[55,+∞)
*/
public class AgePeriodTest {
//if...el
- Jquery 总结
cuishikuan
javajqueryAjaxWebjquery方法
1.$.trim方法用于移除字符串头部和尾部多余的空格。如:$.trim(' Hello ') // Hello2.$.contains方法返回一个布尔值,表示某个DOM元素(第二个参数)是否为另一个DOM元素(第一个参数)的下级元素。如:$.contains(document.documentElement, document.body); 3.$
- 面向对象概念的提出
麦田的设计者
java面向对象面向过程
面向对象中,一切都是由对象展开的,组织代码,封装数据。
在台湾面向对象被翻译为了面向物件编程,这充分说明了,这种编程强调实体。
下面就结合编程语言的发展史,聊一聊面向过程和面向对象。
c语言由贝尔实
- linux网口绑定
被触发
linux
刚在一台IBM Xserver服务器上装了RedHat Linux Enterprise AS 4,为了提高网络的可靠性配置双网卡绑定。
一、环境描述
我的RedHat Linux Enterprise AS 4安装双口的Intel千兆网卡,通过ifconfig -a命令看到eth0和eth1两张网卡。
二、双网卡绑定步骤:
2.1 修改/etc/sysconfig/network
- XML基础语法
肆无忌惮_
xml
一、什么是XML?
XML全称是Extensible Markup Language,可扩展标记语言。很类似HTML。XML的目的是传输数据而非显示数据。XML的标签没有被预定义,你需要自行定义标签。XML被设计为具有自我描述性。是W3C的推荐标准。
二、为什么学习XML?
用来解决程序间数据传输的格式问题
做配置文件
充当小型数据库
三、XML与HTM
- 为网页添加自己喜欢的字体
知了ing
字体 秒表 css
@font-face {
font-family: miaobiao;//定义字体名字
font-style: normal;
font-weight: 400;
src: url('font/DS-DIGI-e.eot');//字体文件
}
使用:
<label style="font-size:18px;font-famil
- redis范围查询应用-查找IP所在城市
矮蛋蛋
redis
原文地址:
http://www.tuicool.com/articles/BrURbqV
需求
根据IP找到对应的城市
原来的解决方案
oracle表(ip_country):
查询IP对应的城市:
1.把a.b.c.d这样格式的IP转为一个数字,例如为把210.21.224.34转为3524648994
2. select city from ip_
- 输入两个整数, 计算百分比
alleni123
java
public static String getPercent(int x, int total){
double result=(x*1.0)/(total*1.0);
System.out.println(result);
DecimalFormat df1=new DecimalFormat("0.0000%");
- 百合——————>怎么学习计算机语言
百合不是茶
java 移动开发
对于一个从没有接触过计算机语言的人来说,一上来就学面向对象,就算是心里上面接受的了,灵魂我觉得也应该是跟不上的,学不好是很正常的现象,计算机语言老师讲的再多,你在课堂上面跟着老师听的再多,我觉得你应该还是学不会的,最主要的原因是你根本没有想过该怎么来学习计算机编程语言,记得大一的时候金山网络公司在湖大招聘我们学校一个才来大学几天的被金山网络录取,一个刚到大学的就能够去和
- linux下tomcat开机自启动
bijian1013
tomcat
方法一:
修改Tomcat/bin/startup.sh 为:
export JAVA_HOME=/home/java1.6.0_27
export CLASSPATH=$CLASSPATH:$JAVA_HOME/lib/tools.jar:$JAVA_HOME/lib/dt.jar:.
export PATH=$JAVA_HOME/bin:$PATH
export CATALINA_H
- spring aop实例
bijian1013
javaspringAOP
1.AdviceMethods.java
package com.bijian.study.spring.aop.schema;
public class AdviceMethods {
public void preGreeting() {
System.out.println("--how are you!--");
}
}
2.beans.x
- [Gson八]GsonBuilder序列化和反序列化选项enableComplexMapKeySerialization
bit1129
serialization
enableComplexMapKeySerialization配置项的含义
Gson在序列化Map时,默认情况下,是调用Key的toString方法得到它的JSON字符串的Key,对于简单类型和字符串类型,这没有问题,但是对于复杂数据对象,如果对象没有覆写toString方法,那么默认的toString方法将得到这个对象的Hash地址。
GsonBuilder用于
- 【Spark九十一】Spark Streaming整合Kafka一些值得关注的问题
bit1129
Stream
包括Spark Streaming在内的实时计算数据可靠性指的是三种级别:
1. At most once,数据最多只能接受一次,有可能接收不到
2. At least once, 数据至少接受一次,有可能重复接收
3. Exactly once 数据保证被处理并且只被处理一次,
具体的多读几遍http://spark.apache.org/docs/lates
- shell脚本批量检测端口是否被占用脚本
ronin47
#!/bin/bash
cat ports |while read line
do#nc -z -w 10 $line
nc -z -w 2 $line 58422>/dev/null2>&1if[ $?-eq 0]then
echo $line:ok
else
echo $line:fail
fi
done
这里的ports 既可以是文件
- java-2.设计包含min函数的栈
bylijinnan
java
具体思路参见:http://zhedahht.blog.163.com/blog/static/25411174200712895228171/
import java.util.ArrayList;
import java.util.List;
public class MinStack {
//maybe we can use origin array rathe
- Netty源码学习-ChannelHandler
bylijinnan
javanetty
一般来说,“有状态”的ChannelHandler不应该是“共享”的,“无状态”的ChannelHandler则可“共享”
例如ObjectEncoder是“共享”的, 但 ObjectDecoder 不是
因为每一次调用decode方法时,可能数据未接收完全(incomplete),
它与上一次decode时接收到的数据“累计”起来才有可能是完整的数据,是“有状态”的
p
- java生成随机数
cngolon
java
方法一:
/**
* 生成随机数
* @author
[email protected]
* @return
*/
public synchronized static String getChargeSequenceNum(String pre){
StringBuffer sequenceNum = new StringBuffer();
Date dateTime = new D
- POI读写海量数据
ctrain
海量数据
import java.io.FileOutputStream;
import java.io.OutputStream;
import org.apache.poi.xssf.streaming.SXSSFRow;
import org.apache.poi.xssf.streaming.SXSSFSheet;
import org.apache.poi.xssf.streaming
- mysql 日期格式化date_format详细使用
daizj
mysqldate_format日期格式转换日期格式化
日期转换函数的详细使用说明
DATE_FORMAT(date,format) Formats the date value according to the format string. The following specifiers may be used in the format string. The&n
- 一个程序员分享8年的开发经验
dcj3sjt126com
程序员
在中国有很多人都认为IT行为是吃青春饭的,如果过了30岁就很难有机会再发展下去!其实现实并不是这样子的,在下从事.NET及JAVA方面的开发的也有8年的时间了,在这里在下想凭借自己的亲身经历,与大家一起探讨一下。
明确入行的目的
很多人干IT这一行都冲着“收入高”这一点的,因为只要学会一点HTML, DIV+CSS,要做一个页面开发人员并不是一件难事,而且做一个页面开发人员更容
- android欢迎界面淡入淡出效果
dcj3sjt126com
android
很多Android应用一开始都会有一个欢迎界面,淡入淡出效果也是用得非常多的,下面来实现一下。
主要代码如下:
package com.myaibang.activity;
import android.app.Activity;import android.content.Intent;import android.os.Bundle;import android.os.CountDown
- linux 复习笔记之常见压缩命令
eksliang
tar解压linux系统常见压缩命令linux压缩命令tar压缩
转载请出自出处:http://eksliang.iteye.com/blog/2109693
linux中常见压缩文件的拓展名
*.gz gzip程序压缩的文件
*.bz2 bzip程序压缩的文件
*.tar tar程序打包的数据,没有经过压缩
*.tar.gz tar程序打包后,并经过gzip程序压缩
*.tar.bz2 tar程序打包后,并经过bzip程序压缩
*.zi
- Android 应用程序发送shell命令
gqdy365
android
项目中需要直接在APP中通过发送shell指令来控制lcd灯,其实按理说应该是方案公司在调好lcd灯驱动之后直接通过service送接口上来给APP,APP调用就可以控制了,这是正规流程,但我们项目的方案商用的mtk方案,方案公司又没人会改,只调好了驱动,让应用程序自己实现灯的控制,这不蛋疼嘛!!!!
发就发吧!
一、关于shell指令:
我们知道,shell指令是Linux里面带的
- java 无损读取文本文件
hw1287789687
读取文件无损读取读取文本文件charset
java 如何无损读取文本文件呢?
以下是有损的
@Deprecated
public static String getFullContent(File file, String charset) {
BufferedReader reader = null;
if (!file.exists()) {
System.out.println("getFull
- Firebase 相关文章索引
justjavac
firebase
Awesome Firebase
最近谷歌收购Firebase的新闻又将Firebase拉入了人们的视野,于是我做了这个 github 项目。
Firebase 是一个数据同步的云服务,不同于 Dropbox 的「文件」,Firebase 同步的是「数据」,服务对象是网站开发者,帮助他们开发具有「实时」(Real-Time)特性的应用。
开发者只需引用一个 API 库文件就可以使用标准 RE
- C++学习重点
lx.asymmetric
C++笔记
1.c++面向对象的三个特性:封装性,继承性以及多态性。
2.标识符的命名规则:由字母和下划线开头,同时由字母、数字或下划线组成;不能与系统关键字重名。
3.c++语言常量包括整型常量、浮点型常量、布尔常量、字符型常量和字符串性常量。
4.运算符按其功能开以分为六类:算术运算符、位运算符、关系运算符、逻辑运算符、赋值运算符和条件运算符。
&n
- java bean和xml相互转换
q821424508
javabeanxmlxml和bean转换java bean和xml转换
这几天在做微信公众号
做的过程中想找个java bean转xml的工具,找了几个用着不知道是配置不好还是怎么回事,都会有一些问题,
然后脑子一热谢了一个javabean和xml的转换的工具里,自己用着还行,虽然有一些约束吧 ,
还是贴出来记录一下
顺便你提一下下,这个转换工具支持属性为集合、数组和非基本属性的对象。
packag
- C 语言初级 位运算
1140566087
位运算c
第十章 位运算 1、位运算对象只能是整形或字符型数据,在VC6.0中int型数据占4个字节 2、位运算符: 运算符 作用 ~ 按位求反 << 左移 >> 右移 & 按位与 ^ 按位异或 | 按位或 他们的优先级从高到低; 3、位运算符的运算功能: a、按位取反: ~01001101 = 101
- 14点睛Spring4.1-脚本编程
wiselyman
spring4
14.1 Scripting脚本编程
脚本语言和java这类静态的语言的主要区别是:脚本语言无需编译,源码直接可运行;
如果我们经常需要修改的某些代码,每一次我们至少要进行编译,打包,重新部署的操作,步骤相当麻烦;
如果我们的应用不允许重启,这在现实的情况中也是很常见的;
在spring中使用脚本编程给上述的应用场景提供了解决方案,即动态加载bean;
spring支持脚本