【大数据】美国新冠肺炎疫情分析——正确版(QDU)
- 【大数据】蔬菜价格分析(QDU)
- 【大数据】美国新冠肺炎疫情分析——错误版(QDU)
- 【大数据】美国新冠肺炎疫情分析——正确版(QDU)
- 【大数据】乳腺癌预测——老师给的链接(QDU)
- 由于kaggle上“猫狗大战”的测试集标签是错的,所以没做出来,用的github上的代码
- 【大数据】《红楼梦》作者分析(QDU)
问题分析
已知从2020年1月28日到2020年9月8日新冠肺炎患者“新增”数量、“总确诊”数量、“总治愈”数量和“总死亡”数量,分析这若干个月内数量变化规律并构建合适的模型预测未来一个月数量的变化趋势。
解决思路
问题缺陷
上周完成的”美国新冠分析“的实验主要的思路是:六次多项式拟合得到”新增数量“的变化,由已知信息计算得到”现存确诊数量“后,根据”新增数量“、”总确诊数量“、”治愈数量“和”死亡数量“之间的关系建立模型,迭代30次获得未来一个月的信息。对美国新冠分析的原意是预测未来”总确诊数量“,但实验中却通过”新增“变化直接得到了”总确诊数量“,本质上是对未来一个月的”治愈“和”死亡“进行预测,与原意严重不符。另一方面,该模型过度依赖一个”新增数量“因素,对于”新增数量“预测的好坏严重影响最终结果,且对于关键因素”新增数量“的预测采用多项式拟合的方式使得不合理性进一步增加,导致最终结果不能令人信服。
改进方案
本次实验的改进主要涉及以下几个方面:
- 预测目标:从上周的错误预测目标”治愈“和”死亡“转变为对”总确诊数量“的预测。
- 关键因素:从实际情况出发,传染病的传播主要受现存确诊人数的影响,即现存确诊越多,传染病的传播越迅速。
上次实验相比,本次实验仍采用根据关系进行迭代的方式计算。
关联已知信息
线性回归的标准形式:
y = X w + b y = Xw+b y=Xw+b
采用最小二乘的方式:
平 方 误 差 : ∑ i = 1 N ( y i − x i T w ) 2 平方误差: \sum_{i=1}^N(y_i-x_i^Tw)^2 平方误差:i=1∑N(yi−xiTw)2
保证平方误差最小,令式子的导数为零可得:
w ^ = ( X T X ) − 1 X T y \hat{w}=(X^TX)^{-1}X^Ty w^=(XTX)−1XTy
定义如下若干变量:
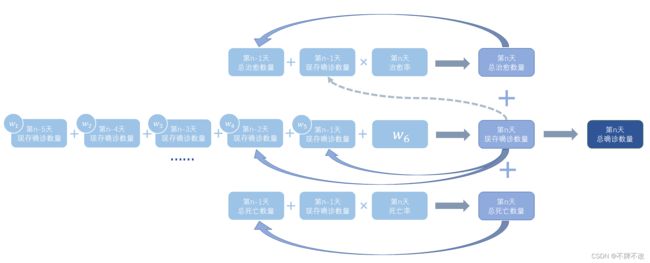
- 治 愈 n 治愈_n 治愈n:表示第n天的治愈总数, 治 愈 n = 治 愈 率 n × 现 存 确 诊 n − 1 + 治 愈 n − 1 治愈_n = 治愈率_n × 现存确诊_{n-1} + 治愈_{n-1} 治愈n=治愈率n×现存确诊n−1+治愈n−1
- 死 亡 n 死亡_n 死亡n:表示第n天的死亡总数, 死 亡 n = 死 亡 率 n × 现 存 确 诊 n − 1 + 死 亡 n − 1 死亡_n = 死亡率_n × 现存确诊_{n-1} + 死亡_{n-1} 死亡n=死亡率n×现存确诊n−1+死亡n−1
- 现 存 确 诊 n 现存确诊_n 现存确诊n:表示第n天现存的确诊人数,由线性回归和迭代而来
- 治 愈 率 n 治愈率_n 治愈率n:表示第n天的治愈率, 治 愈 率 n = ( 治 愈 n − 治 愈 n − 1 ) / 现 存 确 诊 n − 1 治愈率_n = (治愈_n - 治愈_{n-1}) / 现存确诊_{n-1} 治愈率n=(治愈n−治愈n−1)/现存确诊n−1
- 死 亡 率 n 死亡率_n 死亡率n:表示第n天的死亡率, 死 亡 率 n = ( 死 亡 n − 死 亡 n − 1 ) / 现 存 确 诊 n − 1 死亡率_n = (死亡_n - 死亡_{n-1}) / 现存确诊_{n-1} 死亡率n=(死亡n−死亡n−1)/现存确诊n−1
- 总 确 诊 n 总确诊_n 总确诊n:表示第n天的确诊总数, 总 确 诊 n = 现 存 确 诊 n + 治 愈 n + 死 亡 n 总确诊_n=现存确诊_n+治愈_n+死亡_n 总确诊n=现存确诊n+治愈n+死亡n
我建立的线性回归函数主要考虑前五天”现存确诊数量“对当天”现存确诊数量“的影响,即 w = [ [ w 1 ] , [ w 2 ] , [ w 3 ] , [ w 4 ] , [ w 5 ] , [ w 6 ] ] w = [[w_1], [w_2], [w_3], [w_4], [w_5], [w_6]] w=[[w1],[w2],[w3],[w4],[w5],[w6]], w 1 ∼ w 5 w_1 \sim w_5 w1∼w5分别表示 现 存 确 诊 n − 5 、 现 存 确 诊 n − 4 、 现 存 确 诊 n − 3 、 现 存 确 诊 n − 2 、 现 存 确 诊 n − 1 现存确诊_{n-5}、现存确诊_{n-4}、现存确诊_{n-3}、现存确诊_{n-2}、现存确诊_{n-1} 现存确诊n−5、现存确诊n−4、现存确诊n−3、现存确诊n−2、现存确诊n−1的系数, w 6 w_6 w6表示方程中的常数项 b b b。
得到第 n n n天的现存确诊人数满足的方程:
现 存 确 诊 n = w 1 × 现 存 确 诊 n − 5 + w 2 × 现 存 确 诊 n − 4 + w 3 × 现 存 确 诊 n − 3 + w 4 × 现 存 确 诊 n − 2 + w 5 × 现 存 确 诊 n − 1 + w 6 × 1 现存确诊_n = w_1×现存确诊_{n-5} + w_2×现存确诊_{n-4} + w_3×现存确诊_{n-3} + w_4×现存确诊_{n-2} + w_5×现存确诊_{n-1} + w_6×1 现存确诊n=w1×现存确诊n−5+w2×现存确诊n−4+w3×现存确诊n−3+w4×现存确诊n−2+w5×现存确诊n−1+w6×1
构建 X X X,其每一项为 [ 现 存 确 诊 n − 5 , 现 存 确 诊 n − 4 , 现 存 确 诊 n − 3 , 现 存 确 诊 n − 2 , 现 存 确 诊 n − 1 , 1 ] [\space现存确诊_{n-5},\space现存确诊_{n-4},\space现存确诊_{n-3},\space现存确诊_{n-2},\space现存确诊_{n-1},\space 1\space] [ 现存确诊n−5, 现存确诊n−4, 现存确诊n−3, 现存确诊n−2, 现存确诊n−1, 1 ]
迭代思路:
解决方案
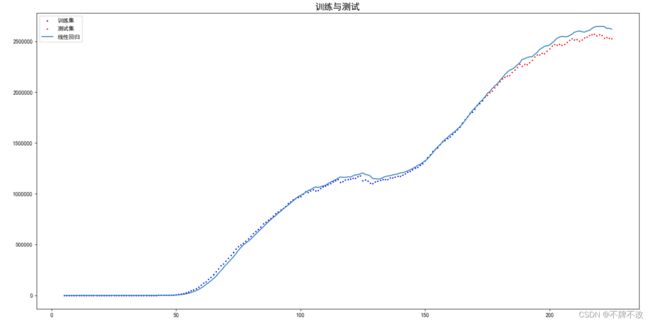
经多次调整训练集与测试集的划分比例,将已知数据集按 0.77 : 0.23 0.77:0.23 0.77:0.23的比例划分为训练集和测试集测试效果较佳且预测效果最合理。因此采用此方案获取的模型进行预测。
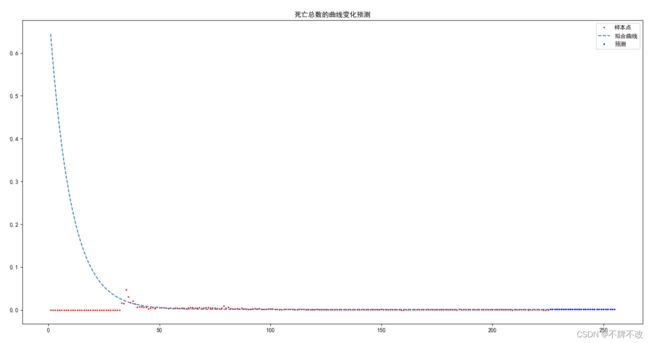
与上周实验相同,采用指数函数拟合治愈率和死亡率。
治愈数量曲线如下:
死亡数量曲线如下:
(对于早期数据采取忽略的方式,初始病患从无到有的过程难以预测,因此采取忽略的策略)
准备好所需变量后开始迭代。
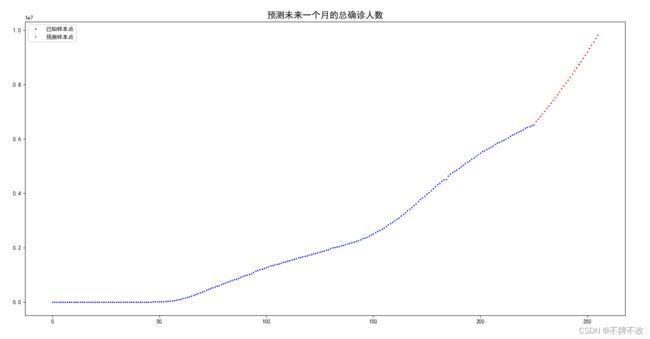
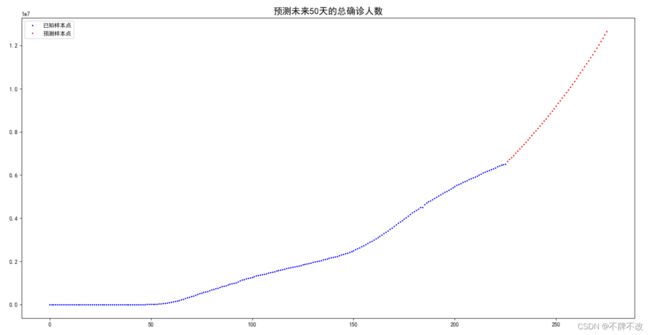
得到最终预测未来一个月总确诊数量的曲线如下:
结果分析
由于摆脱了对”新增数量“的依赖,且将当天的”总确诊数量“与前五天的”总确诊数量“相关联,该模型可以预测未来多个月的总确诊数量的变化。
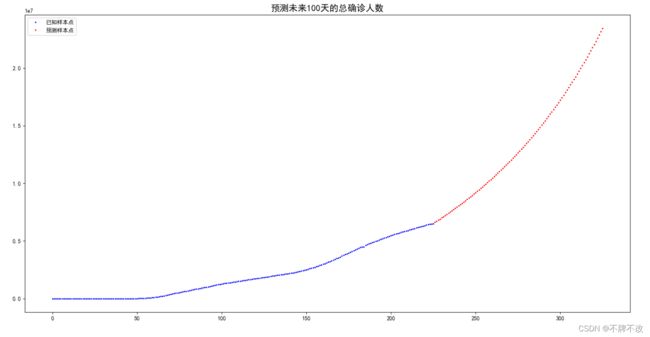
预测未来100天的总确诊人数变化:
从图中可以看出该模型具有一定的合理性。
总结展望
优势:
相比于上周的模型,本次实验模型的建立从实际情况出发,“总确诊数量”本质上受“现存确诊数量”的影响,又考虑到前若干天“现存确诊数量”对当天“现存确诊数的量”的影响,最终建立了比较合理的模型。
本次代码比上周实验更加简短,更加方便理解。
局限性:
未充分考虑自然环境和科学技术等因素对数量的影响,只在对死亡率和治愈率的预测时简单涉及“科学技术的进步”会使得死亡率持续下降并最终趋于平缓,而治愈率也会不断攀升并趋于平缓(考虑到科学技术仍存在一定的发展空间,忽略趋于平缓的情况)
附录(代码)
import numpy as np
from numpy import *
from scipy import linalg
from scipy.optimize import curve_fit
from xlrd import open_workbook
import matplotlib.pyplot as plt
from datetime import datetime
# 获取列的函数
def getcolumn(table, i):
res = []
for val in table.col_values(i):
if i == 0:
if len(val) != 2:
date = val.split('.')
res.append(datetime(int(date[0]), int(date[1]), int(date[2])))
else:
try:
res.append(int(val))
except:
continue
return res
#标准线性回归函数
def standRegres(xArr, yArr):
xMat = mat(xArr)
yMat = mat(yArr).T
xTx = xMat.T * xMat
#判断行列式为零,则无法求逆
if linalg.det(xTx) == 0:
print('the matrix is singular, cannot do inverse')
return
ws = (xTx).I * (xMat.T*yMat)
return ws
# 根据拟合出的系数计算对应的y值
def linearFunc(ws, xArr):
ws = [item[0] for item in ws.tolist()]
return sum(np.array(ws[:5])*np.array(xArr)) + ws[-1:][0]
# 指数函数,用于拟合变化率
def ExponentialFunction(x, a, b, c):
return a * np.exp(-b * x) + c
# 划分数据集的函数
def splitDataset(x, y, test_size = 0.2):
num = len(y)
train_number = round(num*(1-test_size))
X_train = x[0:train_number]
Y_train = y[0:train_number]
X_test = x[train_number:]
Y_test = y[train_number:]
return X_train, X_test, Y_train, Y_test
# 获取数据
original_data = open_workbook(r'C:\Users\23343\Desktop\test.xlsx') # 打开文件
table = original_data.sheets()[0]
col1 = getcolumn(table, 1)
col2 = getcolumn(table, 2)
col3 = getcolumn(table, 3)
col4 = getcolumn(table, 4)
alive = (array(col2) - array(col3) - array(col4)).tolist()
quantity = len(col1)
index = range(quantity)
# 回归获取系数(对现存确诊进行预测)
xArr = [alive[i:i+5] for i in range(quantity-5)] # 每五个划分为一个列表(注意最后一组没有对应的y,因此range(quantity-5),而不是range(quantity-4))
xArr = append(xArr, ones((len(xArr), 1)), axis=1).tolist() # 为每个项添加 1
xArr = [[int(i) for i in item] for item in xArr] # 将浮点转为整数
yArr = [alive[i] for i in range(5, quantity)]
X_train, X_test, Y_train, Y_test = splitDataset(xArr, yArr, 0.23) # 划分数据集
ws = standRegres(X_train, Y_train) # 系数
print(ws)
# """
# 绘图显示效果
xMat = mat(xArr)
yMat = mat(yArr)
yHat = xMat*ws
plt.plot(index[5:len(Y_train)+5], Y_train, 'b*', markersize = '2')
plt.plot(index[len(Y_train)+5:], Y_test, 'r*', markersize = '2')
plt.plot(index[5:], yHat)
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
plt.legend(["训练集", "测试集", "线性回归"])
plt.title("训练与测试", fontsize = '16')
plt.show()
# """
# 设置预测的天数,于此修改,下面预测的天数都会修改
predict_days = 30
# -------- 治愈率预测 --------
rate_recovery = [(col3[i + 1] - col3[i]) / alive[i] for i in range(len(col3)-1)]
rate_recovery = np.array(rate_recovery) # 治愈率 = (当日治愈总数 - 前日治愈总数)/前日现存确诊人数
idx_notzero = np.where((rate_recovery != 0) & (rate_recovery < 0.075))[0] # 去掉前面误差较大的点(最开始治愈率低是因为未找到治愈方式)
idx_notzero = [idx_notzero[i] for i in range(0, len(idx_notzero))]
rate_recovery_discretization = [rate_recovery[i] for i in idx_notzero]
popt, pcov = curve_fit(ExponentialFunction, idx_notzero, rate_recovery_discretization, bounds=([0, -1, 0], [np.inf, 0, 50])) # 指数函数拟合
predict_recovery_rate = ExponentialFunction(range(len(index), len(index)+predict_days), popt[0], popt[1], popt[2]) # 预测未来一个月的治愈率
# -------- 死亡率预测 --------
rate_death = [(col4[i + 1] - col4[i]) / alive[i] for i in range(len(col4) - 1)]
rate_death = np.array(rate_death) # 死亡率 = (当日死亡总数 - 前日死亡总数)/前日现存确诊人数
idx_notzero = np.where((rate_death != 0))[0] # 去掉前面误差较大的点
idx_notzero = [idx_notzero[i] for i in range(0, len(idx_notzero))]
rate_death_discretization = [rate_death[i] for i in idx_notzero]
popt, pcov = curve_fit(ExponentialFunction, idx_notzero, rate_death_discretization, bounds=([0, 0, 0], [np.inf, 1, 10])) # 指数函数拟合
predict_death_rate = ExponentialFunction(range(len(index), len(index)+predict_days), popt[0], popt[1], popt[2]) # 预测未来一个月的死亡率
# 变量准备
allpatients = col2.copy()
currentpatients = alive.copy()
predict_allpatients = []
predict_currentpatients = []
predict_recovery_rate
predict_death_rate
predict_recovery = col3[-1:]
predict_death = col4[-1:]
# 开始迭代
for i in range(predict_days):
predict_recovery.append(predict_recovery[i] + predict_recovery_rate[i] * alive[-1:][0])
predict_death.append(predict_death[i] + predict_death_rate[i] * alive[-1:][0])
currentpatients.append(linearFunc(ws, currentpatients[-5:]))
predict_currentpatients.append(currentpatients[-1:][0])
allpatients.append(currentpatients[-1:][0] + predict_recovery[-1:][0] + predict_death[-1:][0])
predict_allpatients.append(allpatients[-1:][0])
plt.plot(range(quantity), allpatients[:-predict_days], 'b*', markersize = '2')
plt.plot(range(quantity, quantity+predict_days), predict_allpatients, 'r*', markersize = '2')
plt.legend(["已知样本点", "预测样本点"])
plt.title("预测未来" + str(predict_days) + "天的总确诊人数", fontsize = '16')
plt.show()