Deeplung代码复现(二)
文章目录
-
- Deeplung代码复现(二)
-
- 一、训练
- 二、测试
-
- a、生成测试结果
- b、测试结果可视化
- 三、评估
-
- a、生成测试结果的最大FROC及对应epoch
- b、获得召回率并画出FROC曲线
Deeplung代码复现(二)
上一部分主要记录了环境配置,预处理和结节可视化,这一部分主要记录训练、测试和评估。
一、训练
作者采用十折交叉验证的方法进行训练。Luna16数据集一共subset0-subset9十个子集,使用9个训练一个测试。我的Epochs设为100。首先需要修改config_training0.py ~ config_training9.py中的训练集测试集路径。

以config_training0.py为例:我使用subset1 ~ subset9为训练集,以subset0为验证测试集,生成结果的存储文件名 retrft96X 中的X就表示使用哪个subsetX来测试。
config = {'train_data_path':['/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset9/',
'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset1/',
'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset2/',
'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset3/',
'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset4/',
'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset5/',
'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset6/',
'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset7/',
'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset8/'],
'val_data_path':['/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset0/'],
'test_data_path':['/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset0/'],
'train_preprocess_result_path':'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/preprocess/',
'val_preprocess_result_path':'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/preprocess/',
'test_preprocess_result_path':'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/preprocess/',
# 'train_bbox_path':'/media/data1/wentao/tianchi/bbox/train/',
# 'val_bbox_path':'/media/data1/wentao/tianchi/val/',
# 'test_bbox_path':'/media/data1/wentao/tianchi/test/',
'train_annos_path':'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/annotations.csv',
'val_annos_path':'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/annotations.csv',
'test_annos_path':'/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/annotations.csv',
'black_list':[],
'preprocessing_backend':'python',
}
通过下边语句来进行训练:
python main.py --model dpn3d26 -b 8 --save-dir dpn3d26/retrft960/ --epochs 100 --config config_training0
表示:model我使用dpn网络;batch-size设置为8(因为默认的batch-size=16,作者是是用来多块gpu进行训练的,而我只有一块,所以要设置小一点);save-dir是权重存储路径;epochs设为100;参数使用config_training0中设置的。
当更换训练集和测试集时,直接将 /retrft960/ 中的0换成你想用测试集,并且把–config config_training0换成对应的参数文件就可以啦
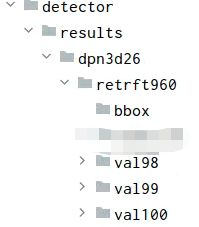
以下为我以subset0为测试集生成的训练权重结果:在/results/dpn3d26/retrft960/路径下生成一系列.ckpt模型参数
 然后同样的方式,把每个subsetX都当作一次测试集,剩下的当作训练集,一共训练10次。
然后同样的方式,把每个subsetX都当作一次测试集,剩下的当作训练集,一共训练10次。
二、测试
a、生成测试结果
1、完成训练后就要进行测试,测试的代码是:
python main.py --model dpn3d26 -b 1 --resume results/dpn3d26/retrft960/100.ckpt --test 1 --save-dir dpn3d26/retrft960/ --config config_training0
以上代码有两个地方需要解释:
- –resume与–save-dir路径中的retrft960,和–config中的config_training0,都表示测试集为subset0,在选用不同的模型进行测试时,修改此处的数字(0-9)就可以了。
- 100.ckpt表示使用第100个epoch生成的权重,叶可以改成其他epoch下训练的到的模型来训练,例如099.ckpt
2、在生成结果后使用如下代码来创建储存你测试结果的文件夹val100,100就表示你是使用第100轮epoch来命名的。
mkdir results/dpn3d26/retrft960/val100
3、然后使用下列语句将测试结果全部移到你创建的val100文件夹下。
mv results/dpn3d26/retrft960/bbox/*.npy results/dpn3d26/retrft960/val100
这样对于一个epoch的测试就完成了,测试结果也保存在了相应的文件夹下,以便评估使用。
下面是我使用subset0做测试集的模型下选择epoch=98,99,100的权重来测试的结果:测试结果保存在了val98、val99、val100三个文件里。
b、测试结果可视化
预测结果会为每个CT产生两个文件:_lbb.npy和_pbb.npy
| 文件 | 含义 |
|---|---|
| _lbb.npy | 从label中获得的,结节标签,是体素坐标(z,y,x,d) |
| _pbb.npy | 在阈值下(sigmod前)所产生的的所有预测结节(p,z,y,x,d) |
因为预测要做的就是把所有结节都找到,无论良性还是恶性,所以本着尽量不丢的原则阈值取小一点得到更多的可能结节。可视化的时候挑一部分进行。
从一个CT的_pbb.npy中读取几个预测结节(因为_pbb.npy中预测的结节已经转换为体素坐标,所以直接读出结节的坐标和所在第几个切片、直径就可以画图,不需要在转换坐标系),全部画出来就会发现有的是很接近通过标签画出来的框的。
以1.3.6.1.4.1.14519.5.2.1.6279.6001.108197895896446896160048741492为例:
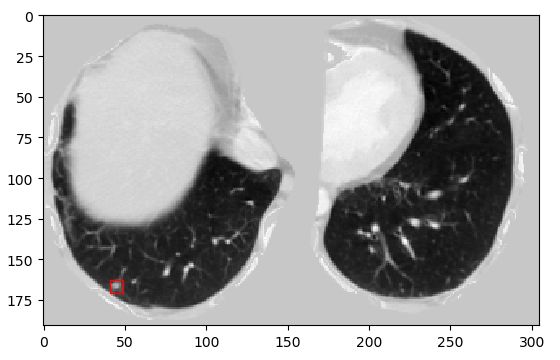
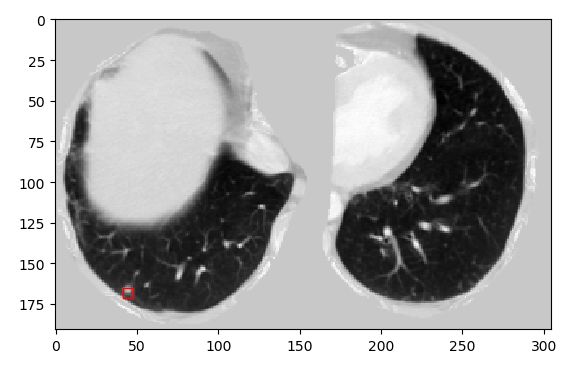
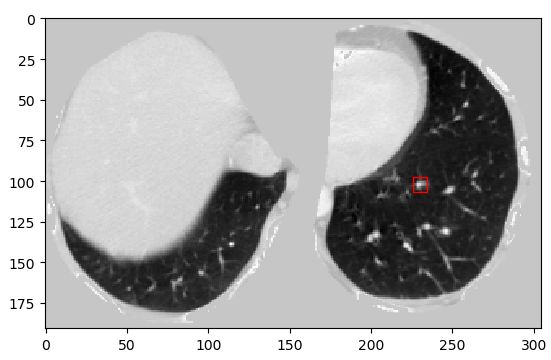
这两张都是从该CT的_pbb.npy中读出的信息所画的框,可以看出第一张基本接近真实标签中的切片数和位置,第二章略有偏差,单页在真实标签结节附近,第三张就是检测到的不在注释中的结节。也可以体现出“尽量不丢”的原则。
三、评估
主要使用代码中的frocwrtdetpepchluna16.py和noduleCADEvaluationLUNA16compare.py。
- frocwrtdetpepchluna16.py用于评估测试的结果,计算最大的FROC值,并且获得最大FROC值对应哪个epoch(你所测试的)。
- noduleCADEvaluationLUNA16.py用来获得召回率并画出FROC曲线。
a、生成测试结果的最大FROC及对应epoch
1、首先要把数据集中的标注信息annotations.csv、annotations_excluded.csv、seriesuids.csv分别按照不同的折自己提取好,做成对应的csv文件
2、在frocwrtdetpepchluna16.py中修改一部分路径:
fold = 0
annotations_filename = '/home/dlut/cfr/lung-canaer/DeepLung-master/evaluationScript/10FoldCsvFiles/annotation0.csv' # 这个路径是填写你需要评估的那一折文件的准标注信息,需要自己从annotations中提取对应折的标注信息,做成一个csv文件
annotations_excluded_filename = '/home/dlut/cfr/lung-canaer/DeepLung-master/evaluationScript/10FoldCsvFiles/annotations_excluded0.csv' # 这个路径同上,也需要自己从annotations_excluded中提取对应折的标注信息,做成一个csv文件
seriesuids_filename = '/home/dlut/cfr/lung-canaer/DeepLung-master/evaluationScript/10FoldCsvFiles/seriesuids0.csv' # 这个路径是需要自己从seriesuids中提取对应折的信息,做成一个csv文件
results_path = '/home/dlut/cfr/lung-canaer/DeepLung-master/detector/results/dpn3d26/retrft960/val' # 这个填写你测试时生成的那些bbox文件夹的路径
sideinfopath = '/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/preprocess/subset0/' # 对应折预处理后的数据(如subset0)
datapath = '/home/dlut/cfr/lung-canaer/DeepLung-master/data/luna16/subset0/' # 对应折的原始数据(如subset0)
通过以下语句设置你要评估的epoch范围:我的是98-100
maxeps = 100
eps = range(98, maxeps+1, 1)
通过detp = [-1.5, -1]来设置你的评估阈值(因为再sigmod前所以是负数)
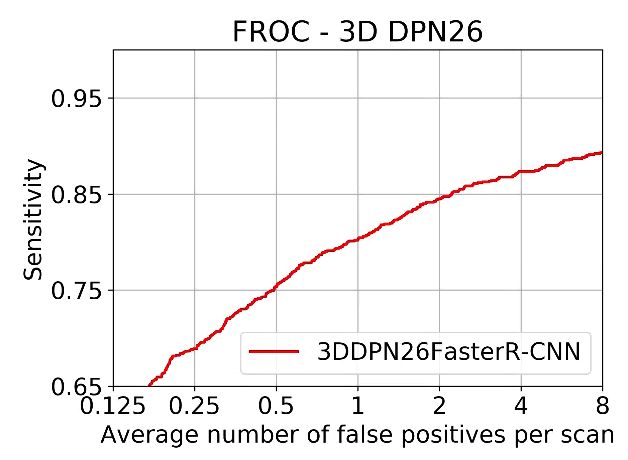
3、然后运行frocwrtdetpepchluna16.py就可以生成一折的评估结果。对每一折都运行一遍,便可以得到逝者的结果,我的结果如下:
b、获得召回率并画出FROC曲线
在noduleCADEvaluationLUNA16compare.py代码中修改一部分路径:
annotations_filename = '/home/dlut/cfr/lung-canaer/DeepLung-master/evaluationScript/annotations/annotations.csv'
annotations_excluded_filename = '/home/dlut/cfr/lung-canaer/DeepLung-master/evaluationScript/annotations/annotations_excluded.csv'
seriesuids_filename = '/home/dlut/cfr/lung-canaer/DeepLung-master/evaluationScript/annotations/seriesuids.csv'
results_filename = '/home/dlut/cfr/lung-canaer/DeepLung-master/evaluationScript/annotations/dpn3d26-100epochs/All-predanno-1.5.csv'
前三个就是数据集里的标签文件,最后一个All-predanno-1.5.csv是每一折的最大epoch下阈值为-1.5的predanno-1.5.csv全部汇集在一个csv文件里构成的,需要自己去操作。