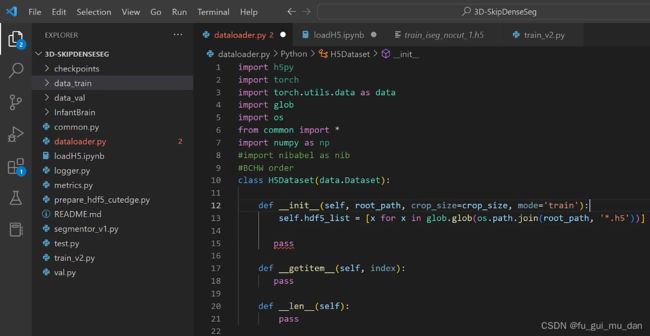
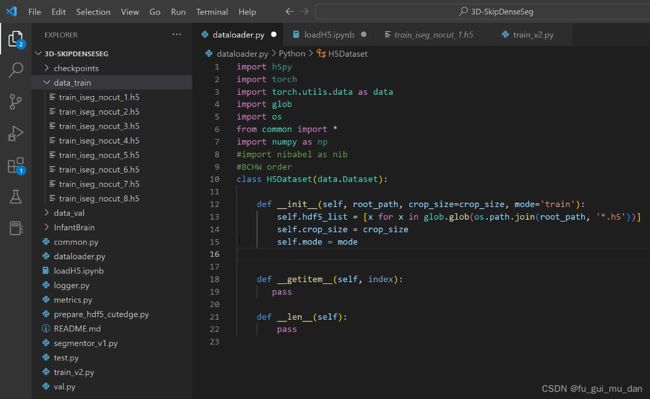
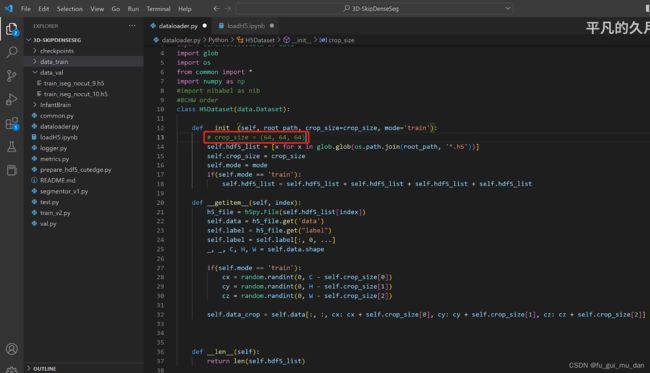
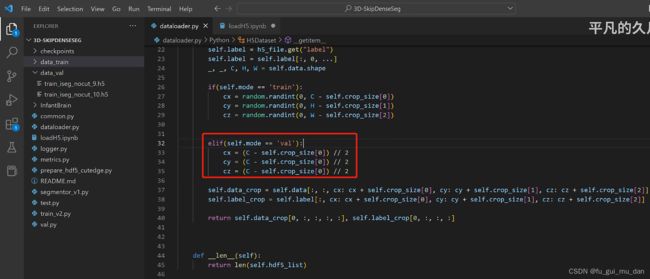
- 【python error】cannot import name ‘TorchDispatchMode‘ from ‘torch.utils._python_dispatch‘
Eternal-Student
JetsonOrinNXPythonpython开发语言
报错:cannotimportname‘TorchDispatchMode’from‘torch.utils._python_dispatch’(/home/nvidia/.conda/envs/pytorch/lib/python3.8/site-packages/torch/utils/_python_dispatch.py)File“/media/nvidia/Ubuntu/xxxxx/ev
- 全自动量化交易软件是否真的可靠?使用过程中有哪些潜在风险需要注意
财云量化
python炒股自动化量化交易程序化交易全自动量化交易软件可靠性潜在风险数据准确性股票量化接口股票API接口
炒股自动化:申请官方API接口,散户也可以python炒股自动化(0),申请券商API接口python炒股自动化(1),量化交易接口区别Python炒股自动化(2):获取股票实时数据和历史数据Python炒股自动化(3):分析取回的实时数据和历史数据Python炒股自动化(4):通过接口向交易所发送订单Python炒股自动化(5):通过接口查询订单,查询账户资产股票量化,Python炒股,CSDN
- 【测试语言篇四】Python进阶篇之json模块
m0_37135615
编程语言pythonphp开发语言
一、json模块介绍JSON(JavaScript对象表示法)是一种轻量级数据格式,用于数据交换。在Python中具有用于编码和解码JSON数据的内置json模块。只需导入它,就可以使用JSON数据了:importjsonJSON的一些优点:JSON作为“字节序列”存在,在我们需要通过网络传输(流)数据的情况下非常有用。与XML相比,JSON小得多,可转化为更快的数据传输和更好的体验。JSON非常
- 嵌入式AI必备技能2-模型的压缩与加速
奥德彪123
嵌入式AI人工智能嵌入式
嵌入式AI必备技能2-模型的压缩与加速引言随着嵌入式AI设备的广泛应用,模型的计算效率和存储需求成为核心挑战。由于嵌入式系统通常资源受限,传统的深度学习模型往往难以直接部署。因此,模型压缩和加速技术应运而生,旨在减少计算量、降低存储需求,同时尽可能保持模型的准确性。本文介绍几种常见的模型压缩与加速方法,包括剪枝、低秩分解、量化、权值共享、知识蒸馏等,并探讨如何综合应用这些技术来优化AI模型。1.常
- DeepSeek API 客户端使用文档
老大白菜
python人工智能数据库
1.简介deep.py是一个用于与DeepSeekAPI交互的Python客户端封装。它提供了简单易用的接口,支持对话历史管理、日志记录等功能,使得与DeepSeekAPI的交互更加便捷和可靠。2.功能特点简单的接口设计自动管理对话历史完整的日志记录灵活的配置选项异常处理机制3.安装依赖pipinstallopenai4.配置环境在项目根目录创建.env文件:#WindowssetDEEPSEEK
- 信息检索系统评估指标的层级分析:从单点精确度到整体性能度量
人工智能深度学习llm检索系统
在构建搜索引擎系统时,有效的评估机制是保证系统质量的关键环节。当用户输入查询词如"machinelearningtutorialspython",系统返回结果列表后,如何客观评估这些结果的相关性和有效性?这正是信息检索评估指标的核心价值所在。分析用户与搜索引擎的交互模式,我们可以观察到以下行为特征:用户主要关注结果列表的前几项对顶部结果的关注度显著高于底部结果用户基于多次搜索体验形成对搜索系统整体
- python系列【仅供参考】:python tornado 集成redis消息订阅的异步任务之后tornado主程序无法启动,解决方案
坦笑&&life
#pythonpythontornadoredis
pythontornado集成redis消息订阅的异步任务之后tornado主程序无法启动,解决方案pythontornado集成redis消息订阅的异步任务之后tornado主程序无法启动,解决方案封装redis异步类pythontornado集成redis消息订阅的异步任务之后tornado主程序无法启动,解决方案封装redis异步类sys_redis_helper.pyimportredis
- Python通过SSH隧道访问数据库
Java菜鸟在北京
pythonsshtunnelparamikoSSH隧道访问数据库
本文介绍通过sshtunnel类库建立SSH隧道,使用paramiko通过SSH来访问数据库。实现了两种建立SSH方式:公私钥验证、密码验证。公私钥可读本地,也可读取AwsS3上的私钥文件。本质上就是在本机建立SSH隧道,然后将访问DB转发到本地SSH内去访问数据库。简单易懂,上代码:fromsshtunnelimportSSHTunnelForwarderfromsqlalchemyimport
- 用Python写一个天气预报小程序
穿梭的编织者
Python脚本python小程序
一、界面效果二、完整代码importtkinterastkfromtkinterimportttkimportrequestsimportjsonfromdatetimeimportdatetimefromPILimportImage,ImageTkimportiofromttkbootstrapimportStyleclassWeatherApp:def__init__(self,root):s
- Python写一个脚本——30行代码——1秒实现PDF任意页码拆分
穿梭的编织者
Python精选pdfpython
一、引入库importosfromPyPDF2importPdfReader,PdfWriter二、定义拆分方法defsplit_pdf(input_path,output_dir,ranges):ifnotos.path.exists(output_dir):os.makedirs(output_dir)withopen(input_path,'rb')asfile:pdf=PdfReader(
- python手写kmeans算法
菜鸟懿
机器学习聚类算法python
kmean聚类是最基础和常见的算法,工程上使用比较常见,spark,sklearn都有实现,本文手写实现kmeans#!/usr/bin/pythonimportsysimportrandomimportmathdefcreate_rand_points(max_x,max_y,count):"""Createcountpoints(0-x),(0-y)."""points=[]foriinran
- Python 科学计算与机器学习入门:NumPy + Scikit-Learn 实战指南
吴师兄大模型
pythonnumpyscikit-learn人工智能开发语言机器学习编程
Langchain系列文章目录01-玩转LangChain:从模型调用到Prompt模板与输出解析的完整指南02-玩转LangChainMemory模块:四种记忆类型详解及应用场景全覆盖03-全面掌握LangChain:从核心链条构建到动态任务分配的实战指南04-玩转LangChain:从文档加载到高效问答系统构建的全程实战05-玩转LangChain:深度评估问答系统的三种高效方法(示例生成、手
- python start函数_Python中10个常用的内置函数
半残大叔霁天
pythonstart函数
大家好,我是小张在3.8版本中,Python解释器有近69个内置函数可供使用,有了它们能极大地提高编码效率,数量虽然不少,但在日常搬砖中只用到其中一部分,根据使用频率和用法,这里列出来几个本人认为不错的内置函数,结合一些例子介绍给大家complex()返回一个形如a+bj的复数,传入参数分为三种情况:参数为空时,返回0j参数为字符串时,将字符串表达式解释为复数形式并返回参数为两个整数(a,b)时,
- cv君独家视角 | AI内幕系列七:EfficientViT模型:基于多尺度线性注意力模块,实现高效的高分辨率密集预测
cv君
cv君独家视角AI内幕系列原创项目级实战项目深度学习与计算机视觉精品1024程序员节EfficientViT高分辨率密集预测任务高分辨率视觉模型Transformer人工智能计算机视觉
专题概况cv君独家视角|AI内幕系列是一个专注于人工智能领域的深度专题,旨在为读者揭开AI所有领域技术的神秘面纱,展示其背后的科学原理和实际应用。通过一系列精心策划的文章,我们将带您深入了解AI的各个领域,从计算机视觉到文本语音等多模态领域,从基础理论到前沿技术,从行业应用到未来趋势。无论您是AI领域的工程师或者专家,还是对这一领域充满好奇的读者,这个系列都将为您提供高价值的见解和启发,为您带来横
- 一个完整的python webSockets游戏服务器,每100ms接收并广播玩家位置
小宝哥Code
Python基础及AI开发python游戏服务器
PythonWebSockets游戏服务器下面是一个完整的PythonWebSockets游戏服务器实现,它每100ms接收并广播玩家位置信息。这个服务器使用websockets和asyncio库来处理WebSocket连接和异步操作。完整代码#!/usr/bin/envpython3"""实时游戏位置广播服务器每100ms接收玩家位置并广播给所有连接的客户端"""importasyncioimp
- 32路模拟采集PCI总线带DIO用什么采集卡
阿尔泰1999
数据分析嵌入式硬件科技
北京阿尔泰科技PCI5659是一-款多功能数据采集卡,具有32路12位100K采集频率,AD带16K字FIFO缓存,保证数据的连续性,并带16路可设方向的DIO功能。产品支持阿尔泰科技最新的ART-DAQ数据管理软件,提供QT、PYTHON、LABVIEW、VC、VB、VB.NET、C#等例子程序。模拟量输入通道数32路精度12位*大采样频率100KsPs多通道采样速度各通道*大采样频率/设置的采
- Python 爬虫实战:艺术品市场趋势分析与交易平台数据抓取
西攻城狮北
python爬虫开发语言
一、引言在当今数字化时代,艺术品市场正经历着前所未有的变革。随着互联网技术的飞速发展,越来越多的艺术品交易转移到了线上平台,这为我们提供了海量的数据资源。通过Python爬虫技术,我们可以抓取艺术品交易平台上的数据,进而分析艺术品市场的趋势,为投资者、收藏家以及艺术爱好者提供有价值的参考。本文将带领读者深入探索Python爬虫在艺术品市场的应用。从爬虫的基本原理到实际代码实现,再到数据的清洗、分析
- 【2025年饿了么春招-3月14日-第二题(200分)- 小红的排列构造】(题目+思路+Java&C++&Python解析+在线测试)
塔子哥学算法
javac++python算法数据结构饿了么
题目内容小红希望你构造一个长度为nnn的排列,满足∑i=1n∗i\sum_{i
- 贪心算法在背包问题上的运用(Python)
MATLAB卡尔曼
智能算法的MATLAB实现贪心算法python算法
背包问题有n个物品,它们有各自的体积和价值,现有给定容量的背包,如何让背包里装入的物品具有最大的价值总和?这就是典型的背包问题(又称为0-1背包问题),也是具体的、没有经过任何延伸的背包问题模型。背包问题的传统求解方法较为复杂,现定义有一个可以载重为8kg的背包,另外还有4个物品,物品的价值和质量数据如下表,不考虑背包的容量。4个物品的总质量大于8kg,所以要想在有限载重的背包携带更多质量的物品,
- NVIDIA显卡型号有哪些?怎么知道自己电脑的型号?
可靠的豆包蟹同志
杂烩积累经验分享
NVIDIA显卡型号显卡分N卡和A卡,这个N卡指的是英伟达(NVIDIA),A卡之前是ATI(后来被AMD收购),现在的A卡指的就是AMD显卡。如果是为了玩游戏或者是学深度学习,选显卡肯定是要选N卡,因为A卡对于游戏优化的没有N卡好。(1)图中的GTX表示是英伟达的一个系列名称,全称叫GeForceGTX,GTX定位高端显卡系列,从低到高排名:GS/GT/GTS/GTX/RTX/Ultra,从20
- 英伟达系列显卡大解析B100、H200、L40S、A100
2301_78234743
java
家里有了变故。。。快手数分秋招一面面经我发现算法岗也不很难进啊(深度学习)算法想转数开…Java零基础校招学习路线突击版(吐血整理)等的花都谢了的华子最后给开了22k,武汉,应该是14a。不过在这几个月里我坚定了搞几年快钱回家和np朋友因骂了hr,boos被封了哈哈哈在央企想被开除需要做什么?2024小米分布式存储研发急招华为2012被毁意向我发现算法岗也不很难进啊(深度学习)在央企想被开除需要做
- 接口测试中加密参数如何处理?
海姐软件测试
接口测试python开发语言测试工具职场和发展
1.加密类型及应对策略①对称加密(AES/DES)特点:加密解密使用同一密钥。处理方法:向开发获取密钥和加密算法(如AES-CBC、AES-ECB)。使用代码或工具解密响应数据:python复制fromCrypto.CipherimportAESimportbase64defdecrypt_aes(key,encrypted_data):cipher=AES.new(key.encode(),AE
- 用Python玩转Hyperledger:构建企业级区块链解决方案
Echo_Wish
Python!实战!perlpythonopencv人工智能
用Python玩转Hyperledger:构建企业级区块链解决方案大家好,我是Echo_Wish。在区块链技术的炙手可热中,“企业级区块链”俨然成为了下一个重磅关键词。相比于公有区块链,企业级区块链更注重隐私性、灵活性和高效性。而在这片“蓝海”中,Hyperledger项目无疑是企业级区块链解决方案的标杆。如果再搭配上Python这种“高效工具”,简直让人事半功倍!那么,如何将Python与Hyp
- eBest AI Hub全场景接入Deepseek
eBest数字化转型方案
人工智能
一、技术赋能,智创未来Deepseek的强大基因将为eBest产品注入新的活力即时智能响应:融合海量行业智慧与互联网搜索精华,提供秒级智能建议;多模态理解能力:突破界限,无缝融合文本、代码与图像理解,精准解析用户的需求;进化式深度学习:不断学习,持续进化,为用户提供日益完善、超越期待的服务体验。二、全场景赋能,体验再次跃升1.智能报表-数据洞察,指尖掌控升级后的智能报表功能,能够根据查询和检
- Android自动化测试工具
海棠如醉
web技术自动化运维
细解自动化测试工具Airtest-CSDN博客以下是几种常见的Android应用自动化测试工具:Appium:支持多种编程语言,如Java、Python、Ruby、JavaScript等。可以用于Web应用程序和原生应用程序的自动化测试,并支持iOS和Android平台。Espresso:由Google开发的AndroidUI测试框架,可用于测试应用程序的用户界面和与用户的交互。Espresso支
- Python说明
一一代码
python
Python的主要特点:1.**易读易写**:Python的语法简洁明了,代码可读性高。2.**跨平台**:Python可以在多种操作系统上运行,如Windows、macOS、Linux等。3.**丰富的库**:Python拥有庞大的标准库和第三方库,涵盖了从Web开发到数据科学的多个领域。4.**动态类型**:Python是动态类型语言,变量不需要显式声明类型。5.**解释型语言**:Pytho
- 使用 Excel 实现绩效看板的自动化
chenchihwen
自动化运维
引言在日常工作中,团队的绩效监控和管理是确保项目顺利进行的重要环节。然而,面临着以下问题:数据分散:系统中的数据难以汇总,缺乏一个宏观的团队执行情况视图。看板缺失:系统本身可能无法提供合适的Dashboard,导致数据分析困难。手动操作繁琐:数据采集、汇总和分析过程繁琐且耗时。本文将介绍如何利用免费的软件和工具(如Python、MySQL、Excel等)实现绩效看板的自动化。通过邮件自动推送和接收
- Python真经:代码修仙录
zzzzjflzdvkk
python开发语言青少年编程python真经
第一章:Python真经的起源在八十年代末,九十年代初,荷兰国境之内,有一位名为GuidovanRossum的修士,于国家数学与计算机科学研究所中,悟出了一门无上真经——Python。此真经融合了诸多上古大能的智慧结晶,如ABC、Modula-3、C、C++、Algol-68、SmallTalk、Unixshell等,终成一体,化为Python真经。Python真经自诞生之日起,便遵循GPL(GN
- python提示unmatched_Python自动化学习--异常提示
weixin_39933356
举例:打开一个不存在的文件时:>>open("abc.txt","r")会提示错误Traceback(mostrecentcalllast):File"D:/project1/test.py",line11,inopen("abc.txt","r")FileNotFoundError:[Errno2]Nosuchfileordirectory:'abc.txt'这时可以用try....except
- pyenv 管理多个 Python 版本(1)
数据科学工厂
python
引言你是否曾希望参与一个支持多个Python版本的项目,但又不知道如何轻松地测试所有这些版本?你是否对Python的最新版本感到好奇?或许你想尝试这些新功能,但又不想冒险破坏你的开发环境。幸运的是,如果你使用pyenv,管理多个Python版本并不复杂。本文[1]将向你展示如何高效地在项目上工作,同时减少因尝试使用正确版本的Python而产生的困扰。通过本文,你将学会:安装多个Python版本安装
- 矩阵求逆(JAVA)初等行变换
qiuwanchi
矩阵求逆(JAVA)
package gaodai.matrix;
import gaodai.determinant.DeterminantCalculation;
import java.util.ArrayList;
import java.util.List;
import java.util.Scanner;
/**
* 矩阵求逆(初等行变换)
* @author 邱万迟
*
- JDK timer
antlove
javajdkschedulecodetimer
1.java.util.Timer.schedule(TimerTask task, long delay):多长时间(毫秒)后执行任务
2.java.util.Timer.schedule(TimerTask task, Date time):设定某个时间执行任务
3.java.util.Timer.schedule(TimerTask task, long delay,longperiod
- JVM调优总结 -Xms -Xmx -Xmn -Xss
coder_xpf
jvm应用服务器
堆大小设置JVM 中最大堆大小有三方面限制:相关操作系统的数据模型(32-bt还是64-bit)限制;系统的可用虚拟内存限制;系统的可用物理内存限制。32位系统下,一般限制在1.5G~2G;64为操作系统对内存无限制。我在Windows Server 2003 系统,3.5G物理内存,JDK5.0下测试,最大可设置为1478m。
典型设置:
java -Xmx
- JDBC连接数据库
Array_06
jdbc
package Util;
import java.sql.Connection;
import java.sql.DriverManager;
import java.sql.ResultSet;
import java.sql.SQLException;
import java.sql.Statement;
public class JDBCUtil {
//完
- Unsupported major.minor version 51.0(jdk版本错误)
oloz
java
java.lang.UnsupportedClassVersionError: cn/support/cache/CacheType : Unsupported major.minor version 51.0 (unable to load class cn.support.cache.CacheType)
at org.apache.catalina.loader.WebappClassL
- 用多个线程处理1个List集合
362217990
多线程threadlist集合
昨天发了一个提问,启动5个线程将一个List中的内容,然后将5个线程的内容拼接起来,由于时间比较急迫,自己就写了一个Demo,希望对菜鸟有参考意义。。
import java.util.ArrayList;
import java.util.List;
import java.util.concurrent.CountDownLatch;
public c
- JSP简单访问数据库
香水浓
sqlmysqljsp
学习使用javaBean,代码很烂,仅为留个脚印
public class DBHelper {
private String driverName;
private String url;
private String user;
private String password;
private Connection connection;
privat
- Flex4中使用组件添加柱状图、饼状图等图表
AdyZhang
Flex
1.添加一个最简单的柱状图
? 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28
<?xml version=
"1.0"&n
- Android 5.0 - ProgressBar 进度条无法展示到按钮的前面
aijuans
android
在低于SDK < 21 的版本中,ProgressBar 可以展示到按钮前面,并且为之在按钮的中间,但是切换到android 5.0后进度条ProgressBar 展示顺序变化了,按钮再前面,ProgressBar 在后面了我的xml配置文件如下:
[html]
view plain
copy
<RelativeLa
- 查询汇总的sql
baalwolf
sql
select list.listname, list.createtime,listcount from dream_list as list , (select listid,count(listid) as listcount from dream_list_user group by listid order by count(
- Linux du命令和df命令区别
BigBird2012
linux
1,两者区别
du,disk usage,是通过搜索文件来计算每个文件的大小然后累加,du能看到的文件只是一些当前存在的,没有被删除的。他计算的大小就是当前他认为存在的所有文件大小的累加和。
- AngularJS中的$apply,用还是不用?
bijian1013
JavaScriptAngularJS$apply
在AngularJS开发中,何时应该调用$scope.$apply(),何时不应该调用。下面我们透彻地解释这个问题。
但是首先,让我们把$apply转换成一种简化的形式。
scope.$apply就像一个懒惰的工人。它需要按照命
- [Zookeeper学习笔记十]Zookeeper源代码分析之ClientCnxn数据序列化和反序列化
bit1129
zookeeper
ClientCnxn是Zookeeper客户端和Zookeeper服务器端进行通信和事件通知处理的主要类,它内部包含两个类,1. SendThread 2. EventThread, SendThread负责客户端和服务器端的数据通信,也包括事件信息的传输,EventThread主要在客户端回调注册的Watchers进行通知处理
ClientCnxn构造方法
&
- 【Java命令一】jmap
bit1129
Java命令
jmap命令的用法:
[hadoop@hadoop sbin]$ jmap
Usage:
jmap [option] <pid>
(to connect to running process)
jmap [option] <executable <core>
(to connect to a
- Apache 服务器安全防护及实战
ronin47
此文转自IBM.
Apache 服务简介
Web 服务器也称为 WWW 服务器或 HTTP 服务器 (HTTP Server),它是 Internet 上最常见也是使用最频繁的服务器之一,Web 服务器能够为用户提供网页浏览、论坛访问等等服务。
由于用户在通过 Web 浏览器访问信息资源的过程中,无须再关心一些技术性的细节,而且界面非常友好,因而 Web 在 Internet 上一推出就得到
- unity 3d实例化位置出现布置?
brotherlamp
unity教程unityunity资料unity视频unity自学
问:unity 3d实例化位置出现布置?
答:实例化的同时就可以指定被实例化的物体的位置,即 position
Instantiate (original : Object, position : Vector3, rotation : Quaternion) : Object
这样你不需要再用Transform.Position了,
如果你省略了第二个参数(
- 《重构,改善现有代码的设计》第八章 Duplicate Observed Data
bylijinnan
java重构
import java.awt.Color;
import java.awt.Container;
import java.awt.FlowLayout;
import java.awt.Label;
import java.awt.TextField;
import java.awt.event.FocusAdapter;
import java.awt.event.FocusE
- struts2更改struts.xml配置目录
chiangfai
struts.xml
struts2默认是读取classes目录下的配置文件,要更改配置文件目录,比如放在WEB-INF下,路径应该写成../struts.xml(非/WEB-INF/struts.xml)
web.xml文件修改如下:
<filter>
<filter-name>struts2</filter-name>
<filter-class&g
- redis做缓存时的一点优化
chenchao051
redishadooppipeline
最近集群上有个job,其中需要短时间内频繁访问缓存,大概7亿多次。我这边的缓存是使用redis来做的,问题就来了。
首先,redis中存的是普通kv,没有考虑使用hash等解结构,那么以为着这个job需要访问7亿多次redis,导致效率低,且出现很多redi
- mysql导出数据不输出标题行
daizj
mysql数据导出去掉第一行去掉标题
当想使用数据库中的某些数据,想将其导入到文件中,而想去掉第一行的标题是可以加上-N参数
如通过下面命令导出数据:
mysql -uuserName -ppasswd -hhost -Pport -Ddatabase -e " select * from tableName" > exportResult.txt
结果为:
studentid
- phpexcel导出excel表简单入门示例
dcj3sjt126com
PHPExcelphpexcel
先下载PHPEXCEL类文件,放在class目录下面,然后新建一个index.php文件,内容如下
<?php
error_reporting(E_ALL);
ini_set('display_errors', TRUE);
ini_set('display_startup_errors', TRUE);
if (PHP_SAPI == 'cli')
die('
- 爱情格言
dcj3sjt126com
格言
1) I love you not because of who you are, but because of who I am when I am with you. 我爱你,不是因为你是一个怎样的人,而是因为我喜欢与你在一起时的感觉。 2) No man or woman is worth your tears, and the one who is, won‘t
- 转 Activity 详解——Activity文档翻译
e200702084
androidUIsqlite配置管理网络应用
activity 展现在用户面前的经常是全屏窗口,你也可以将 activity 作为浮动窗口来使用(使用设置了 windowIsFloating 的主题),或者嵌入到其他的 activity (使用 ActivityGroup )中。 当用户离开 activity 时你可以在 onPause() 进行相应的操作 。更重要的是,用户做的任何改变都应该在该点上提交 ( 经常提交到 ContentPro
- win7安装MongoDB服务
geeksun
mongodb
1. 下载MongoDB的windows版本:mongodb-win32-x86_64-2008plus-ssl-3.0.4.zip,Linux版本也在这里下载,下载地址: http://www.mongodb.org/downloads
2. 解压MongoDB在D:\server\mongodb, 在D:\server\mongodb下创建d
- Javascript魔法方法:__defineGetter__,__defineSetter__
hongtoushizi
js
转载自: http://www.blackglory.me/javascript-magic-method-definegetter-definesetter/
在javascript的类中,可以用defineGetter和defineSetter_控制成员变量的Get和Set行为
例如,在一个图书类中,我们自动为Book加上书名符号:
function Book(name){
- 错误的日期格式可能导致走nginx proxy cache时不能进行304响应
jinnianshilongnian
cache
昨天在整合某些系统的nginx配置时,出现了当使用nginx cache时无法返回304响应的情况,出问题的响应头: Content-Type:text/html; charset=gb2312 Date:Mon, 05 Jan 2015 01:58:05 GMT Expires:Mon , 05 Jan 15 02:03:00 GMT Last-Modified:Mon, 05
- 数据源架构模式之行数据入口
home198979
PHP架构行数据入口
注:看不懂的请勿踩,此文章非针对java,java爱好者可直接略过。
一、概念
行数据入口(Row Data Gateway):充当数据源中单条记录入口的对象,每行一个实例。
二、简单实现行数据入口
为了方便理解,还是先简单实现:
<?php
/**
* 行数据入口类
*/
class OrderGateway {
/*定义元数
- Linux各个目录的作用及内容
pda158
linux脚本
1)根目录“/” 根目录位于目录结构的最顶层,用斜线(/)表示,类似于
Windows
操作系统的“C:\“,包含Fedora操作系统中所有的目录和文件。 2)/bin /bin 目录又称为二进制目录,包含了那些供系统管理员和普通用户使用的重要
linux命令的二进制映像。该目录存放的内容包括各种可执行文件,还有某些可执行文件的符号连接。常用的命令有:cp、d
- ubuntu12.04上编译openjdk7
ol_beta
HotSpotjvmjdkOpenJDK
获取源码
从openjdk代码仓库获取(比较慢)
安装mercurial Mercurial是一个版本管理工具。 sudo apt-get install mercurial
将以下内容添加到$HOME/.hgrc文件中,如果没有则自己创建一个: [extensions] forest=/home/lichengwu/hgforest-crew/forest.py fe
- 将数据库字段转换成设计文档所需的字段
vipbooks
设计模式工作正则表达式
哈哈,出差这么久终于回来了,回家的感觉真好!
PowerDesigner的物理数据库一出来,设计文档中要改的字段就多得不计其数,如果要把PowerDesigner中的字段一个个Copy到设计文档中,那将会是一件非常痛苦的事情。