简单的聚类算法——KMeans代码
简单来讲,聚类就是在还没有类别的情况下,将物体经过算法自动归为不同的类。而分类是已知类别的情况下,将物体分到不同的类中。这个类别就是标签,所以聚类也可以视作无监督分类算法,这个监督就是指有没有提前认为分好类别。
概述
KMeans算法也叫K均值算法,是最常用的聚类算法,主要思想是:在给定K值和随机初始K个中心点的情况下,把每个点(假设是二维数据)分到离其最近的类簇中心点所代表的类簇中,所有点分配完毕之后,根据一个类簇内的所有点重新计算该类簇的中心点(取平均值),然后再迭代的进行分配点和更新类簇中心点的步骤,直至类簇中心点的变化很小,或者达到指定的迭代次数,分类停止。
以二维空间为例
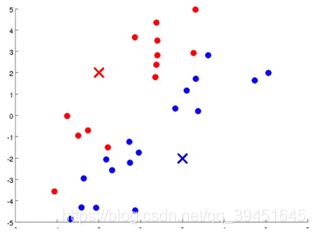
首先随机取两个点作为基准点(叉点),然后逐一计算每个点到这两个基准点的欧式距离(也可以有其他距离、比如余弦距离等),距离哪个基准点近,就将该点归到哪个类中。至此第一次迭代结束。
之后,分别从两部分所有点的坐标中取出平均值的坐标点,再次逐一计算所有点与这两个坐标点的欧式距离,距离哪个坐标距离小,就归到哪个类中。这时会有很多点改变所属类。至此第二次迭代更新结束。
之后不断重复该迭代更新过程,不断更新平均值的坐标点,不断更新所有点的所属类,直到所有点的类别不再改变,算法结束。
上代码
test.py文件
from numpy import *
import kmeans #文件名
from sklearn.datasets.samples_generator import make_blobs
#数据是聚类样本生成器随机生成的4簇二维点,共1000个
dataSet, _ = make_blobs(n_samples=1000,
n_features=2,
centers=4,
)
dataSet = mat(dataSet) #转换成矩阵方便操作
k = 4
#调用kmeans文件中的kmeans方法
centroids, clusterAssment = kmeans.kmeans(dataSet, k)
文件名kmeans.py
from numpy import *
import matplotlib.pyplot as plt
# 求这两个矩阵的距离
def euclDistance(vector1, vector2):
return sqrt(sum(power(vector2 - vector1, 2))) #求得欧氏距离
# 在样本集中随机选取k个样本点作为初始质心
def initCentroids(dataSet, k):
numSamples, dim = dataSet.shape # 获取矩阵的行数、列数。行数即为样本数量
centroids = zeros((k, dim)) #生成一个大小为k*2的零矩阵
for i in range(k): #随机取点
index = int(random.uniform(0, numSamples))
centroids[i, :] = dataSet[index, :]
return centroids
# dataSet为一个矩阵。由于数据是n个平面上的点,因此可以看做是n*2的矩阵
# k为将dataSet矩阵中的样本分成k个类
def kmeans(dataSet, k):
numSamples = dataSet.shape[0] #获取样本点数量
clusterAssment = mat(zeros((numSamples, 2))) # 得到一个N*2的零矩阵,记录点的聚类结果
clusterChanged = True #标记,如果每个点的所属类别不再变动,那么聚类已稳定
centroids = initCentroids(dataSet, k) # 在样本集中随机选取k个样本点作为初始质心
while clusterChanged:
clusterChanged = False
# 遍历每个样本点
for i in range(numSamples):
minDist = 100000.0
minIndex = 0
# 计算每个样本点与质点之间的距离,将其归内到距离最小的那一簇
for j in range(k):
distance = euclDistance(centroids[j, :], dataSet[i, :])
if distance < minDist: # 样本点与4个质心逐一比较,记录最小距离和坐标
minDist = distance
minIndex = j
# k个簇里面与第i个样本距离最小的簇标号和距离保存在clusterAssment中
# 若所有的样本不在变化,则退出while循环
if clusterAssment[i, 0] != minIndex:
clusterChanged = True
clusterAssment[i, :] = minIndex, minDist ** 2 # 两个**表示的是minDist的平方
# 更新每个类别的质心,质心为每个类中所有坐标的平均值
for j in range(k):
# 这句意思不明白的可以看文末链接
pointsInCluster = dataSet[nonzero(clusterAssment[:, 0].A == j)[0]]
# axis=0意思是矩阵列方向上求平均值,返回一个1*n的矩阵
centroids[j, :] = mean(pointsInCluster, axis=0)
return centroids, clusterAssment
#画出图像
def showCluster(dataSet , k, centroids, clusterAssment):
numSamples, dim = dataSet.shape
mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'sr', 'dr', ', 'pr']
if k > len(mark):
print("Sorry! Your k is too large! please contact wojiushimogui")
return 1
# draw all samples
for i in range(numSamples):
markIndex = int(clusterAssment[i, 0]) # 为样本指定颜色
plt.plot(dataSet[i, 0], dataSet[i, 1], mark[markIndex])
mark = ['Dr', 'Db', 'Dg', 'Dk', '^b', '+b', 'sb', 'db', ', 'pb']
# draw the centroids
for i in range(k):
plt.plot(centroids[i, 0], centroids[i, 1], mark[i], markersize=12)
plt.show()
运行结果
参考
https://blog.csdn.net/xinjieyuan/article/details/81477120
