模式识别和机器学习实战-降维算法(PCA)- Python实现 - 半导体数据进行降维处理和人脸检测
文章目录
- 前言
- 一、 降维算法
-
- 1.算法介绍
- 2.主成分分析PCA
- 3.在Numpy中实现PCA
- 二、利用PCA对半导体制造数据降维
- 三、实战
- 四、代码
-
- 1.主成分分析PCA
- 2.对半导体数据进行降维处理
- 3.人脸检测算法
前言
降维是指采用某种映射方法,将原高维空间中的数据点映射到低维度的空间中。通过降维可以减少 冗余信息所造成的误差,提高识别的精度,或者通过降维算法来寻找数据内部的本质结构特征。
先介绍和解释步骤,分段介绍;最后再给出完整源码,只需修改一下文件路径即可运行。
而具体的数据集文件和代码,在如下链接里:降维算法(PCA)- Python实现
一、 降维算法
1.算法介绍
降维是指采用某种映射方法,将原高维空间中的数据点映射到低维度的空间中。通过降维可以减少 冗余信息所造成的误差,提高识别的精度,或者通过降维算法来寻找数据内部的本质结构特征。
2.主成分分析PCA
PCA是最常用的线性降维方法,它的目标是通过某种线性投影,将高维的数据映射到低维的空间中表示,并期望在所投影的维度上数据的方差最大,以此使用较少的数据维度,同时保留住较多的原数据点的特性。
3.在Numpy中实现PCA
将数据转换成前N个主成分的伪代码大致如下:
1.去除平均值
2.计算协方差矩阵
3.计算协方差矩阵的特征值和特征向量
4.将特征值从大到小排序
5.保留最上面的N个特征向量
6.将数据转换到上述N个特征向量构建的新空间中
二、利用PCA对半导体制造数据降维
半导体制造数据很大,并且包含了590个特征。该数据包含很多的缺失值,这些缺失值是以NaN标识的,对于这些缺失值,我们用平均值来代替缺失值,平均值通过那些非NaN得到。
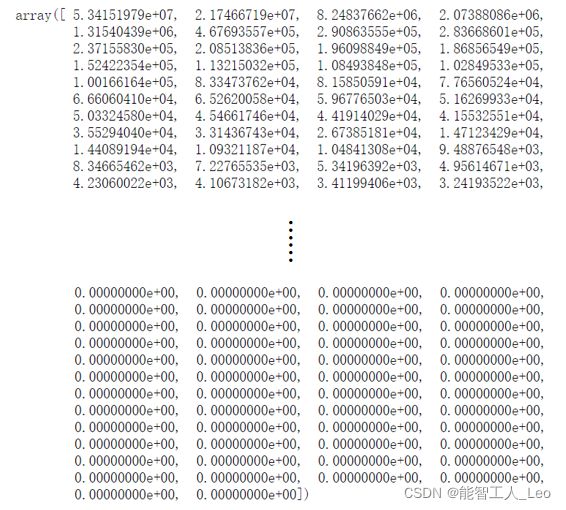
去除了所有NaN之后,接下来考虑在该数据集上应用PCA,首先确认所需特征和可以去除特征的数目。PCA会给出数据中所包含的信息量。从pca()函数中借用以下代码来观察一下特征值的结果。
特征值的结果如下所示:有超过20%的特征值都是0;最前面15个值的数量级大于100000
半导体数据中前7个主成分所占的方差百分比:
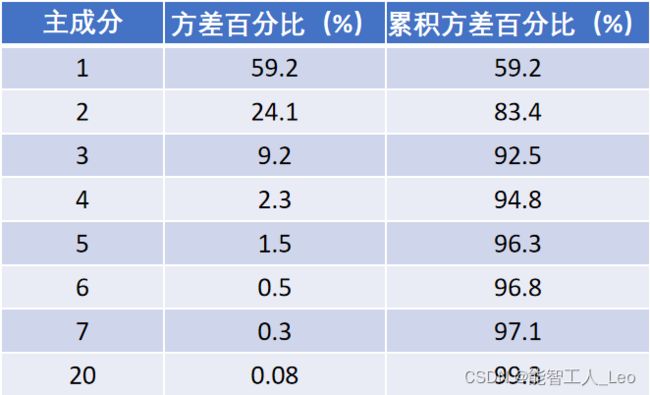
具体的主成分数目,必须通过在实验中取不同的值来确定。有效的主成分数目则取决于数据集和具体应用。
得到主成分数目之后将该属木输入到PCA算法中,降维之后的数据就可以在分类器中使用了
三、实战
1.根据之前的分析,确定半导体数据的主成分数目(可以对不同的主成分数目的降维效果进行分析),对半导体数据进行降维:
对主成分的方差占比分析,前7个主成分已经达到了97.12%,之后百分比递减,为方便计算只选取前7个降维特征。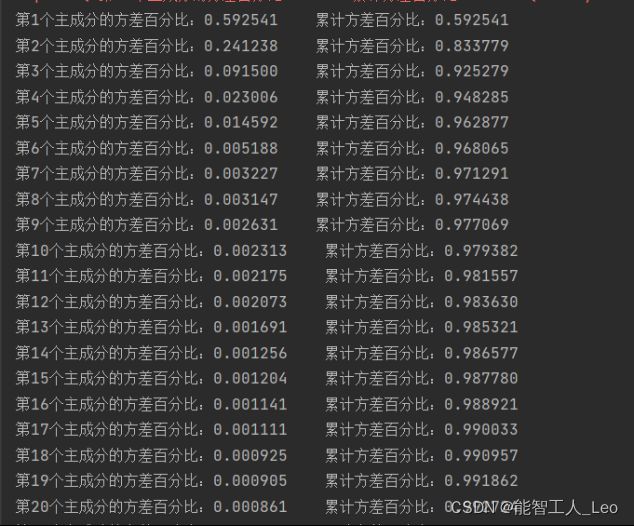
2.data-Recognition文件夹当中是yale人脸数据集(文件夹内有对于数据集的详细描述)利用主成分分析方法,选择合适的主成分数目对数据进行降维:
得到的最佳降维后数据集,如图为centerlight,取前8个主成分即可达到95%
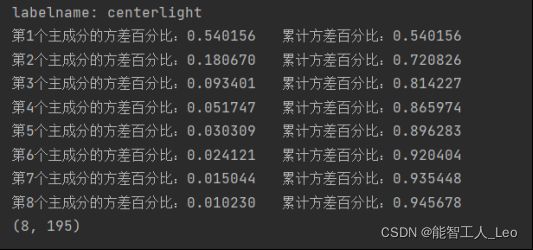
3.基于2的结果,针对data-Detection文件夹内的内容,设计一种人脸检测算法,请使用一个矩形(或正方形)来表示在给定的图像中检测到的人脸…
(利用opencv里的检测算法)
四、代码
1.主成分分析PCA
#导入数据
def loadDataSet(fileName, delim='\t'):
fr = open(fileName)
stringArr = [line.strip().split(delim) for line in fr.readlines()]
datArr = [list(map(float, line)) for line in stringArr]
return mat(datArr)
datamat = loadDataSet("testSet.txt")
#PCA实现
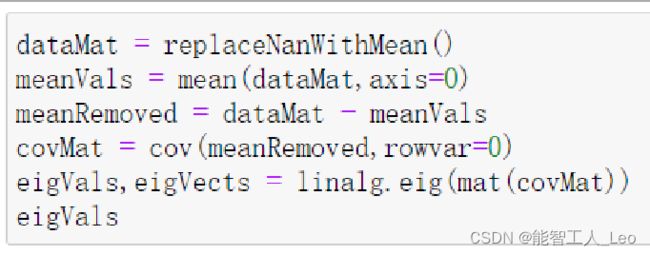
def pca(dataMat, topNfeat=9999999):
meanVals = mean(dataMat, axis=0) # 计算平均值
meanRemoved = dataMat - meanVals # 去中心化
covMat = cov(meanRemoved, rowvar=0) # 计算协方差矩阵
eigVals, eigVects = linalg.eig(mat(covMat)) # 得到所有的特征值eigVals和特征向量eigVects
eigValInd = argsort(eigVals) # 排序
eigValInd = eigValInd[:-(topNfeat + 1):-1] # 保留至想要的维度
redEigVects = eigVects[:, eigValInd] # ...从大到小排列特征向量,得到投影矩阵
lowDDataMat = meanRemoved * redEigVects # 投影到新的特征空间后的数据集
reconMat = (lowDDataMat * redEigVects.T) + meanVals
return lowDDataMat, reconMat
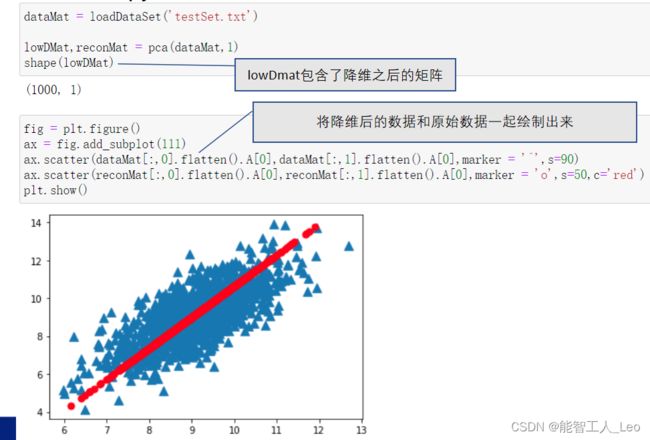
lowDDataMat, reconMat = pca(datamat, 1)
#可视化
fig = plt.figure()
ax = fig.add_subplot(111)
ax.scatter(datamat[:, 0].flatten().A[0], datamat[:, 1].flatten().A[0], marker='X', s=90, c='blue')
ax.scatter(reconMat[:, 0].flatten().A[0], reconMat[:, 1].flatten().A[0], marker='o', s=50, c='red')
plt.show()
2.对半导体数据进行降维处理
#对缺失值的处理,用未缺失样本的平均值代替
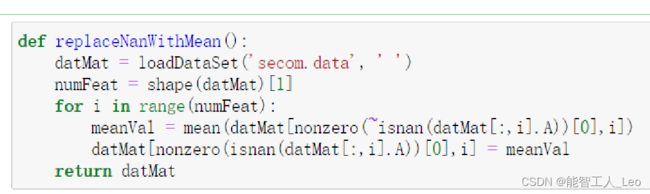
def replaceNaNWithMean():
datMat = loadDataSet('secom.data', ' ')
numFeat = shape(datMat)[1]
for i in range(numFeat):
meanVal = mean(datMat[nonzero(~isnan(datMat[:, i].A))[0], i])
datMat[nonzero(isnan(datMat[:, i].A))[0], i] = meanVal
return datMat
#用pca降维时,计算每个主成分的方差,获得贡献率
def PCA(dataMat, topNfeat=9999999):
meanVals = mean(dataMat, axis=0) # 计算平均值
meanRemoved = dataMat - meanVals # 去中心化
covMat = cov(meanRemoved, rowvar=0) # 计算协方差矩阵
eigVals, eigVects = linalg.eig(mat(covMat)) # 得到所有的特征值eigVals和特征向量eigVects
eigValInd = argsort(eigVals) # 排序
eigValInd = eigValInd[:-(topNfeat + 1):-1] # 保留至想要的维度
redEigVects = eigVects[:, eigValInd] # ...从大到小排列特征向量
lowDDataMat = meanRemoved * redEigVects # 投影到新的特征空间后的数据集
reconMat = (lowDDataMat * redEigVects.T) + meanVals
tot = sum(eigVals) # 计算特征值的和
var_exp = [(i / tot) for i in sorted(eigVals, reverse=True)] # 按照降序排列特征值,并计算贡献率
cum_var_exp = cumsum(var_exp) # 累计贡献度
for i in range(len(var_exp)):
print("第%d个主成分的方差百分比:%f 累计方差百分比:%f" % (i + 1,
float(var_exp[i]),float(cum_var_exp[i]))) # 输出贡献度
return lowDDataMat, reconMat
#调用pca获得降维后的前七个主成分数据集
dataMat = replaceNaNWithMean()
lowDDataMat, reconMat = PCA(dataMat, 7) #前7个主成分所占的方差已经累计达97.1%
print(lowDDataMat) #得到降维后的数据集
print("数据集个数:%d"% shape(lowDDataMat)[0],"主成分个数:%d"% shape(lowDDataMat)[1])
3.人脸检测算法
from numpy import *
import cv2
import os
import matplotlib.pyplot as plt
import matplotlib.image as mpimg
def loaddata():
data = []
label = ["centerlight", "glasses", "happy", "leftlight", "noglasses", "normal", "rightlight", "sad", "sleepy","surprised", "wink"]
for i in range(15):
data.append({})
for j in label:
h = str(i + 1)
if i < 9:
h = "0" + h
data[i][j] = mpimg.imread("project1-data-Recognition1//subject{}.{}.pgm".format(h, j))
return data, label # 数据集和标签
def PCA(dataMat, topNfeat=999999):
meanVals = mean(dataMat, axis=0) # 计算平均值
meanRemoved = dataMat - meanVals # 去中心化
covMat = cov(meanRemoved, rowvar=0) # 计算协方差矩阵
eigVals, eigVects = linalg.eig(mat(covMat)) # 得到所有的特征值eigVals和特征向量eigVects
eigValInd = argsort(eigVals) # 排序
eigValInd = eigValInd[:-(topNfeat + 1):-1] # 保留至想要的维度
redEigVects = eigVects[:, eigValInd] # ...从大到小排列特征向量
lowDDataMat = meanRemoved * redEigVects # 投影到新的特征空间后的数据集
reconMat = (lowDDataMat * redEigVects.T) + meanVals
tot = sum(eigVals) # 计算特征值的和
var_exp = [(i / tot) for i in sorted(eigVals, reverse=True)] # 按照降序排列特征值,并计算贡献率
cum_var_exp = cumsum(var_exp) # 累计贡献度
for i in range(len(var_exp)):
if float(cum_var_exp[i]) < 0.95:
print("第%d个主成分的方差百分比:%f 累计方差百分比:%f" % (i + 1, float(var_exp[i]), float(cum_var_exp[i]))) # 输出贡献度
else:
break
return lowDDataMat[:i], reconMat[:i] #返回我们需要的维度的数据集
datamat, label = loaddata()
def subjectDataMat(): # 得到所有数据降维后的数据集
subjectDataMat = []
for i in label:
print("\nlabelname: %s " % i)
lowDDataMat, reconMat = PCA(datamat[0][i])
subjectDataMat.append(lowDDataMat)
return subjectDataMat
def traingdata(N): # 获得训练集,N为输入的个数3,5,7...
traingdata=[]
for i in label:
for j in range(N): # 随机选择N个
num = random.randint(1, 11)
traingdata.append(datamat[0][i][num])
lowDtraingdata, recontraingdata = PCA(traingdata)
return lowDtraingdata
datamat, label = loaddata()
traingdata(3)