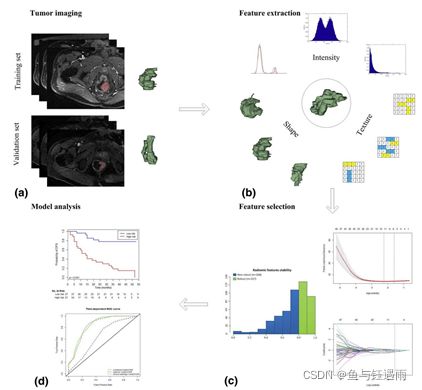
影像组学训练营 第三天(共三天)
文章目录
- 影像组学论文剖析
-
- 文章宏观分析
- 文章背景介绍
- 影像组学模型构建
- 高质量标准化MRI影像数据获取及分析
- 感兴趣体积VOI分割
- 图像预处理
- 特征提取
- 特征一致性评价
- 第七步 特征选择
- 数据库
-
- 一,数据库简介:
- 四,相关研究Research Activities
- 问答环节
-
- 1.老师好 现在有什么方法做影像组学于基因组学的联合分析么
- 老师,想做超声方向,请问怎么查找超声组学的相关文章,用什么关键词呢?
- 老师好 基因数据和影像学数据一定是得同样本么? 发高分,60例患者够么?
影像组学论文剖析
今天要分享2篇比较基础的影像组学文章
检验一下这三天的成果,看看结构中有哪些不变的东西,同时,学习一下文章的细节,那我们现在开始!
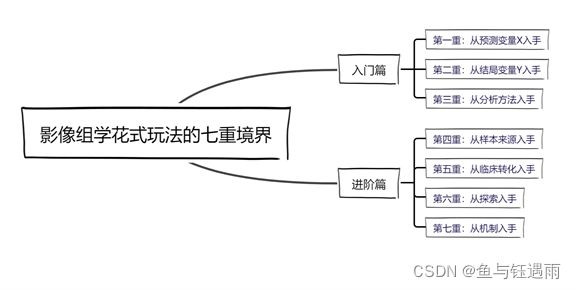
这张图大家应该不陌生,我们昨天已经讲过了影像组学7重境界的前三重。
文章宏观分析
但我们今天从最基础的开始,单预测变量X(单模态,单靶区)+单结局变量Y+单分析方法(单模型)套路的文章;
虽说是单预测变量X,但也是结合了临床指标的;
文章在18年发到5分,还是有很多可取之处,如果细节再做好一点,绝对不止5分;

我在过程中也会稍微提一些他没做好的细节,我们首先看题目。两个红线,给我们两个信息,研究的类型是“预后型研究”,人群为“局部晚期直肠癌LARC”,预测变量X是“影像组学标签”。
我们先从PICOS原则来对文章的宏观要素进行剖析;
当然PICOS原则对于预测模型,I干预和C对照就合为了预测模型这一项;也有把他俩合为一个叫Factor的说法;
研究对象(P):采用新辅助放化疗(nCRT)治疗后进行全直肠系膜切除术(TME)的局部晚期直肠癌(LARC)患者(108名纳入)—治疗方案统一,人群的同质性棒棒的;
预测模型(I/C):LASSO-cox回归模型构建的影像组学signature
结局指标(O):无病生存期DFS(生存终点,生存时间)
研究类型(S):回顾性队列研究
可能有同学对模型还比较陌生,我简单说下;其实也就是个数学公式,高端点可以叫算法,而预测模型就是通过已知的数据(预测变量X)来预测未知(结局变量Y);
临床预测模型的本质就是通过回归建模分析,定量的描述X对Y影响的程度有多大。常用的回归模型包括:多元线性回归(研究一个因变量和多个自变量之间的关系)、Logistic回归(研究终点结局出现与否)、Cox回归(研究生存结局+生存时间的影响)等。这也与我昨天埋下的伏笔有关;不知道大家能不能找到昨天我说过不同的结局变量Y决定研究方法不同类似的话;本文用了LASSO-Cox回归模型,听起来挺高级,其实就是在Cox回归前用LASSO回归在进行一次特征筛选;
我们再来说下这个O结局;有人知道DFS是什么嘛?无病生存期,主要针对手术患者;
这属于我们昨天提到的哪种结局?
① 辅助诊断(如肿瘤的良恶性判读和肿瘤术前分期等)
② 分子分型(如分子的高低表达等)
③ 近期疗效(如判断EGFR突变患者使用TKI靶向治疗的耐药评估或新辅助放化疗患者的病理学完全缓解评估等)
④ 远期疗效预测(如对原发性肝癌术后复发预测及对鼻咽癌患者的无进展生存期预测等)
⑤ 毒副反应评估(如放射性肺炎、脑损伤等)
自己研究时也要先想自己结局的类别以及资料类型;
而且哦,需要注意的是,DFS,OS,PFS等结局不同文章定义是不同的,这也是需要我们重点关注的。比如,DFS的定义:经过治疗后未发现肿瘤,结局指标为疾病复发或死亡,不关心死亡原因,只要有疾病的复发、转移、死亡,均定义为结局事件的发生。本文将DFS定义为 TME 手术和疾病进展之间的间隔,包括局部肿瘤复发、远处转移和任何原因导致的死亡,或最后一次随访的日期(失访)。其中局部复发定义为盆腔复发,远处转移定义为局部与区域以外的新增病灶。所有局部复发和远处转移病例均由多学科团队根据临床检查、血清癌胚抗原水平、胸部和腹腔 CT 和/或腹腔 MRI、内窥镜检查和活检诊断。
大体来看,定义还是比较明确的。 值得大家学习!同样我们需要明确研究的设备和研究目的;影像种类: 3T磁场强度的LAVA动态增强扫描序列的横断面MR影像
研究目的:比较影像组学特征标签和LARC临床预后因子对目标患者DFS的预测能力;
至此,文章宏观要素分析部分结束了
文章背景介绍
全直肠系膜切除术(TME)前进行新辅助放化疗(nCRT)目前被认为是局部晚期直肠癌(LARC)患者的标准联合治疗方式。但是,肿瘤的远端转移还是成为该治疗方案失败的主要原因。因此,识别影响生存和术前风险分层的不良预后特征,并对 nCRT 后发生不良后果的高风险个体进行预测将有助于选择个体化治疗策略,增加患者临床获益。
目前,利用临床影像预后因子的风险分层,从而确定直肠癌患者是否能从nCRT中获益。
①术前直肠系膜筋膜(MRF)侵犯
②术前壁外静脉侵犯(EMVI)
③ nCRT后EMVI阳性
④恶性肿瘤浸润超过固有基层外缘深度T3期 (如图所示)
⑤nCRT后的病理T分期(ypT)、N分期(ypN)
主要包括以下几种:
但问题在于这些临床影像预后因素的分层结果有显著的异质性。因此需要更有效的预后标志物改善LARC患者的风险分层,个性化治疗和预后。本文就是针对这个问题开展研究,想说明Radscore风险分层比临床的更好
我们可以看到,他提的临床问题是个好问题,影像组学研究也是临床研究,所以一个好的临床问题是文章高低的关键之一,到这里,背景部分结束。其实这里面大多都不用太了解,自己研究的背景和自己的领域相关,只要了解作者想干什么就行了。
下面就比较重要了,大家打起精神来:
影像组学模型构建
(a)高质量标准化MRI影像数据获取+感兴趣体积VOI分割
(b)影像组学特征提取,包括肿瘤直方图强度、形状特征、纹理特征、小波变换
(c)通过组内系数和 LASSO-Cox 回归模型选择特征选择与构建
(d)KM 曲线进行生存分析和ROC曲线的预测模型性能评估
随后绘制了列线图,对结合了临床特征和影像组学特征的预测模型结果进行可视化。
我们可以看出本文的影像组学过程十分简单,你上你也行,下面为大家说说细节:
第一步:纳入排除标准制定
纳入标准:
① 局部晚期直肠腺癌患者(>=T3 ,初始 MRI 显示淋巴结阳性或阴性)
② 肿瘤起源于肛缘 15 厘米以内的患者
③ 在TME 前接受 nCRT 治疗的患者
④ 在 nCRT 之前进行了盆腔 MRI 和胸部、腹部和盆腔CT可以进行肿瘤分期的患者
⑤ 接受文中Protocol规定的具体nCRT治理方案且在6-8周内进行TME的患者
⑥ 符合随访要求的患者
排除标准
① 有恶性肿瘤病史的患者
② 既往接受过盆腔放疗的患者
③ 有 MRI 禁忌症以及图像质量不足以进行分析的患者
最终,共纳入108名患者,按1:1随机分为训练集和验证集(中位DFS分别为34.5个月和22.5个月,P=0.847)
当然这是我整理好的,其实文章方法部分写的不太整洁
我们文章可以写的清晰明了些
不过本文的纳排标准的优点就是比较完整
其实纳排标准的构建也是有套路的
按PICOS原则:
为大家按PICOS原则为大家整理了一下
P:
年龄:无限定;
性别:无限定;
疾病状态:首诊;
疾病分期:局部晚期;
治疗方案:nCRT+TME;
诊断标准:无;
新增条目:肿瘤部位(这个算疾病相关参数),限定了距离肛缘15cm以内;
I/C:术前的影像组学模型
影像设备:MRI ;
扫描序列:LAVA动态增强扫面序列,T2自旋回波序列(Spin Echo, SE);
靶区:肿瘤区;
影像检查时间点:TME前,nCRT后;
层厚:3mm;
具体扫描参数:重复时间[TR]/ 回波时间 [TE] :5160/151 msec,翻转角:90°,回波链长度ETL,:19,编码矩阵512*512等;
O:
DFS(规定了最短的随访时间>3年,避免因为一些随访时间短而未观察到结局事件的发生导致的假阴性;)
S就是回顾队列,不说了;
这样是不是更清晰了呢;
第一次看有点懵正常,君君姐的专栏和平鑫而论专栏会分享这样的文章,倒是后大家量的积累就会有质的飞跃;
下面进行第二步
高质量标准化MRI影像数据获取及分析
在配备相控阵体线圈的 3T 扫描仪(SignaHDx,General Electric,Milwaukee,WI)上进行MRI检测。执行常规临床成像方案,最终,获得T2加权影像序列。
这里面文章并没有对获取的影像进行预处理操作,是个问题,没去批次效应,就感觉不是很规范了;日后会提提去批次效应的套路和细节
第三步
感兴趣体积VOI分割
利用ITK-SNAP 软件(www.itk-snap.org)对多增强 MRI 的第五阶段(造影剂注射后 60 秒)的 3D pre-nCRT MR 图像进行手动分割。
分割均由具有 15 年直肠 MR 图像解读经验的胃肠道医师执行,并由具有 20 年经验的高级医师进行验证。
第四步
图像预处理
作者在VOI分割之后,进行了图像处理操作,为了减少数据的可变性,并且易于计算定量放射组学特征。作者使用两步过程将原始图像转换为标准化输入:① 使用双三次重采样来标准化图像尺度;② 利用直方图匹配最小化患者MR图像之间强度分布的差异。使得转换后的 MR 图像具有相似的强度分布。
第五步
特征提取
本文使用MatLab v. R2015b 进行影像组学特征提取,最终得到485个特征。包括一阶统计特征(能量,熵,平均数,标准差,最大值等)、基于形状和基于尺寸的特征(最大3D直径,体积,表面积等)、纹理特征(灰度共生矩阵GLCM和灰度游程矩阵GLRLM)、基于小波变换特征。
这里提一下,一般来说影像组学特征包含以下四个类别:
① 形态学特征,描述VOI/ROI大小的特征。包括体积、表面积、肿瘤直径等(包括2D和3D)。
② 一阶灰度特征,VOI/ROI内不同灰度和概率分部的相关统计特征。包括最大值、最小值、平均数、标准差、方差、偏度和峰度、熵和能量等。
③ 二阶和高阶纹理特征,用于描绘VOI/ROI种灰度值的空间分部关系。包括绝对梯度(Absolute Gradient)、灰度共生矩阵 (GLCM)、灰度游程矩阵 (GLRLM)、灰度大小区域矩阵 (GLSZM)、邻域灰度差矩阵 (NGTDM)、灰度依赖矩阵 (GLDM)等
④ 基于滤波器和变换的其他特征。如傅立叶变换、拉普拉斯变换(LoG)(图像预处理)、Gabor变换、小波变换(深层挖掘)等。
特征提取是影像组学的核心步骤,如果能提取到各个类别的特征,会增加影像组学特征的质量,便于得到差异显著的影像组学特征。
大家做下笔记
第六步
特征一致性评价
随后进行对提取特征一致性评价:具有15 年直肠 MR 图像解读经验的胃肠道医师分割随机选择的25名患者的进行2次分割。VOI中 485 个放射组学特征中有 45% 被纳入分析(组内相关系数ICC > 0.8),并用于后续研究。所有影像组学特征均通过 z-score 转换进行归一化。
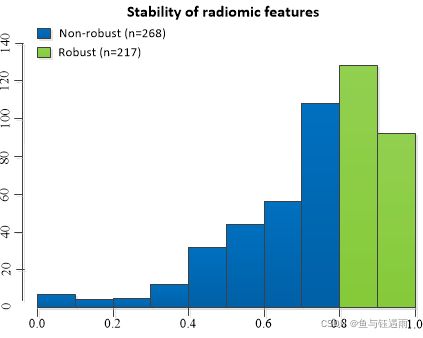
一般而言,由于分割工作由两名或多名医师完成,大多数影像组学文章的特征一致性评价部分是不同医师同一时间进行勾画评价Inter-observer-ICC(观察者间间一致性)进行比较和同一个医生不同时间点多次勾画的Intra-observer-ICC(观察者内一致性)。本文分割工作仅由一名医师完成,所以令其对相同的图像进行两次分割,进行Intra-observer-ICC的比较。
第七步 特征选择
在特征提取和数据预处理后,研究者得到了217个“鲁棒性”较好的影像组学特征。但是将所有模型直接纳入Cox模型显然是不合理的,因为过多的特征纳入既会产生过拟合问题,并使模型可解释性变弱,也会产生多重共线性问题使模型失真。
所以这里利用了机器学习中的LASSO回归算法(也是影像组学特征选择步骤有监督降维最经典的算法),使用L1范数进行收缩惩罚,将一些对结局贡献不大的特征排除在模型外。
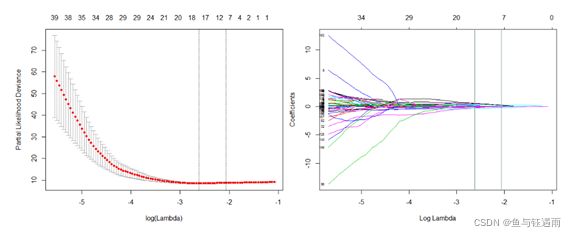
从图中我们可以看出,随着正则化系数λ的增大,各个特征的系数逐渐趋向于0,当然,也有部分特征迅速到0。不同的λ区域对应了不同的特征数目,通过计算最合适的λ值从而确定选择的特征。最终,通过10倍交叉验证,筛选了三个强度特征和五个纹理特征。所选特征的Cox模型的非零系数,组合成影像组学标签。然后使用所选特征的加权线性组合确定每位患者的Radscore。至此,LASSO-Cox模型建立完毕。
方法构建步骤到此结束,我们来看看结果:
患者基线:基线看table 1
根据以往的文献,本研究将性别、年龄、肿瘤分期(IIA,IIIB,IIIC)、原发肿瘤的大小和范围T、局部淋巴结受累情况N、新辅助放化疗前癌胚抗原水平(<4.75,>=4.75)、新辅助放化疗后癌胚抗原水平(<2.11,>=2.11)、壁外血管侵犯(EMVI)(-,+)、直肠系膜筋膜侵犯MRF(-,+)、病理T分期N分期(ypTN)(ypT0N0,non ypT0N0)、肿瘤局部复发、远端转移、随访时间等因素进行基线的描述,评估变量在两个数据集之间的均衡性。
然后就是对影像组学signature的性能评估,利用构建的LASSO-Cox模型预测每位患者的影像组学风险组,将训练集影像组学评分中位数作为截断值(cutoff=-14)应用于训练集和验证集中进行分层。低于-14分的患者被分为低风险组,高于-14分的患者被分为高风险组。
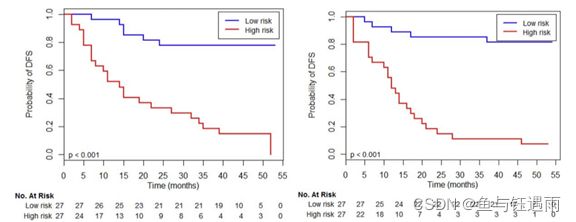
结果显示
训练集中 LARC 患者的影像组学特征与 DFS 之间存在显着相关性(HR = 6.83, 95% CI 3.65–12.79, P < 0.001),并在验证集中得到证实(HR = 2.92, 95% CI 1.91–4.47,P < 0.001)。风险组生存率通过LoG-RANK检验进行比较。
作者随后对训练集和测试集所有的病例进行单因素分析,确定了具有潜在预测预后价值的临床影像学因子。
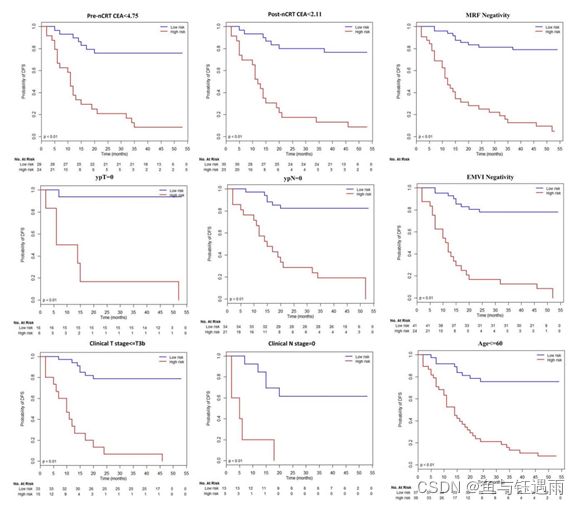
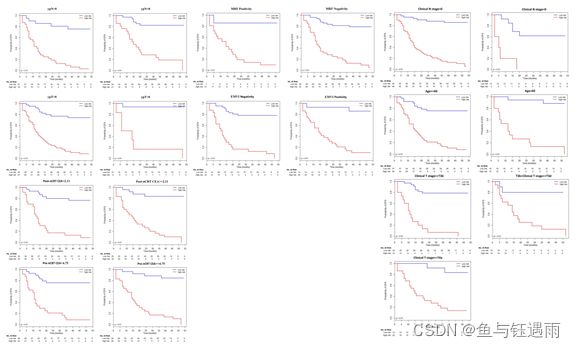
结果显示
单因素分析结果得出的直肠癌复发和转移的临床影像危险因素包括:ypT (HR = 1.37, 95% CI 1.07–1.74, P = 0.009), ypN (HR = 1.27, 95% CI 1.10–1.46, P < 0.001), EMVI (HR = 1.33, 95% CI 1.06 –1.68, P = 0.013) ,MRF (HR = 1.53, 95% CI 1.24–1.90, P < 0.001) 。基于单因素分析结果,使用确定的风险因素和影像组学标签进行基于 Cox 回归模型的多因素分析。结果表明9个特征中,MRF (P = 0.032)、ypN (P = 0.027) 和影像组学评分 (P < 0.001) 是独立预测因子。但这里有个问题,作者均没有展示单因素分析和多因素分析的Table。而且连多因素分析的HR都没有里面就有猫腻了。我们主要学方法,不管他,随后绘制列线图,根据上一步得到的DSF相关的独立预测因子MRF、ypN和影像组学评分构建列线图。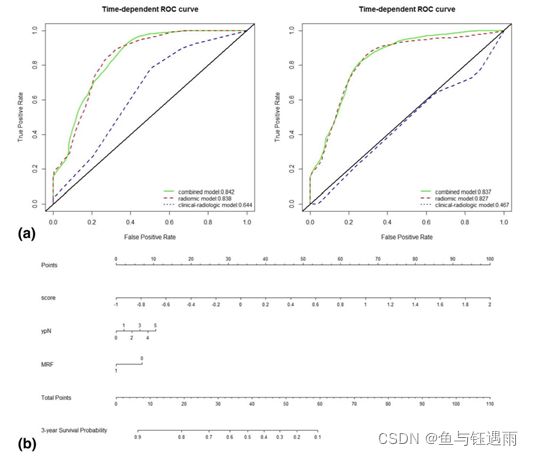
Figure 4
结果显示
(a) 训练集和测试集三年DFS的时间依赖型ROC曲线,相比于影像组学模型和临床影响特征模型,联合模型有更好的预测效果。
(b) 通过训练集的MRF、ypNRadscore和开发的列线图
本文仅对列线图的区分度进行了评价(其实也不算评价了),并没有对列线图的校准度、临床获益等进行评价。可见作者对列线图的评价体系认知是不完整的,这是几行代码就能出来的加分项目,大家写文章时千万不要忘了哦
作者对列线图结果少了两个评价,一般来说,区分度进、校准度、临床获益才是完整的;
下面总结一下,这是一篇17年的文章,挑选本文的目的是让大家能够了解到影像组学模型构建的最原始方式:单模态、单靶标、单模型、单结局变量,样本量也不大,100例出头。整个构建的过程也基本是固定的。
① 构建纳入排除标准
② 高质量影像数据获取
③ 感兴趣区ROI/VOI的分割
④ 影像组学特征提取
⑤ 特征的一致性评价
⑥ 特征选择
⑦ 模型构建
⑧ 模型评价
但涉及到每一个具体的步骤,都有很多细节需要考虑,只有多了解不同类型的文章,才能做到融会贯通,领悟其中的精髓。
分享结束,大家有什么问题吗。
数据库
那大家想知道,如果没有原始影像Dicom数据该去哪里瞧一瞧吗?比如公共数据库?那在哪里,有哪些数据?发表过啥样的文章?

TCIA数据库(The Cancer Imaging Archive)
今天给大家介绍的就是一个白嫖通道——TCIA数据库,一个癌症研究的医学图像的大规模公用数据库。
一,数据库简介:
癌症图像档案 (The Cancer Imaging Archive,TCIA)是一项可以挖掘和提供可供公众下载大量癌症医学图像的数据库(TCIA网址:https://www.cancerimagingarchive.net/)。
弟弟已经把单中心回顾性套路讲的非常棒了,医院有数据,就可以模仿试试,那我们再来补充,没有数据如何入局
首先,分两类人群,第一,有时间,有精力,对科研充满热情,那就是自己去学习,也关注我们的讲习营,无论r还是python.不在话下
那我们,先来学习,大家自取所需,首先,我们要知道TCIA数据库中目前收录的数据涉及8种疾病,包含卵巢癌,肾癌,低级别胶质瘤,高级
别胶质瘤,肝癌,乳腺癌,膀胱癌,头颈鳞癌、肺癌。
如果没有你的疾病领域,也是巧妇难为无米之炊;但是有的话,尽快采矿;
也可以看下国自然的影像组学中标课题的类型,对我们自己研究设计也有参考
铺垫了一下,那我们再来回到数据库
咋用呢
TCIA网址:https://www.cancerimagingarchive.net/
先打开网址
在如上界面可以点击①上传数据,也可以点击② 探索需要下载的数据集。
当点击access the data 就可以跳转如下界面:在TCIA中,数据合集叫“collections”;典型的病人与常见病(如肺癌)相关的影像、影像形态或类型(MRI、CT和digital histopathology等)或研究重点。DICOM是TCIA用于放射影像的主要文件格式。也提供了可用的与图像相关的支持数据,如患者结果、治疗方案及效果、基因组学和专家分析数据等。
肺癌是影像组学龙头,其次是乳腺癌,生殖系肿瘤
New Collection proposals由TCIA Advisory Group审阅。如果获得批准,数据收集中心(DCC)将为图像提供商提供亲身实践的(hands-on)支持,以再审核和管理他们的数据。数据经过处理后,可以通过四种不同的方式供用户访问:
TCIA还鼓励创建数据分析中心(DACs), DACs通过连接到TCIA REST API或镜像Collections,为可视化或分析TCIA数据提供了额外的功能。
1,从首页访问Collection摘要页面,其中提供了每个数据集的详细说明以及直接下载链接,快速获取给定Collection的所有图像和支持数据。
为了提高TCIA的collections的价值,TCIA也鼓励研究人员发表他们的分析结果。潜在的分析可能包括肿瘤分割、放射组学特征、导出/再处理图像和影像学家评估。用户可以在分析结果目录中查看其他TCIA用户发布的分析。
2,影像学和组织病理学数据门户提供更高级的搜索、浏览和过滤功能,以选择图像子集或从满足搜索标准的多个集合下载图像。编程接口(REST API)允许软件开发人员在他们的脚本和应用程序中构建对TCIA数据的访问。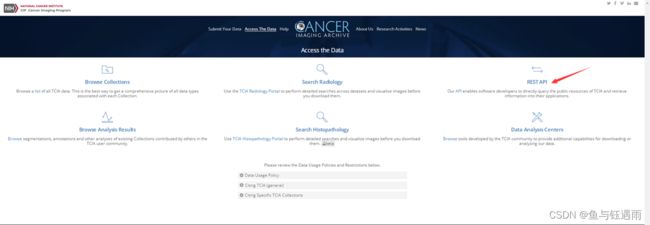
二,数据库使用
接下来咱们介绍一下数据库主要模块的使用,其他的模块大家需要的可以自行进入查看~
1) 获取数据Access The Data:
2)数据入口控制面板Data Portals Dashboard

3)数据浏览Browse Data Collections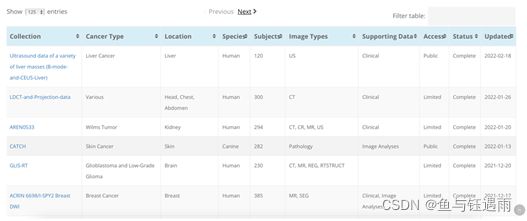
4)浏览分析结果Browse Analysis Results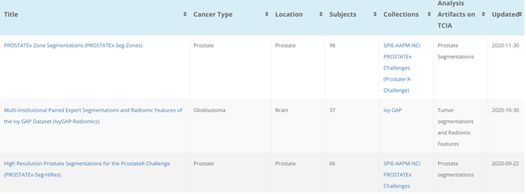
为了提高TCIA collections的价值,TCIA 鼓励研究界发表他们对现有TCIA图像收藏的分析。此类数据的包括影像学家或病理学家注释、图像分类、分割、放射组学特征或导出/再处理图像。与提交新的图像集合类似,这些数据由TCIA顾问组进行相关性审查,并使用他们的正常流程进行策划,以确保数据被再审核。
然而,TCIA不能保证分析本身的质量(例如,在给定扫描上分割的准确性)。所以在决定这些分析是否有用之前,我们还要仔细审查数据和 相关的出版物。
5)检索影像学Search Radiology Portal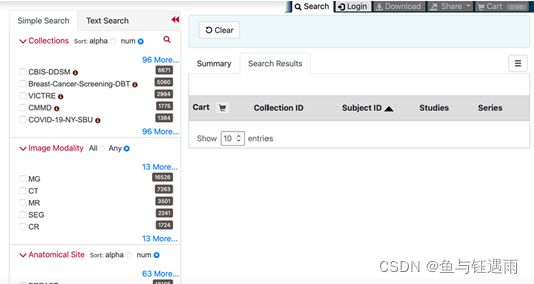
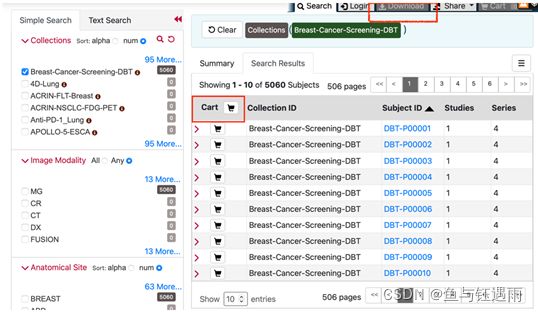
左侧边框栏用于查找数据,右侧用于查看,选择数据集添加购物车后下载,是不是跟TCGA挺像?
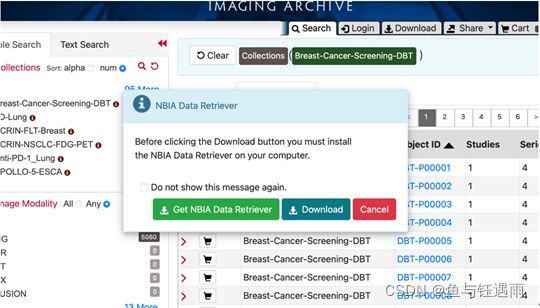
想要下载和打开数据必须安装NBIA Data Retriever,会自动弹出提示指引下载
下载好的文件最终文件是.dcm格式。dcm文件是一种数位成像,广泛运用于医学领域,但并不是仅仅局限于医学。本身dcm一种特殊的图像文件,它可以用来存储各种图像信息,这种特殊的图像文件大家需要自行下载专用的软件打开。
但是在这里注意哦,有些浏览器,或者,大部分,都会弹出一个插件,让你下载,教程也非常详细,按照网页给的步骤,一般不成问题哦;还要注意,Dicom文件,是非常庞大的;例如,截图一个最近看的肺癌的文章,原始Dicom文件就有几十个G,需要网络好和内存够哦;
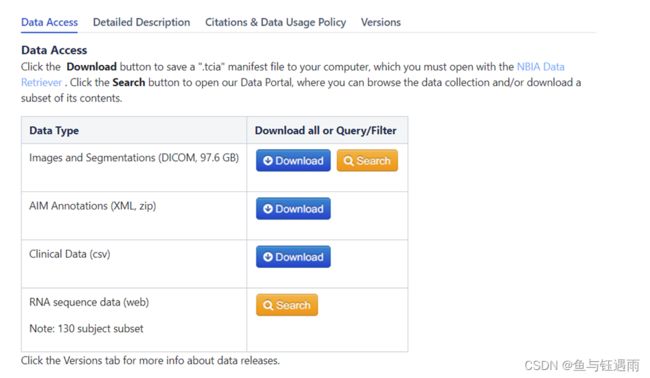
这个就有97G多,TCIA不仅有影像还有病理资料哦
TCIA不仅有影像还有病理资料哦
6) 搜索组织病理学Search Histopathology Portal
最近就看到了几篇病理组学,影像组学、基因组学等联合的文章
CPTAC:从临床蛋白组学肿瘤分析联盟(Clinical Proteomic Tumor Analysis Consortium,CPTAC)的collections中搜索和可视化数据,以分析可能与相应的蛋白组学、基因组学和临床数据相关的癌症表型
可以白嫖多组学了
关于发表了影像组或病理组文章,如何提交数据
这里简单提及
TCIA数据集New TCIA Dataset

这里介绍了向TCIA提交新的影像数据集的过程。如果是利用了现有的TCIA数据,希望发布你的分析,也可以在这里找到操作说明。
- 现有TCIA数据集的分析Analysis of Existing TCIA Datasets
除了发布新的TCIA数据集,TCIA鼓励发布来自现有TCIA数据集的分析。例子(见之前上传的分析数据集)包括图像标签、注释、器官/肿瘤片段和放射体/病理特征。
- Submission and De-identification Overview:
这部分是提供TCIA关于数据收集、识别和管理的协议细节。
四,相关研究Research Activities
-
基于TCIA的研究Publications Based on TCIA
-
蛋白质基因组学成像Imaging Proteogenomics
这里列举了引用TCIA的数据进行研究的工作,并进行了分类。
TCIA支持一个寻求连接癌症表型和基因型的研究社区。为了实现这一目标,TCIA连接临床图像与患者基因组数据和蛋白质组数据的数据集。要访问相应基因组和/或蛋白质组数据的数据集,请使用主页上的“Supporting Data”栏来筛选“Genomics”和/或“Proteomics”数据集。这些数据中有一部分是由研究团体提供的,但其中大部分是作为NIH大规模数据收集活动的一部分收集的。
临床试验的影像学数据提供了将影像学特征与临床试验分析、相关临床数据和患者结果联系起来的机会。从2019年开始,NCI癌症成像计划信息学实验室正在支持一个额外的TCIA图像数据收集中心,该中心特别关注临床试验数据。
2019年才开始
大把空间哦
许多TCIA临床试验数据集将来自NCI国家临床试验网络(NCTN),该网络是组织和临床医生的集合,协调和支持美国和加拿大超过3000个地点的癌症临床试验。NCTN为NCI-funded的治疗、筛查和诊断试验提供基础设施,以改善癌症患者的生活。NCTN明确要求在https://nctn-data-archive.nci.nih.gov/view-trials中共享患者级临床数据,而TCIA作为该档案的图像存储库。
其中临床试验成像Imaging Clinical Trials板块也可以自己探索
任何科研都是充满挑战哦,大浪淘沙
这里的挑战Challenge Competitions:
TCIA收集的数据已经并将继续用于图像分析挑战或比赛,如图像分割或肿瘤分类。但是由于影像组学流程太多
回顾下鑫弟讲的非常细致的影像组学流程
如果同样的患者人群,同一批人做,都有可能做出来有些不一样的结果,特别是细节部分,大方向可能是一致滴
但是,公共数据
略过人工勾画,或者仪器测两遍,算ICC等,直接用它的数据集和代码
那就更容易复现了
总结:TCIA数据库提供了大量的影像学和组织病理学图像数据集,可以搜索、浏览和过滤功能,还能从满足搜索标准的多个集合下载图像。同时也允许软件开发人员在他们的脚本和应用程序中构建对TCIA数据的访问。TCIA还创建数据分析中心(DACs),为可视化或分析TCIA数据提供了额外的功能。TCIA还鼓励研究人员在TCIA发表他们的分析结果。潜在的分析可能包括肿瘤分割、放射组学特征、导出/再处理图像和影像学家评估。用户可以在分析结果目录中查看其他TCIA用户发布的分析。
一个数据库,一晚上肯定探索的很浅哦
PS:酸菜校长曾经讲过,如果想吃透一个数据库,就把能点的链接都点点,能翻译的英文都掌握了,一般就学会了。
最后补充一句,非肿瘤,能不能做影像组学
TCIA官网有酱一句话:事实证明,影像组学对COVID-19感染患者使用成像技术对患者分类、对预后不良的风险评估(特别是在危险人群中)和随访都很有价值。
问答环节
1.老师好 现在有什么方法做影像组学于基因组学的联合分析么
首先,对数据进行正态检验,方差分析,如果基因和影像组特征值,都符合正态分布;那就用皮尔森相关,我把文献截图给你;

老师,想做超声方向,请问怎么查找超声组学的相关文章,用什么关键词呢?
你如果不知道你的主题词,你就用仙桃,输入想输入的中文看到想要的词,再贴去各种文献数据库
老师好 基因数据和影像学数据一定是得同样本么? 发高分,60例患者够么?
发高分,最好是同样本,而且,还要把位置对应更好,60例,除非是单细胞和PET。不然,有点难哦。
影像组学联合单细胞,几十例可以发10分。