核支持向量机(SVM)(笔记)
最近在自学图灵教材《Python机器学习基础教程》,在csdn以博客的形式做些笔记。
之前在逻辑回归与线性支持向量机中学习了可以用于分类的线性支持向量机(LinearSVC),而核支持向量机(SVM)可以用于更复杂的模型,这些模型无法被输入空间的超平面定义。对于支持向量机而言,它可以用于分类(SVC中实现)和回归(SVR中实现),本文只涉及分类。
线性模型与非线性特征
首先我们来看一下下面的数据集
import mglearn
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.datasets import make_blobs
X, y = make_blobs(centers=4, random_state=8)
y = y % 2
mglearn.discrete_scatter(X[:, 0], X[:, 1], y)
plt.xlabel("Feature 0")
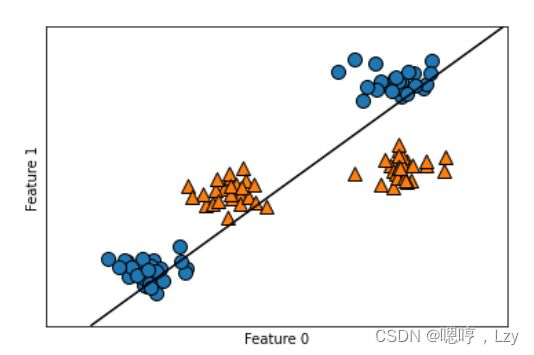
plt.ylabel("Feature 1")可以发现,这个二分类数据集并不是线性可分的,也就是说无法用一条直线来划分数据集,但是线性分类模型却只能用一条直线来划分。(下面用 LinearSVC举例)
from sklearn.svm import LinearSVC
linear_svm = LinearSVC().fit(X, y)
mglearn.plots.plot_2d_separator(linear_svm, X)
mglearn.discrete_scatter(X[:, 0], X[:, 1], y)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")可以发现线性支持向量机的效果并不好。
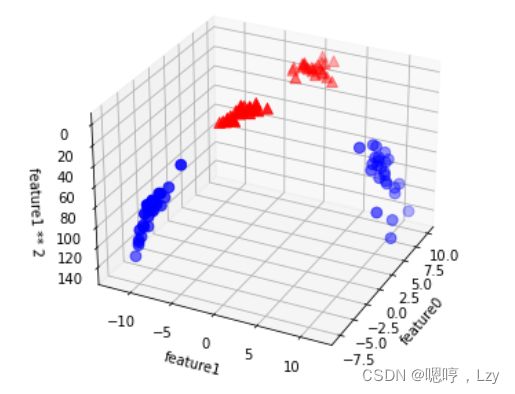
现在我们对输入特征进行扩展,比如说添加第二个特征的平方(feature1 ** 2)作为一个新特征。现在我们将每个数据点表示为三维点 (feature0, feature1, feature1 ** 2),而 不是二维点 (feature0, feature1)。
# 添加第二个特征的平方,作为一个新特征
X_new = np.hstack([X, X[:, 1:] ** 2])
from mpl_toolkits.mplot3d import Axes3D, axes3d
figure = plt.figure()
# 3D可视化
ax = Axes3D(figure, elev=-152, azim=-26)
# 首先画出所有y == 0的点,然后画出所有y == 1的点
mask = y == 0
ax.scatter(X_new[mask, 0], X_new[mask, 1], X_new[mask, 2], c='b',
cmap=mglearn.cm2, s=60)
ax.scatter(X_new[~mask, 0], X_new[~mask, 1], X_new[~mask, 2], c='r', marker='^',
cmap=mglearn.cm2, s=60)
ax.set_xlabel("feature0")
ax.set_ylabel("feature1")
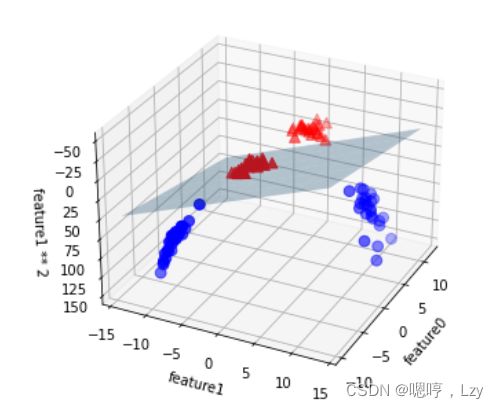
ax.set_zlabel("feature1 ** 2")在数据的新表示中,现在可以用线性模型(三维空间中的平面)将这两个类别分开。我们可以用线性模型拟合扩展后的数据来验证这一点。
linear_svm_3d = LinearSVC().fit(X_new, y)
coef, intercept = linear_svm_3d.coef_.ravel(), linear_svm_3d.intercept_
# 显示线性决策边界
figure = plt.figure()
ax = Axes3D(figure, elev=-152, azim=-26)
xx = np.linspace(X_new[:, 0].min() - 2, X_new[:, 0].max() + 2, 50)
yy = np.linspace(X_new[:, 1].min() - 2, X_new[:, 1].max() + 2, 50)
XX, YY = np.meshgrid(xx, yy)
ZZ = (coef[0] * XX + coef[1] * YY + intercept) / -coef[2]
ax.plot_surface(XX, YY, ZZ, rstride=8, cstride=8, alpha=0.3)
ax.scatter(X_new[mask, 0], X_new[mask, 1], X_new[mask, 2], c='b',
cmap=mglearn.cm2, s=60)
ax.scatter(X_new[~mask, 0], X_new[~mask, 1], X_new[~mask, 2], c='r', marker='^',
cmap=mglearn.cm2, s=60)
ax.set_xlabel("feature0")
ax.set_ylabel("feature1")
ax.set_zlabel("feature1 ** 2")我们发现线性支持向量机在新数据集上的表现为一个平面,完美划分了两个数据集,但是如果将线性SVM模型看作是原始数据集上的函数,那么它实际上就不是线性的了。它不再是一条直线,而是一个椭圆。
核技巧
从上面的例子可以看出,向数据集中添加非线性特征,可以让线性模型变得更有效。但问题是,我们应该添加哪些特征呢?这个问题很难,我们大多数不知道添加什么样的特征。但是有一种巧妙的数学方法,让我们可以在更高维空间中学习分类器,而不用实际计算可能非常大的新的数据表示。这种技巧叫作核技巧(kernel trick),它的原理是直接计算扩展特征表示中数据点之间的距离(更准确地说是内积),而不用实际对扩展进行计算。
对于支持向量机,将数据映射到更高维空间中有两种常用的方法:一种是多项式核,在一 定阶数内计算原始特征所有可能的多项式(比如 feature1 ** 2 * feature2 ** 5);另一 种是径向基函数(radial basis function,RBF)核,也叫高斯核。高斯核有点难以解释,因为它对应无限维的特征空间。一种对高斯核的解释是它考虑所有阶数的所有可能的多项式,但阶数越高,特征的重要性越小。不过在实践中,核 SVM 背后的数学细节并不是很重要,可以简单地总结出使用 RBF 核 SVM 进行预测的方法。
SVM
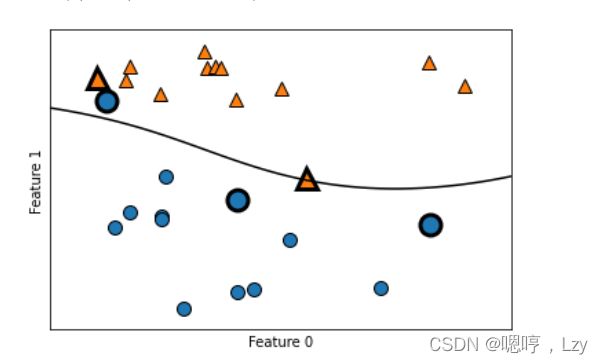
在训练过程中,SVM 学习每个训练数据点对于表示两个类别之间的决策边界的重要性。通常只有一部分训练数据点对于定义决策边界来说很重要:位于类别之间边界上的那些点。 这些点叫作支持向量(support vector),支持向量机正是由此得名。
想要对新样本点进行预测,需要测量它与每个支持向量之间的距离。分类决策是基于它与支持向量之间的距离以及在训练过程中学到的支持向量重要性(保存在 SVC 的 dual_coef_ 属性中)来做出的。
数据点之间的距离由高斯核给出: Krbf (x1, x2) = exp (-γ‖x1 - x2‖2 ) 这里 x1 和 x2 是数据点,‖x1 - x2‖ 表示欧氏距离,γ(gamma)是控制高斯核宽度的参数。
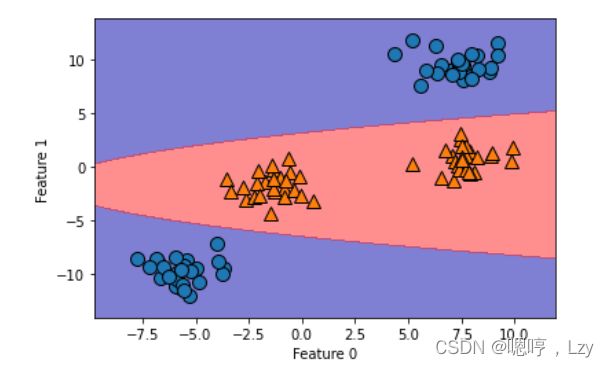
如下图中加粗的点就是支持向量
上图中,SVM给出了一个平滑的非线性边界。而对于SVM的应用主要用到两个参数:C和gama
SVM调参
gamma 参数是上面给出的公式中的参数,用于控制高斯核的宽度。它决定了点与点之间 “靠近”是指多大的距离。C 参数是正则化参数,与线性模型中用到的类似。它限制每个点的重要性(或者更确切地说,每个点的 dual_coef_)。
接下来我们来比对一下不同大小的参数的效果
fig, axes = plt.subplots(3, 3, figsize=(15, 10))
for ax, C in zip(axes, [-1, 0, 3]):
for a, gamma in zip(ax, range(-1, 2)):
mglearn.plots.plot_svm(log_C=C, log_gamma=gamma, ax=a)
axes[0, 0].legend(["class 0", "class 1", "sv class 0", "sv class 1"],
ncol=4, loc=(.9, 1.2))从左到右,我们将参数 gamma 的值从 0.1 增加到 10。gamma 较小,说明高斯核的半径较大, 许多点都被看作比较靠近。这一点可以在图中看出:左侧的图决策边界非常平滑,越向右 的图决策边界更关注单个点。小的 gamma 值表示决策边界变化很慢,生成的是复杂度较低的模型,而大的 gamma 值则会生成更为复杂的模型。 从上到下,我们将参数 C 的值从 0.1 增加到 1000。与线性模型相同,C 值很小,说明模型非常受限,每个数据点的影响范围都有限。你可以看到,左上角的图中,决策边界看起来 几乎是线性的,误分类的点对边界几乎没有任何影响。再看左下角的图,增大 C 之后这些 点对模型的影响变大,使得决策边界发生弯曲来将这些点正确分类。
我们将 RBF 核 SVM 应用到乳腺癌数据集上。默认情况下,C=1,gamma=1/n_features:
from sklearn.datasets import load_breast_cancer
cancer=load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(
cancer.data, cancer.target, random_state=0)
svc = SVC()
svc.fit(X_train, y_train)
print("Accuracy on training set: {:.2f}".format(svc.score(X_train, y_train)))
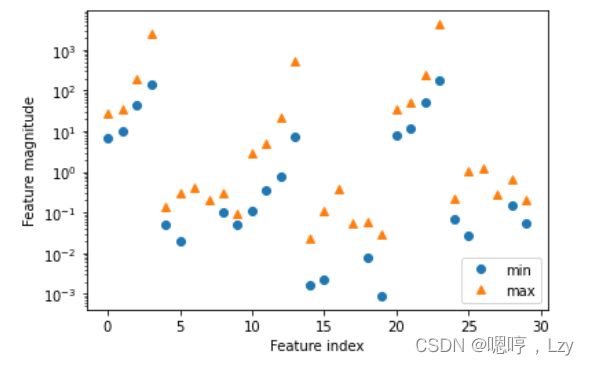
print("Accuracy on test set: {:.2f}".format(svc.score(X_test, y_test)))这个模型在训练集上的分数十分完美,但在测试集上的精度只有 63%,存在相当严重的过 拟合。虽然 SVM 的表现通常都很好,但它对参数的设定和数据的缩放非常敏感。特别地, 它要求所有特征有相似的变化范围。我们来看一下每个特征的最小值和最大值
plt.plot(X_train.min(axis=0), 'o', label="min")
plt.plot(X_train.max(axis=0), '^', label="max")
plt.legend(loc=4)
plt.xlabel("Feature index")
plt.ylabel("Feature magnitude")
plt.yscale("log")
从这张图中,我们可以确定乳腺癌数据集的特征具有完全不同的数量级。这对其他模型来 说(比如线性模型)可能是小问题,但对核 SVM 却有极大影响。我们来研究处理这个问 题的几种方法。
为SVM预处理数据
解决这个问题的一种方法就是对每个特征进行缩放,使其大致都位于同一范围。核 SVM 常用的缩放方法就是将所有特征缩放到 0 和 1 之间。
# 计算训练集中每个特征的最小值
min_on_training = X_train.min(axis=0)
# 计算训练集中每个特征的范围(最大值-最小值)
range_on_training = (X_train - min_on_training).max(axis=0)
# 减去最小值,然后除以范围
# 这样每个特征都是min=0和max=1
X_train_scaled = (X_train - min_on_training) / range_on_training
print("Minimum for each feature\n{}".format(X_train_scaled.min(axis=0)))
print("Maximum for each feature\n {}".format(X_train_scaled.max(axis=0)))然后我们来看看效果
# 利用训练集的最小值和范围对测试集做相同的变换
X_test_scaled = (X_test - min_on_training) / range_on_training
svc = SVC()
svc.fit(X_train_scaled, y_train)
print("Accuracy on training set: {:.3f}".format(svc.score(X_train_scaled, y_train)))
print("Accuracy on test set: {:.3f}".format(svc.score(X_test_scaled, y_test)))
数据缩放的作用很大!实际上模型现在处于欠拟合的状态,因为训练集和测试集的性能非 常接近,但还没有接近 100% 的精度。从这里开始,我们可以尝试增大 C 或 gamma 来拟合 更为复杂的模型。
svc=SVC(C=1000)
svc.fit(X_train_scaled,y_train)
print("Accuracy on training set:{:.3f}".format(svc.score(X_train_scaled,y_train)))
print("Accuracy on test set:{:.3f} ".format(svc.score(X_test_scaled,y_test)))总结
核支持向量机是非常强大的模型,在各种数据集上的表现都很好。SVM 允许决策边界很复杂,即使数据只有几个特征。它在低维数据和高维数据(即很少特征和很多特征)上的表现都很好,但对样本个数的缩放表现不好。在有多达 10 000 个样本的数据上运行 SVM 可能表现良好,但如果数据量达到 100 000 甚至更大,在运行时间和内存使用方面可能会 面临挑战。 SVM 的另一个缺点是,预处理数据和调参都需要非常小心。这也是为什么如今很多应用 中用的都是基于树的模型,比如随机森林或梯度提升(需要很少的预处理,甚至不需要预 处理)。此外,SVM 模型很难检查,可能很难理解为什么会这么预测,而且也难以将模型 向非专家进行解释。