SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
点击关注,桓峰基因
Topic 6. 克隆进化之 Canopy
Topic 7. 克隆进化之 Cardelino
Topic 8. 克隆进化之 RobustClone
SCS【1】今天开启单细胞之旅,述说单细胞测序的前世今生
SCS【2】单细胞转录组 之 cellranger
SCS【3】单细胞转录组数据 GEO 下载及读取
SCS【4】单细胞转录组数据可视化分析 (Seurat 4.0)
SCS【5】单细胞转录组数据可视化分析 (scater)
SCS【6】单细胞转录组之细胞类型自动注释 (SingleR)
SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞
SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
这期继续介绍 Monocle 3 软件包用于筛选每个cluster的标记基因。
前言
单细胞转录组测序(scRNA-seq)实验使我们能够发现新的细胞类型,并帮助我们了解它们是如何在发育过程中产生的。Monocle 3包提供了一个分析单细胞基因表达实验的工具包。
Monocle 3可以执行三种主要类型的分析:
-
聚类、分类和计数细胞。单细胞RNA-Seq实验允许发现新的(可能是罕见的)细胞亚型。
-
构建单细胞轨迹。在发育、疾病和整个生命过程中,细胞从一种状态过渡到另一种状态。Monocle 3可以发现这些转变。
-
差异表达分析。对新细胞类型和状态的描述,首先要与其他更容易理解的细胞进行比较。Monocle 3包括一个复杂的,但易于使用的表达系统。
工作流程图如下:
也就是流程图中的黄色框框的部分,包括筛选每个 cluster 中的标记基因,并对此进行细胞的注释。
软件包安装
软件包除了需要我们安装Monocle 3之外,还需要安装 garnett 用于细胞注释的软件包,安装如下:
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(version = "3.14")
BiocManager::install(c('BiocGenerics', 'DelayedArray', 'DelayedMatrixStats',
'limma', 'lme4', 'S4Vectors', 'SingleCellExperiment',
'SummarizedExperiment', 'batchelor', 'Matrix.utils',
'HDF5Array', 'terra', 'ggrastr'))
install.packages("devtools")
devtools::install_github('cole-trapnell-lab/monocle3')
## Install the monocle3 branch of garnett
BiocManager::install(c("org.Mm.eg.db", "org.Hs.eg.db"))
devtools::install_github("cole-trapnell-lab/garnett", ref="monocle3")
# install gene database for worm
BiocManager::install("org.Ce.eg.db")
数据读取
数据读取我们可以参考上期 SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞 。这里数据读取也完整的给出来,我们对数据分析到第四步利用 UMAP对数据进行降维聚类,当然有需要也可以选择 t-SNE 方法,如下:
library(monocle3)
expression_matrix <- readRDS("cao_l2_expression.rds")
cell_metadata <- readRDS("cao_l2_colData.rds")
gene_annotation <- readRDS("cao_l2_rowData.rds")
# Make the CDS object
cds <- new_cell_data_set(expression_matrix, cell_metadata = cell_metadata, gene_metadata = gene_annotation)
cds
## Step 1: Normalize and pre-process the data
cds <- preprocess_cds(cds, num_dim = 100, method = c("PCA", "LSI"))
plot_pc_variance_explained(cds)
## Step 2: Remove batch effects with cell alignment cds <- align_cds(cds,
## alignment_group = 'batch')
## Step 3: Reduce the dimensions using 'UMAP', 'tSNE', 'PCA', 'LSI', 'Aligned'
cds <- reduce_dimension(cds, reduction_method = "UMAP")
## Step 4: Cluster the cells
cds <- cluster_cells(cds)
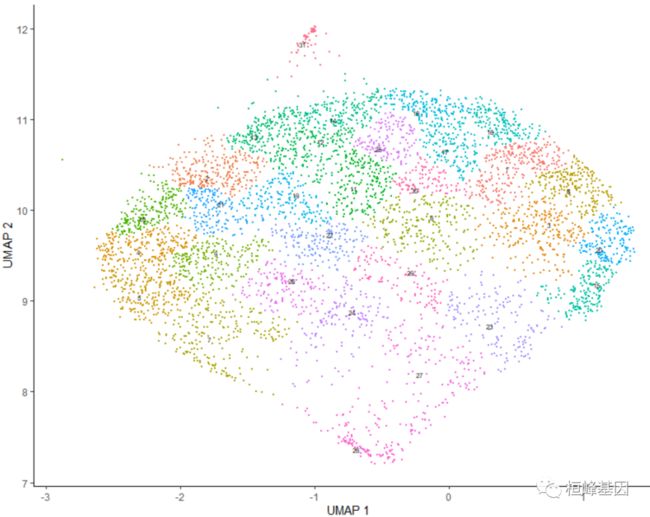
plot_cells(cds)
例子实操
一旦细胞聚集起来,我们就想知道是什么基因使它们彼此不同。要做到这一点,首先调用 top_markers()函数,数据框 marker_test_res 包含了许多关于每个分区中每个基因的具体表达方式的度量。我们可以根据 cluster 、partition 或 colData(cds) 中的任何变量对细胞进行分组。您可以根据一个或多个特异性指标对其进行排序,并选取每个聚类取最上面的基因。例如,pseudo_R2 就是这样一个度量。我们可以根据 pseudo_R2 对 markers 进行排序,如下所示:
筛选标记基因
library(dplyr)
marker_test_res <- top_markers(cds, group_cells_by = "partition", reference_cells = 1000,
cores = 8)
top_specific_markers <- marker_test_res %>%
filter(fraction_expressing >= 0.1) %>%
group_by(cell_group) %>%
top_n(1, pseudo_R2)
top_specific_marker_ids <- unique(top_specific_markers %>%
pull(gene_id))
我们可以用 plot_genes_by_group 函数绘制每组中表达每种 markers 的细胞的表达和比例:
plot_genes_by_group(cds, top_specific_marker_ids, group_cells_by = "partition", ordering_type = "maximal_on_diag",
max.size = 3)
查看多个 markers 通常会提供更多信息,你可以通过将top_n()的第一个参数改为 3 来实现:
####### top_n(3)##
top_specific_markers <- marker_test_res %>%
filter(fraction_expressing >= 0.1) %>%
group_by(cell_group) %>%
top_n(3, pseudo_R2)
top_specific_marker_ids <- unique(top_specific_markers %>%
pull(gene_id))
plot_genes_by_group(cds, top_specific_marker_ids, group_cells_by = "partition", ordering_type = "cluster_row_col",
max.size = 3)
细胞类型注释
识别数据集中每个细胞的类型对于许多下游分析是至关重要的。有几种方法可以做到这一点。一种常用的方法是首先将细胞聚类,然后根据其基因表达谱为每一簇分配一种细胞类型。
1. 根据类型注释细胞
我们已经看到了如何使用top_markers()。回顾与标记基因相关的文献,通常会给出表达该基因的簇的身份的强烈指示。在Cao & Packer >等人的研究中,作者查阅了文献和基因表达数据库中限制在每个聚类的标记,以便分配colData(cds)$cao_cell_type中包含的身份。
要基于cluster分配细胞类型,我们首先在colData(cds)中创建一个新列,并用 partitions(cds) 的值初始化它(也可以使用clusters(cds),这取决于你的数据集):
colData(cds)$assigned_cell_type <- as.character(partitions(cds))
colData(cds)$assigned_cell_type <- dplyr::recode(colData(cds)$assigned_cell_type,
`1` = "Body wall muscle", `2` = "Germline", `3` = "Motor neurons", `4` = "Seam cells",
`5` = "Sex myoblasts", `6` = "Socket cells", `7` = "Marginal_cell", `8` = "Coelomocyte",
`9` = "Am/PH sheath cells", `10` = "Ciliated neurons", `11` = "Intestinal/rectal muscle",
`12` = "Excretory gland", `13` = "Chemosensory neurons", `14` = "Interneurons",
`15` = "Unclassified eurons", `16` = "Ciliated neurons", `17` = "Pharyngeal gland cells",
`18` = "Unclassified neurons", `19` = "Chemosensory neurons", `20` = "Ciliated neurons",
`21` = "Ciliated neurons", `22` = "Inner labial neuron", `23` = "Ciliated neurons",
`24` = "Ciliated neurons", `25` = "Ciliated neurons", `26` = "Hypodermal cells",
`27` = "Mesodermal cells", `28` = "Motor neurons", `29` = "Pharyngeal gland cells",
`30` = "Ciliated neurons", `31` = "Excretory cells", `32` = "Amphid neuron",
`33` = "Pharyngeal muscle")
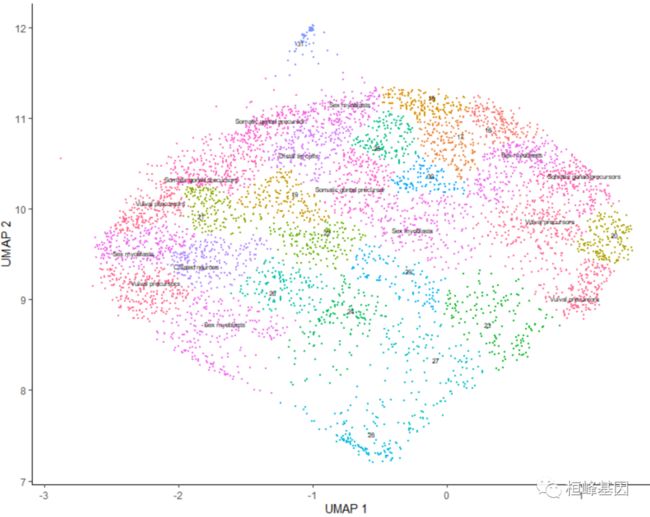
plot_cells(cds, group_cells_by = "partition", color_cells_by = "assigned_cell_type")
Partition 7 有一些子结构,仅从top_markers()的输出中看不出它对应的单元格类型是什么。所以我们可以用choose_cells()函数将它分离出来以便进一步分析:
cds_subset <- choose_cells(cds)
现在我们有了一个更小的cell_data_set对象,只包含我们想要筛选的细胞。我们可以使用 graph_test() 来识别在该分区的不同细胞子集中差异表达的基因:
pr_graph_test_res <- graph_test(cds_subset, neighbor_graph = "knn", cores = 8)
pr_deg_ids <- row.names(subset(pr_graph_test_res, morans_I > 0.01 & q_value < 0.05))
gene_module_df <- find_gene_modules(cds_subset[pr_deg_ids, ], resolution = 0.001)
plot_cells(cds_subset, genes = gene_module_df, show_trajectory_graph = FALSE, label_cell_groups = FALSE)
也可以探索每个模块中的基因,或对它们进行 GO 富集分析,以收集关于存在哪些细胞类型的解读。假设做完这些之后,我们对分区中的细胞类型有了很好的了解。让我们以更好的分辨率重新聚类的细胞,然后看看它们是如何与分区中聚类重叠的:
cds_subset <- cluster_cells(cds_subset, resolution = 0.01)
plot_cells(cds_subset, color_cells_by = "cluster")
基于模式的排列方式,我们将进行以下分配:
colData(cds_subset)$assigned_cell_type <- as.character(clusters(cds_subset)[colnames(cds_subset)])
colData(cds_subset)$assigned_cell_type <- dplyr::recode(colData(cds_subset)$assigned_cell_type,
`1` = "Sex myoblasts", `2` = "Somatic gonad precursors", `3` = "Vulval precursors",
`4` = "Sex myoblasts", `5` = "Vulval precursors", `6` = "Somatic gonad precursors",
`7` = "Sex myoblasts", `8` = "Sex myoblasts", `9` = "Ciliated neurons", `10` = "Vulval precursors",
`11` = "Somatic gonad precursor", `12` = "Distal tip cells", `13` = "Somatic gonad precursor",
`14` = "Sex myoblasts", `15` = "Vulval precursors")
plot_cells(cds_subset, group_cells_by = "cluster", color_cells_by = "assigned_cell_type")
现在,我们可以将 cds_subset 对象中的注释传输回完整的数据集。在这个阶段,我们还会过滤掉低质量的细胞:
colData(cds)[colnames(cds_subset), ]$assigned_cell_type <- colData(cds_subset)$assigned_cell_type
cds <- cds[, colData(cds)$assigned_cell_type != "Failed QC" | is.na(colData(cds)$assigned_cell_type)]
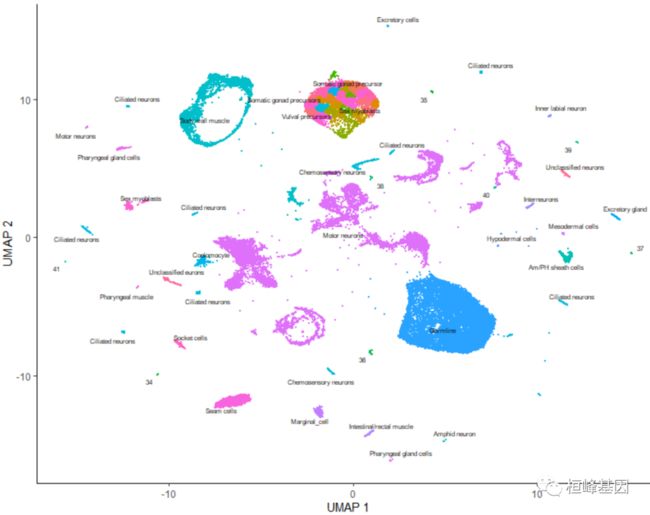
plot_cells(cds, group_cells_by = "partition", color_cells_by = "assigned_cell_type",
labels_per_group = 5)
2. Automated annotation with Garnett
上面按类型手动注释单元格的过程可能很费力,如果底层聚类发生变化,则必须重新执行。最近开发了Garnett,这是一个自动注释细胞的软件工具包。Garnett 根据标记基因对细胞进行分类。如果您已经经历了手工注释细胞的麻烦,Monocle可以生成一个标记基因文件,可以与 Garnett 一起使用。这将帮助您在将来注释其他数据集,或者在将来改进分析和更新聚类时重新注释这个数据集。
要生成一个Garnett文件,首先找到每个带注释的细胞类型所表示的顶部标记:
## Automated annotation with Garnett
assigned_type_marker_test_res <- top_markers(cds, group_cells_by = "assigned_cell_type",
reference_cells = 1000, cores = 8)
# Require that markers have at least JS specificty score > 0.5 and be
# significant in the logistic test for identifying their cell type:
garnett_markers <- assigned_type_marker_test_res %>%
filter(marker_test_q_value < 0.01 & specificity >= 0.5) %>%
group_by(cell_group) %>%
top_n(5, marker_score)
# Exclude genes that are good markers for more than one cell type:
garnett_markers <- garnett_markers %>%
group_by(gene_short_name) %>%
filter(n() == 1)
head(garnett_markers)
generate_garnett_marker_file(garnett_markers, file = "./marker_file.txt")
最终将生成如下的文本文件:
> Cell type 35
expressed: abf-2, Y45F3A.8, Y62F5A.9, F48E3.8, R05G6.9
> Cell type 36
expressed: col-12, col-89, col-130, col-159, col-167
> Cell type 40
expressed: col-14, col-97, col-113, R07B1.8, grl-6
> Cell type Body wall muscle
expressed: col-107, iff-1, plk-3, ram-2, T19H12.2
> Cell type Germline
expressed: csq-1, icl-1, F37H8.5, hum-9, F41C3.5
> Cell type Seam cells
expressed: cup-4, inos-1, Y73F4A.1, ZC116.3, aman-1
现在根据你的标记文件像这样训练一个 Garnett 分类器,现在我们已经训练了一个分类器 worm_classifier,我们可以使用它来根据类型注释 L2 细胞,以下是 Garnett 对这些细胞的注释过程:
library(garnett)
worm_classifier <- train_cell_classifier(cds = cds, marker_file = "./marker_file.txt",
db = org.Ce.eg.db::org.Ce.eg.db, cds_gene_id_type = "ENSEMBL", num_unknown = 50,
marker_file_gene_id_type = "SYMBOL", cores = 8)
cds <- classify_cells(cds, worm_classifier, db = org.Ce.eg.db::org.Ce.eg.db, cluster_extend = TRUE,
cds_gene_id_type = "ENSEMBL")
plot_cells(cds, group_cells_by = "partition", color_cells_by = "cluster_ext_type")
我们这期先分析第一部分,内容过多,一次完成有点太乱了,目前单细胞测序的费用也在降低,单细胞系列可算是目前的测序神器,有这方面需求的老师,联系桓峰基因,提供最高端的科研服务!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
有想进生信交流群的老师可以扫最后一个二维码加微信,备注“单位+姓名+目的”,有些想发广告的就免打扰吧,还得费力气把你踢出去!
References:
- G. X. Y. Zheng, et al, Massively parallel digital transcriptional profiling of single cells. Nat. Commun. 8, 14049 (2017). doi:10.1038/ncomms14049pmid:28091601