用python实现遗传算法
Hello,大家好!最近事情比较多,一个月没有写公众号了,但也积累了些不错的内容可以分享,今天就给大家介绍的是遗传算法,并用python加以实现。
在遗传算法的学习过程中,在CSDN上看到一篇已有人分享的python代码,因此直接借鉴过来,并结合《数学建模与数学实验》进行补充。(电子书和原博主链接见文末)
遗传算法目录
- 遗传算法介绍
- 操作思路
-
- 初始化
- 编码
- 个体评价
- 选择
- 交叉
- 变异
- Python实现
-
- 初始化
- 编码
- 个体评价
- 选择
- 变异
- 交叉:单点交叉&两点交叉
- 参考资料
- 获得代码
遗传算法介绍
遗传算法(Genetic Algorithm),是由美国的J.Holland教授于1975年首先提出的,是受达尔文的进化论的启发,借鉴生物进化过程而提出的一种启发式搜索算法。
生存竞争,适者生存:对环境适应度高的个体参与繁殖的机会比较多,后代就会越来越多。适应度低的个体参与繁殖的机会比较少,后代就会越来越少。
简单来说就是一个繁殖的过程,会发生基因交叉(Crossover),基因突变(Mutation),适应度(Fitness)低的个体会被逐步淘汰,而适应度高的个体会越来越多。
操作思路
初始化
初始化进化代数计数器t←0,最大进化代数T(一般100500),初始化变异概率α(一般0.00010.2)、交叉概率β(一般0.40.99),随机生成M个(一般20100)个体作为初始群体P(t)。
编码
遗传算法中,首要问题就是如何对解进行编码(解的形式),编码影响到交叉、变异等运算,很大程度上决定了遗传算法的效率。
二进制编码:每个基因值为符号0和1所组成的二进制数;
格雷编码:与二进制编码类似,连续两个整数所对应编码仅一码之差;
实数编码:每个基因值用某一范围内的一个实数来表示;
符号编码:染色体编码串中的基因值取自一个无数值含义,而只有代码含义的符号集。
个体评价
计算P(t)中各个个体的适应度值。适应度函数:也称评价函数,是根据目标函数确定的用于区分群体中个体好坏的标准,适应度函数值的大小是对个体的优胜劣汰的依据。
选择
选择运算的作用是对个体进行优胜劣汰:从父代群体中选取一些适应度高个体遗传到下一代群体中。
轮盘赌法:又称比例选择算子,个体i被选中的概率Pi与其适应度成正比:
p i = f i ∑ i = 1 N f i p_i = \frac{f_i}{\sum_{i=1}^N f_i} pi=∑i=1Nfifi
两两竞争:从父代中随机地选取两个个体,比较适应度值,保存优秀个体,淘汰较差的个体
排序选择:根据各个体的适应度大小进行排序,然后基于所排序号进行选择。
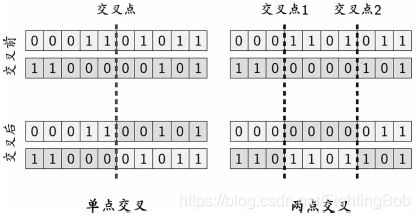
交叉
是指对两个相互配对的染色体,依据交叉概率按某种方式相互交换其部分基因,从而形成两个新的个体。
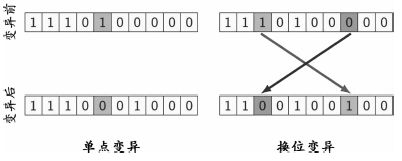
变异
是对群体中的个体的某些基因座上的基因值作变动,模拟生物在繁殖过程,新产生的染色体中的基因会以一定的概率出错。
Python实现
初始化
DNA_SIZE = 24
POP_SIZE = 100
CROSSOVER_RATE = 0.8
MUTATION_RATE = 0.005
N_GENERATIONS = 100
编码
在遗传算法中,最常用的就是二进制编码,我们可以利用np.random.randint()来实现:
pop = np.random.randint(2, size = (POP_SIZE, DNA_SIZE*2))
参数说明:
numpy.random.randint(low, high=None, size=None, dtype=‘l’)
1、low:生成的数值最低要大于等于low;当hign = None时,生成的数值要在[0, low)区间内;
2、high:如果使用这个值,则生成的数值在[low, high)区间;
3、size:输出随机数的尺寸。默认是None的,仅仅返回满足要求的单一随机数;
4、dtype:想要输出的格式。如int64、int等等。
一般来说DNA长度越长,所包含的信息越多,结果就越精确,当然还得具体问题具体分析。
虽然二进制编码实现容易,便于操作,但是对于一些连续函数的优化问题,由于其随机性使得其局部搜索能力较差,当解迫近于最优解后,由于其变异后表现型变化很大,不连续,所以会远离最优解,达不到稳定。
格雷码的好处就在于能够提高遗传算法的局部搜索能力,相比二进制精度更高。
因此,给大家介绍如何把二进制转化为格雷码(原理大家自行查阅):
def BToG(pop):
pop_g = []
for i in pop:
new = [i[0]]
for j in range(1,len(i)):
new.append(new[j-1]^i[j])
pop_g.append(new)
return np.array(pop_g)
参数说明:
“^”是按位异或运算符:当两对应的二进位相异时,结果为1,相同结果为0。
当然,对其进行编码后还需要再将其解码回二进制:
def jiema(pop_g):
pop = []
for i in pop_g:
new = [i[0]]
for j in range(1,len(i)):
if i[j] == 0:
new.append(new[j-1])
else:
if new[j-1] == 0:
new.append(1)
else:
new.append(0)
pop.append(new)
return np.array(pop)
不同的编码,后续所对应的变换和突变也会有所不同,因为格雷码和二进制码比较相似,因此在这就先介绍这两种,其它编码欢迎加好友一起探讨学习。
个体评价
通常评价函数可以由目标函数直接或间接改造得到。比如,目标函数或目标函数的倒数/相反数经常被直接用作适应度函数。一般情况下,适应度是非负的,并且总是希望适应度越大越好。比较好的适应度函数应单值、连续、非负、最大化。
def get_fitness(pop):
x1, x2 = translateDNA(pop)
pred = F(x1, x2)
return (pred - np.min(pred))
注:
translateDNA()是自定义的函数,用于将二进制码转换为数值,在测试部分会提到;F()是目标函数。
该评价函数是为了评价最大值的适应度,减去最小的结果是为了避免出现负数的情况;同理,如果我们想要最小值的适应度,就可以改成np.max (pred)-pred即可。
选择
两两竞争和排序选择都容易产生局部最优解的情况,因此遗传算法中更多使用的是轮盘赌法:
def select(pop, fitness):
idx = np.random.choice(np.arange(POP_SIZE), size=POP_SIZE, replace=True,
p=(fitness)/(fitness.sum()))
return pop[idx]
参数说明:
numpy.random.choice(a, size=None, replace=True, p=None)
1、从a(只要是ndarray都可以,但必须是一维的)中随机抽取数字,并组成指定大小(size)的数组;
2、replace:True表示可以取相同数字,False表示不可以取相同数字;
3、数组p:与数组a相对应,表示取数组a中每个元素的概率,默认为选取每个元素的概率相同。
变异
def mutation(child, MUTATION_RATE=0.003):
if np.random.rand() < MUTATION_RATE:
mutate_point = np.random.randint(0, DNA_SIZE)
child[mutate_point] = child[mutate_point]^1
交叉:单点交叉&两点交叉
单点交叉:
def crossover_and_mutation(pop, CROSSOVER_RATE = 0.8):
new_pop = []
for father in pop:
child = father
if np.random.rand() < CROSSOVER_RATE:
mother = pop[np.random.randint(POP_SIZE)]
cross_points = np.random.randint(low=0, high=DNA_SIZE*2)
child[cross_points:] = mother[cross_points:]
mutation(child)
new_pop.append(child)
return new_pop
两点交叉:
def crossover_and_mutation(pop, CROSSOVER_RATE = 0.8):
new_pop = []
for father in pop:
child = father
if np.random.rand() < CROSSOVER_RATE:
mother = pop[np.random.randint(POP_SIZE)]
cross_points_1 = np.random.randint(low=0, high=DNA_SIZE*2)
cross_points_2 = np.random.randint(low= cross_points_1, high=DNA_SIZE*2)
child[cross_points_1: cross_points_2] = mother[cross_points_1:cross_points_2]
mutation(child)
new_pop.append(child)
return new_pop
到此,遗传算法中所有的步骤我们均已实现,后面我们就以原博主所用的例子将所有步骤串起来,来进行测试。(此次测试与原博主不同的是采用格雷编码以及两点交叉遗传)

我们再拿原博主的代码跑一次做个对比。
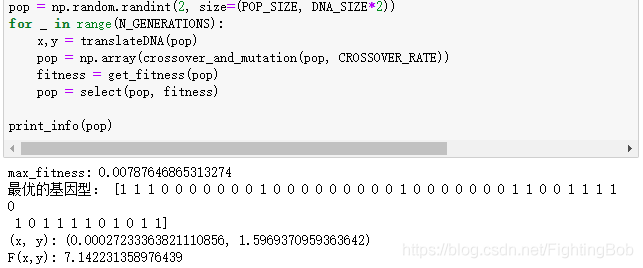
从结果中可以看出,虽然我们用了新的编码和遗传形式进行计算,但结果并没有原博主的好,所以具体问题具体分析,用算法前得多加思考,多加尝试。
参考资料
1、https://blog.csdn.net/u014484715/article/details/40046307
2、《数学建模与数学实验》
获得代码
以下是我的个人公众号,本文完整代码已上传,关注公众号回复“遗传算法”,即可获得,回复“《数学建模与数学实验》”(注意要打“《》”),即可获得该书的电子版,谢谢大家支持。
