【Deep Learning 1】GA遗传算法
本文以一个案例题目出发,详细描述了遗传算法过程,并做了两个实验复现题目
实验一:纯手打原生代码复现案例
实验二:使用第三方库scikit-opt复现案例
一、Introduction
遗传算法源自自然界生物的遗传和进化过程:通过染色体之间的选择、交叉和变异来形成。同时符合自然界优胜劣汰的规则。因此遗传算法本质上是一种全局优化搜索算法,即已知评价方程和参数范围,求解目标函数最优解。
二、 Principle
2.1 算法总体流程
算法流程
1 设计编码器和解码器
2 初始化种群
3 个体适应度评价
4 交叉运算
5 变异运算
6 选择运算
7 判断遗传进化是否达到迭代阈值
接下来我们使用GA来解决以下问题
题目:求解以下函数的最小值
题目分析:我们目标是求解该函数的最小值。限制条件为x的取值范围在-20~20
2.2 编码器和解码器
GA最先要做的事情就是对自变量X编码成字符串,每个字符类似于生物学中的染色体,而一条字符串就是一个个体。该题目中变量x整数为20-(-20)=40个。很多时候我们变量是有精度,因此我们需要对其进行扩充,如40*10=400个。
接下来我们要考虑使用什么规则来对X进行编码,最常见的方法是使用0、1构成的二进制编码
因此我们采用9位二进制数表示x值。
代码上我们设计Encoder编码器函数和Decoder解码器函数进行编码和解码。
2.2 初始化种群
初始化种群主要是随机产生几个个体。假设我们随机产生了4个个体
| 个体编码 | 数值 | 染色体 |
| 1号 | 11.7 | 100111101 |
| 2号 | -11.6 | 001010100 |
| 3号 | -6.9 | 010000011 |
| 4号 | 6.2 | 100000110 |
2.3 个体适应度评价
衡量一条染色体质量的指标即适应度,通常来说就是将个体代入目标函数的函数值
| 个体编码 | 数值 | 染色体 | f(x) |
| 1号 | 11.7 | 100111101 | 273.78 |
| 2号 | -11.6 | 001010100 | 269.12 |
| 3号 | -6.9 | 010000011 | 95.22 |
| 4号 | 6.2 | 100000110 | 76.88 |
2.4 交叉
染色体在进化的过程中会不断进行两两交叉配对。染色体具体交叉配对的过程为从几号位置染色体交换染色体
| 个体编码 | 染色体 | f(x) | 交换个体编码对象 | 交换位置 |
| 1号 | 100111101 | 273.78 | 3号 | 5 |
| 2号 | 001010100 | 269.12 | 4号 | 4 |
| 3号 | 010000011 | 95.22 | 1号 | 8 |
| 4号 | 100000110 | 76.88 | 2号 | 8 |
交换后的结果为
| 个体编码 | 染色体 | f(x) |
| 1号 | 100110011 | 228.97 |
| 2号 | 001000110 | 338.0 |
| 3号 | 010000011 | 95.22 |
| 4号 | 100000110 | 76.88 |
2.5 变异
看过《异形》的小伙伴都知道生物可能会发生变异。而变异的具体过程就是染色体发生突变。本例子中我们需要设定一个变异概率的值,某条染色体0、1互换。若变异后的超出变量x的阈值,则变异失败
| 个体编码 | 染色体 | 是否发生变异 | 变异位置 | 是否成功变异 | 变异结果 |
| 1号 | 100110011 | 是 | 3 | 是 | 100010011 |
| 2号 | 001000110 | 否 | - | - | - |
| 3号 | 010000011 | 否 | - | - | - |
| 4号 | 100000110 | 否 | - | - | - |
2.6 选择
在自然界中有着优胜劣汰的规则,因此我们需要挑选出适应度高的个体。在自然界中,有着良好基因的个体往往有更大概率存活下来,根据该规律,传统的使用的是轮盘赌算法,即适应度与被选择的概率成正比,适应度越高,被选择的概论也就越高。
但是因为所求的是最小值,即函数值越高,概率越低,因此需要特别设计一个规则。
作者想出了两种方案
第一种方案,是从适应度函数下手如下,那么求解最小值等价于求解该适应度函数的最大值,这样可以继续使用传统的轮盘赌算法
第二种方案,是从轮盘赌算法下手,f(x)概率越小,分子越小,f(x0值越小,种群适应度越高。且概率之和为1,下述的代码也是采用本方案。这里的n是初始化种群的个体数量
注意这里被选中次数之和与选中前的个数总和一致
| 个体编码 | 数值 | 染色体 | f(x) | 被选择的概率 | 被选中的次数 |
| 1 | 7.5 | 100010011 | 112.5 | 0.2056 | 1 |
| 2 | -13.0 | 001000110 | 338.0 | 0.2078 | - |
| 3 | -6.9 | 010000011 | 95.22 | 0.2889 | 1 |
| 4 | 6.2 | 100000110 | 76.88 | 0.2974 | 2 |
选择之后进行繁衍,更新表格数据为
| 个体编码 | 染色体 | f(x) | 初始f(x)(这里做对比) |
| 1 | 100010011 | 112.5 | 273.78 |
| 2 | 010000011 | 95.22 | 269.12 |
| 3 | 100000110 | 76.88 | 95.22 |
| 4 | 100000110 | 76.88 | 76.88 |
可以看到经过一轮的遗传进化之后,种群的f(x)普遍降低,种群的适应度总体提高不少
最后判断是否达到最大迭代次数,若没有,则重新回到2.3 步骤
三、Experiment
3.1 方案一:原生代码
Code
import numpy as np
from matplotlib import pyplot as plt
PRECISION = 10.0 # 自变量精度
INDIVIDUALS_NUM = 50 # 初始化个体数量
EVOLUTION_NUM = 10000 # 进化次数
LOWER_LIMIT = -20 # 染色体下限值
UPPER_LIMIT = 20 # 染色体上限值
CROSS_RATE = 0.6 # 交叉概率
MUTATION_RATE = 0.005 # 变异概率
def encoder(x):
result = []
for i in x:
i = bin(int((i + 20) * PRECISION))[2:]
for j in range(9 - len(i)):
i = '0' + i
result.append(i)
return result
def decoder(x):
result = []
for i in x:
i = int(i, 2) - 200
i = i / PRECISION
result.append(i)
return result
def initialize():
def transform(x):
return (x - 200) / PRECISION
p = np.random.randint(0, 400, size=INDIVIDUALS_NUM)
p = encoder(list(map(transform, p)))
return p
def choose(x, ada):
x, ada = np.asarray(x), np.asarray(ada)
#print('概率情况',(1-ada / ada.sum())/(INDIVIDUALS_NUM-1))
index = np.random.choice(np.arange(INDIVIDUALS_NUM), size=INDIVIDUALS_NUM, replace=True, p=(1-ada / ada.sum())/(INDIVIDUALS_NUM-1))
#print('\nChoose index:', index)
return x[index]
def threshold_limit(x):
l = []
l.append(x)
x = decoder(l)[0]
if x >= -20 and x <= 20:
return True
else:
return False
def cross(x):
result = []
for chromosome in x:
chromosome_A = list(chromosome)
if np.random.rand() < CROSS_RATE:
chromosome_B = x[np.random.randint(INDIVIDUALS_NUM)]
cross_points = np.random.randint(low=0, high=8)
# 观察交叉后的数据会不会超过自变量x的阈值
fake = chromosome_A
fake[cross_points:] = list(chromosome_B)[cross_points:]
if threshold_limit(''.join(fake)):
chromosome_A = fake
result.append(''.join(chromosome_A))
return result
def mutations(x):
result = []
for chromosome in x:
if np.random.rand() < MUTATION_RATE:
mut_points = np.random.randint(0, 8)
chromosome = list(chromosome)
# 观察变异后的数据会不会超过自变量x的阈值
fake = chromosome
fake[mut_points] = '1' if chromosome[mut_points] == '0' else '0'
if threshold_limit(''.join(fake)):
chromosome = fake
result.append(''.join(chromosome))
return result
def adaptability(list):
result = []
for i in list:
result.append(2 * pow(i, 2))
return result
def best_chr(x):
dec = decoder(x)
ada = adaptability(dec)
best_index = np.argmin(ada)
return (ada[best_index])
if __name__ == '__main__':
# 初始化种群
pop = initialize()
#print(decoder(pop))
print('Initializing Populations', pop)
best=[]
for i in range(EVOLUTION_NUM):
ada = adaptability(decoder(pop))
#print('Adaptability', ada)
pop = cross(pop)
#print('Cross', pop)
pop = mutations(pop)
#print('Chromosome',pop)
#print('hanshuzhi',decoder(pop))
pop = choose(pop, ada)
#print('Choose', pop)
best.append(best_chr(pop))
plt.plot(range(EVOLUTION_NUM), best)
plt.ylabel('f(x)')
plt.xlabel('Epoch')
plt.show()
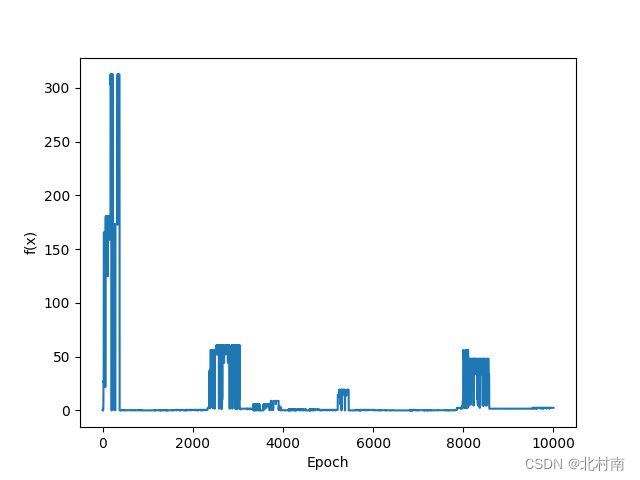
Result
这个结果非常有意思
1 目标函数值最优解为0,所求的结果大部分接近0,且最终也趋向于0,说明该算法是有效的
2 中间有几个凸集,主要是因为有染色体变异的情况,但最终还是趋向于0了
3 这其实反映了生物进化的过程,有些时候发生了生物变异,它具有很强的适应性,在某个时期具有一定的地位。但是随着生物的不断遗传进化,物竞天择的自然规律,最终,最适应的染色体始终占据了主导地位
3.2 方案二:第三方库
安装第三方库
pip install scikit-opt
Code
from sko.GA import GA
def adapt(x):
return 2 * pow(x, 2)
# func 适应度函数
# n_dim 自变量的个数
# size_pop 种群初始化个体数量
# max_iter 进化迭代次数
# prob_mut 变异概率
# lb 自变量下限
# ub 自变量上限
# precision 精度
ga = GA(func=adapt, n_dim=1, size_pop=50, max_iter=800, prob_mut=0.001, lb=-20, ub=20, precision=1e-2)
best_x, best_y = ga.run()
print('best_x:', best_x, '\n', 'best_y:', best_y)