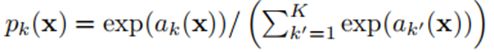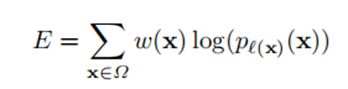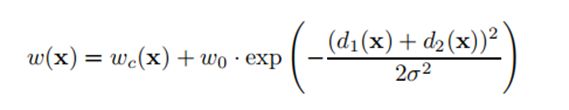【论文】U-Net: Convolutional Networks for Biomedical Image Segmentation
论文题目:U-Net: Convolutional Networks for Biomedical Image Segmentation
u-net,用于生物医学图像分割的卷积网络
1. Introduction
深度神经网络在视觉领域发展非常迅猛,人们希望使用神经网络处理生物医学图像,实现图像中目标的定位和分割。 但是对于2015年那时候来说,神经网络在生物医学图像领域的应用仍有很多问题和限制。主要有以下三个问题:
- Successful training of deep networks requires many thousand annotated training samples, which are usually beyond reach in biomedical tasks.
- A sliding-window convolutional network(the prior best method in biomedical tasks) is quite slow and redundant, and there is a trade-off between localization accuracy and the use of context.
- Another challenge in many cell segmentation tasks is the separation of touching objects of the same class.
- 第一个问题,对于大部分神经网络来说,要想获得良好的性能,需要用大量的标签数据进行训练,这对生物医学图像来说,是非常困难并且难以实现的。
- 第二个问题,用神经网络处理生物医学图像需要对图像进行像素级别的分类,而2015年那时候处理生物医学图像的最好的网络是滑动窗口卷积网络,它在每个像素的滑动窗口里训练,来对每个像素进行分类。鉴于此,他虽然能够实现分割,但是速度慢,有大量重叠冗余的训练,并且不能平衡好定位精度和全局信息的关系,因为较大的滑动窗口需要更大的池化操作,这会降低定位精度,而较小的滑动窗口限制了网络对全局信息的使用。
- 第三个问题,在分割细胞图像时,如何分离互相接触的同类细胞,这也是一个挑战。
针对于上述三个问题,这篇文章提出了一种新的网络结构和训练策略,只使用有限的标签数据集来实现高效的生物医学图像分割。 它的主要工作在以下三个方面,正好对应解决了前面所说的三个问题:
We present a new network and training strategy that relies on the strong use of data augmentation to use the available annotated samples more efficiently.
- We use excessive data augmentation by applying elastic deformations to the available little training images.
- We present a u-shaped network, which works with very few training images and propagate context information to higher resolution layers to yield more precise segmentations.
- We propose the use of a weighted loss to separate touching cells, where the separating background labels between touching cells obtain a large weight in the loss function.
- 首先:对量少的训练图像进行弹性变形来大幅扩展数据,从而解决训练数据集的限制。
- 然后:提出一个U型网络结构,也就是我们说的U-Net,通过在常规的压缩层后增加扩展层,将图像的全局信息传播到分辨率更高的层,以产生更精确的分割;并且它只需要少量的训练数据。
- 最后:给接触细胞的边界赋予更大的权重,使用加权损失函数来分离接触的细胞。
下面,详细看看这些工作是如何实现的。
2. Data augmentation
当只有很少的训练样本可用时,数据扩充对于教会网络所需的不变性和鲁棒性是至关重要的。(平移和旋转不变性,对变形和灰度值变化有鲁棒性)
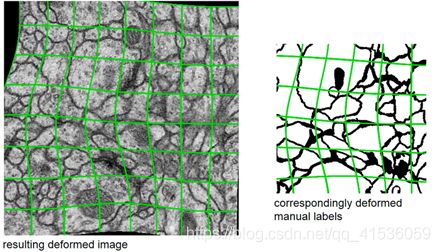
通过弹性形变处理训练数据从而扩展图像,在生物医学分割中特别重要也很常见,因为变形是生物组织中最常见的变化,可以有效地模拟真实的变形。因此可以有效增强网络的鲁棒性。
Random elastic deformations
- Generate smooth deformations using random displacement vectors on a coarse 3 by 3 grid.
- The displacements are sampled from a Gaussian distribution with 10 pixels standard deviation. Per-pixel displacements are then computed using bicubic interpolation.
这里采用随机弹性变化:在粗糙的3×3网格上使用随机位移矢量生成平滑变形,这个随机位移矢量从一个10像素标准差的高斯分布中采样,然后使用双三次插值计算像素位移。
3. Network Architecture
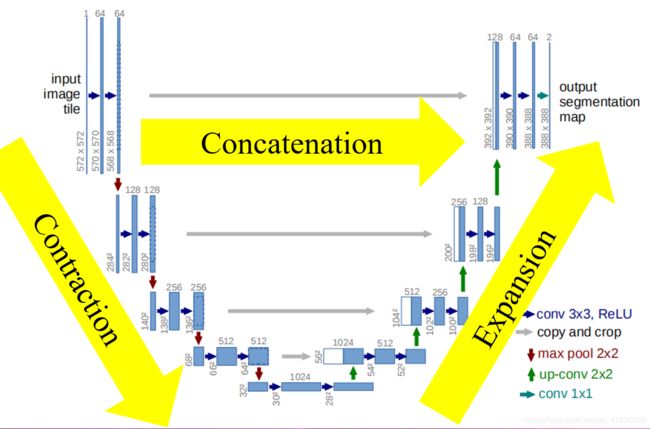
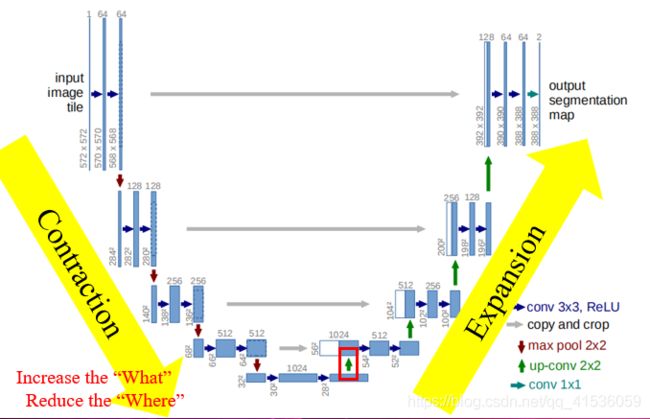
如上图所示,U-net是一个端到端的网络,输入为二维图像,输出为分割结果,每个蓝框对应一个多通道特征图,白框表示复制的特征图,上方是feature channels,左下方是x-y size。
网络由左边的收缩网络和右边的扩张网络组成,这两部分网络是完全对称的,并在相同层之间通过concatenation操作进行连接。这样的网络结构像一个U,所以称他为U-net。
收缩网络部分,是典型的卷积网络结构,每一层由两次卷积和最大池化操作组成;concatenation部分包括复制和裁剪操作;扩张网络部分,每一层由一次上卷积、一次拼接、两次卷积操作组成;最后一层用一次卷积输出结果。
下面详细介绍这个网络:
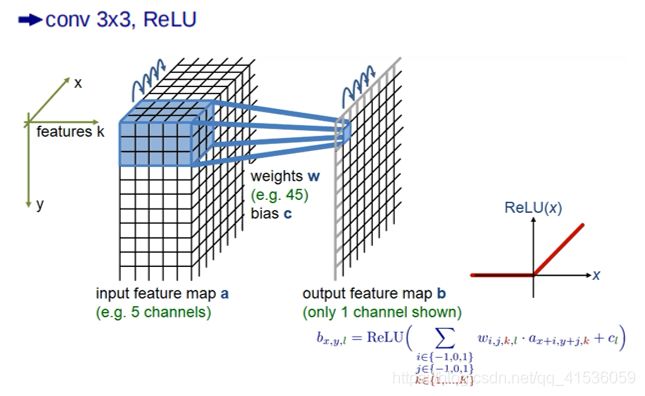
收缩网络由4层组成,它的每一层,对于输入图像,首先进行两次3x3的卷积,进行特征提取。然后,用ReLU激活函数增强非线性,避免了梯度爆炸和梯度消失问题。每次进行3x3的卷积,图像就会丢掉一个像素宽度的边界信息。因此,图像尺寸在不断减小。
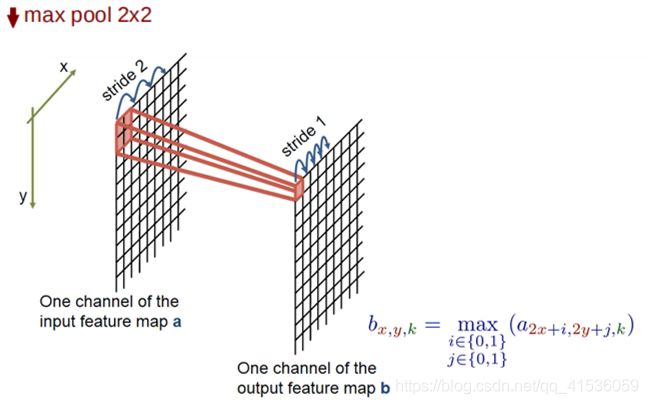
两次卷积过后,进行2x2的max pool最大池化操作,来进行特征选择和信息过滤。max-pooling提取显著特征的同时降低模型的参数,从而降低模型的过拟合。操作过后的feature map的分辨率会减小为原来的一半,并且图像的x-y 尺寸也会减半。
像这样的卷积池化操作重复四次,构成收缩网络,完成对整个图像的特征提取和分析,但是降低了图像的分辨率,影响目标位置的确定。
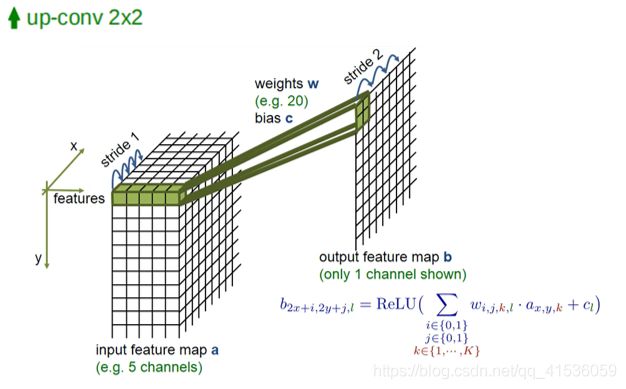
而后续的扩张网络就弥补解决这方面的缺陷。它与收缩网络是对称的,首先对输入图像进行2x2的上卷积操作来上采样,将feature map还原到原图大小。
Up-conv后的feature map的分辨率变为原来的两倍,图像的x-y 尺寸也会变为原来的两倍。这个操作和maxpool是对称的。
然后,将收缩路径中对应层的feature map复制过来,进行裁剪,并与上采样输出进行拼接,再进行两次3x3的卷积操作。
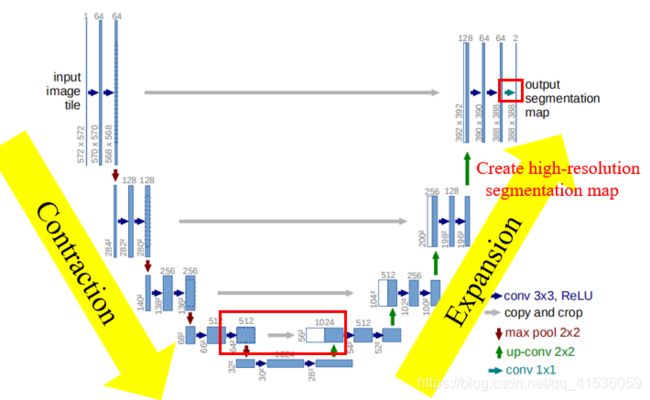
通过重复这样上卷积、连接、卷积的操作,网络可以利用收缩网络的高分辨率特征,增强输出的分辨率,并利用大量的特征通道,将图像的全局信息传播到更高的分辨率层,以实现对目标物体更精确的定位。
最后,由最后一层的1x1卷积计算每个像素的类别,是foreground还是background,输出高分辨率、高精度的分割图。
4. Training
这篇文章里用了能量函数Energy function这个概念,能量函数最早在热力学中被定义,能量函数值越小,系统趋于稳定,这和损失函数的意义相同。
在训练时,这篇文章使用加权损失函数来分离接触的细胞。
首先,使用softmax对每个像素位置各个通道的响应值进行归一化,其中,K是类别的数量,ak(x)表示特征通道k处像素x的响应值,这里的pk(x)表示近似最大值函数,ak(x)最大时,pk(x)约等于1,其他的k处pk(x)=0。
然后使用交叉熵加权损失函数在每个位置惩罚pl(x)(x)和1的偏差,其中,L(x)表示每个像素所属的真正的类别,w(x)表示该像素的权重。
这个权重是在开始训练网络之前计算出来的,权重函数的计算公式如下:
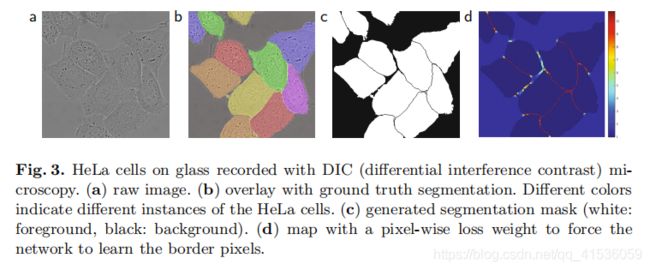
其中,wc(x)为图像的二值图,d1:当前像素到最近细胞边界的距离,d2:当前像素到第二个最近细胞边界的距离。这两个都是指定的常数。它首先通过形态学方法处理分割真值,得到包含接触细胞边界的二值图,然后用类似正太分布函数计算当前像素与最近细胞边界和次近细胞边界的距离,来增大边界像素的权值,减小非边界像素的权值。
通过这个公式,给接触细胞的边界赋予了更大的权重,使网络能够区分出图像中接触细胞之间的小边界。
5. Experiments
下面通过一些实验,来评估u-net的性能。
 |
 |
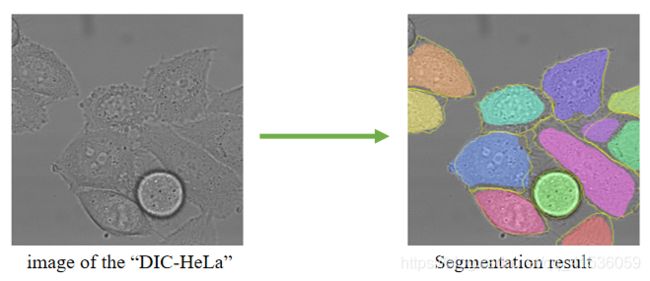
| PhC-U373 | DIC-HeLa |
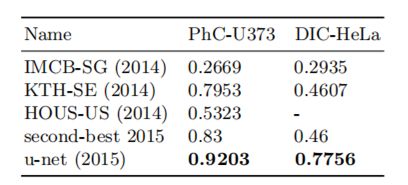
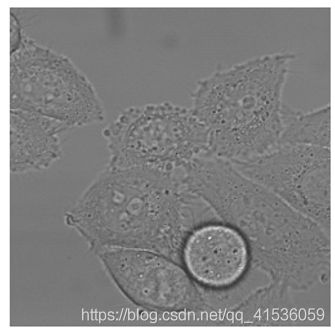
论文里使用了两个数据集,分别是胶质母细胞瘤细胞PhC-U373、宫颈癌细胞DIC-HeLa,他们分别有35张、20张标签图像数据。
在这样少量的数据集上训练u-net,并使用IoU交并比来计算分割精确度。下面表格中,在两个数据集上,u-net的IoU分别为0.92、0.77,相较于当时其他算法,它的准确率遥遥领先。
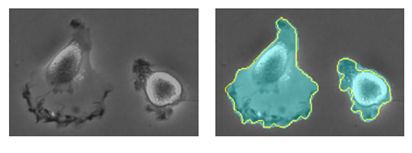
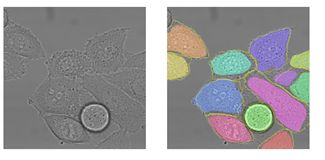
下面这些分割示例图也可以看出,对于边界不明显、含有无关内部边界、形状变化大、和背景相似度高、相互接触的细胞结构,u-net的分割结果还是比较好的。
Segmentation of a 512x512 image takes less than a second on a recent GPU(2015)
并且在那时候(2015)的GPU上分割图像用时不到一秒,U-net的速度是挺快的。
6. Conclusion
- The U-net architecture consists of a contracting path to capture context and a symmetric expanding path that enables precise localization.
- A pixel-wise loss weight map is introduced to force the network to learn the border pixels.
- Thanks to data augmentation with elastic deformations, it only needs very few annotated images and has a very reasonable training time of only 10 hours on a NVidia Titan GPU (6 GB).
- The u-net architecture achieves very good performance on very different biomedical segmentation applications.
总结u-net的工作,分为以下几点:
- u-net网络结构包含收缩部分和扩展部分,分别用来提取特征信息和提高定位精度;
- 网络使用像素级损失函数来学习接触细胞的边界;
- 使用弹性形变来扩充数据,解决生物医学图像标签数据集量少的问题,并且提高了训练速度;
- u-net能够应用在很多不同的医学图像分割中。