聚类算法及其应用
1. 聚类算法都是无监督学习吗?
什么是聚类算法?聚类是一种机器学习技术,它涉及到数据点的分组。给定一组数据点,我们可以使用聚类算法将每个数据点划分为一个特定的组。理论上,同一组中的数据点应该具有相似的属性和/或特征,而不同组中的数据点应该具有高度不同的属性和/或特征。聚类是一种无监督学习的方法,是许多领域中常用的统计数据分析技术。
常用的算法包括K-MEANS、高斯混合模型(Gaussian Mixed Model,GMM)、自组织映射神经网络(Self-Organizing Map,SOM)
2. k-means(k均值)算法
2.1 算法过程
K-均值是最普及的聚类算法,算法接受一个未标记的数据集,然后将数据聚类成不同的组。
K-均值是一个迭代算法,假设我们想要将数据聚类成 n 个组,其方法为:
- 首先选择个随机的点,称为聚类中心(cluster centroids);
- 对于数据集中的每一个数据,按照距离个中心点的距离,将其与距离最近的中心点关联起来,与同一个中心点关联的所有点聚成一类。
- 计算每一个组的平均值,将该组所关联的中心点移动到平均值的位置。
- 重复步骤,直至中心点不再变化。
用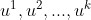 来表示聚类中心,用(1),(2),…,()来存储与第个实例数据最近的聚类中心的索引,K-均值算法的伪代码如下:
来表示聚类中心,用(1),(2),…,()来存储与第个实例数据最近的聚类中心的索引,K-均值算法的伪代码如下:
Repeat {
for i = 1 to m
c(i) := index (form 1 to K) of cluster centroid closest to x(i)
for k = 1 to K
μk := average (mean) of points assigned to cluster k
}
算法分为两个步骤,第一个 for 循环是赋值步骤,即:对于每一个样例,计算其应该属于的类。第二个 for 循环是聚类中心的移动,即:对于每一个类,重新计算该类的质心。
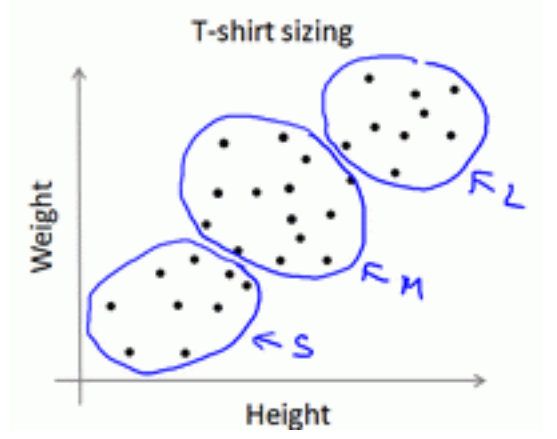
K-均值算法也可以很便利地用于将数据分为许多不同组,即使在没有非常明显区分的组群的情况下也可以。下图所示的数据集包含身高和体重两项特征构成的,利用 K-均值算法将数据分为三类,用于帮助确定将要生产的 T-恤衫的三种尺寸。
2.2 损失函数
K-均值最小化问题,是要最小化所有的数据点与其所关联的聚类中心点之间的距离之和,因此 K-均值的代价函数(又称畸变函数 Distortion function)为:
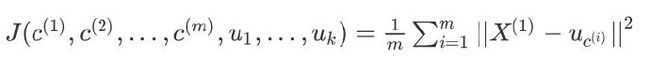
2.3 k值的选择
在运行 K-均值算法的之前,我们首先要随机初始化所有的聚类中心点,下面介绍怎样做:
- 我们应该选择 < ,即聚类中心点的个数要小于所有训练集实例的数量。
- 随机选择个训练实例,然后令个聚类中心分别与这个训练实例相等K-均值的一个问题在于,它有可能会停留在一个局部最小值处,而这取决于初始化的情况。
为了解决这个问题,我们通常需要多次运行 K-均值算法,每一次都重新进行随机初始化,最后再比较多次运行 K-均值的结果,选择代价函数最小的结果。这种方法在较小的时候(2–10)还是可行的,但是如果较大,这么做也可能不会有明显地改善。
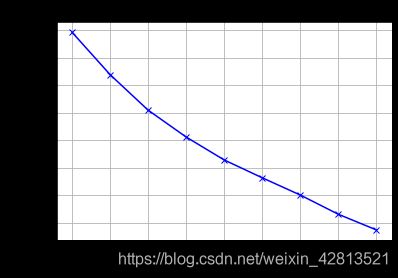
没有所谓最好的选择聚类数的方法,通常是需要根据不同的问题,人工进行选择的。选择的时候思考我们运用 K-均值算法聚类的动机是什么。有一个可能会谈及的方法叫作**“肘部法则”**。关 于“肘部法则”,我们所需要做的是改变值,也就是聚类类别数目的总数。我们用一个聚类来运行 K 均值聚类方法。这就意味着,所有的数据都会分到一个聚类里,然后计算成本函数或者计算畸变函数。代表聚类数字。
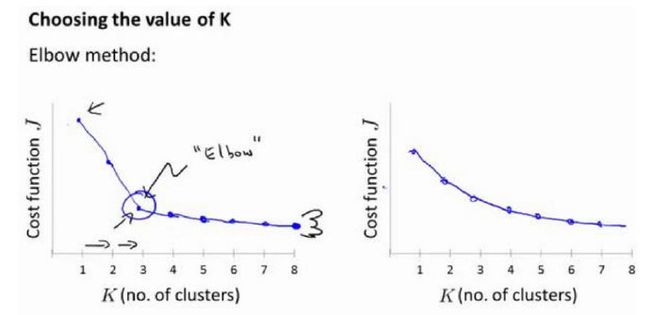
我们可能会得到一条类似于这样的曲线。像一个人的肘部。这就是“肘部法则”所做的,让我们来看这样一个图,看起来就好像有一个很清楚的肘在那儿。你会发现这种模式,它的畸变值会迅速下降,从 1 到 2,从 2 到 3 之后,你会在 3 的时候达到一个肘点。在此之后,畸变值就下降的非常慢,看起来就像使用 3 个聚类来进行聚类是正确的,**这是因为那个点是曲线的肘点,畸变值下降得很快, = 3之后就下降得很慢,那么我们就选 = 3。**当你应用“肘部法则”的时候,如果你得到了一个像上面这样的图,那么这将是一种用来选择聚类个数的合理方法。
2.4 KNN与K-means区别?
K最近邻(k-Nearest Neighbor,KNN)分类算法,是一个理论上比较成熟的方法,也是最简单的机器学习算法之一。
KNN:
1.KNN是分类算法
2.属于监督学习
3.训练数据集是带label的数据
4.没有明显的前期训练过程,属于memory based learning
5.K的含义:一个样本x,对它进行分类,就从训练数据集中,在x附近找离它最近的K个数据点,这K个数据点,类别c占的个数最多,就把x的label设为c。
K-Means
1.K-Means是聚类算法
2.属于非监督学习
3.训练数据集是无label的数据,是杂乱无章的,经过聚类后变得有序,先无序,后有序。
4.有明显的前期训练过程
5.K的含义:K是人工固定好的数字,假设数据集合可以分为K个蔟,那么就利用训练数据来训练出这K个分类。
相似点
都包含这样的过程,给定一个点,在数据集中找离它最近的点。即二者都用到了NN(Nears Neighbor)算法思想。
2.5 K-Means优缺点及改进
k-means:在大数据的条件下,会耗费大量的时间和内存。 优化k-means的建议:
-
减少聚类的数目K。因为,每个样本都要跟类中心计算距离。
-
减少样本的特征维度。比如说,通过PCA等进行降维。
-
考察其他的聚类算法,通过选取toy数据,去测试不同聚类算法的性能。
-
hadoop集群,K-means算法是很容易进行并行计算的。
-
算法可能找到局部最优的聚类,而不是全局最优的聚类。使用改进的二分k-means算法。
二分k-means算法:首先将整个数据集看成一个簇,然后进行一次k-means(k=2)算法将该簇一分为二,并计算每个簇的误差平方和,选择平方和最大的簇迭代上述过程再次一分为二,直至簇数达到用户指定的k为止,此时可以达到的全局最优。
3. 高斯混合模型(GMM)
3.1 GMM的思想
高斯混合模型(Gaussian Mixed Model,GMM)也是一种常见的聚类算法,与K均值算法类似,同样使用了EM算法进行迭代计算。高斯混合模型假设每个簇的数据都是符合高斯分布(又叫正态分布)的,当前数据呈现的分布就是各个簇的高斯分布叠加在一起的结果。
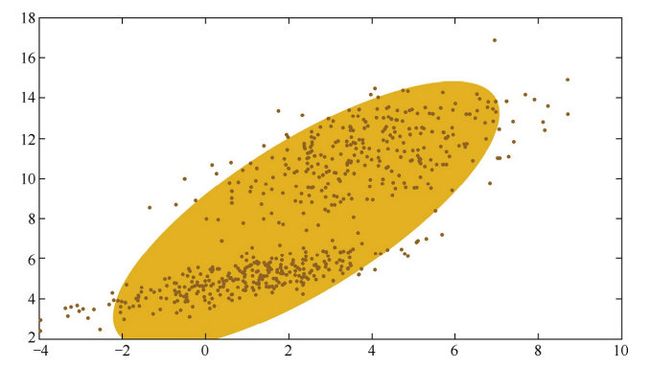
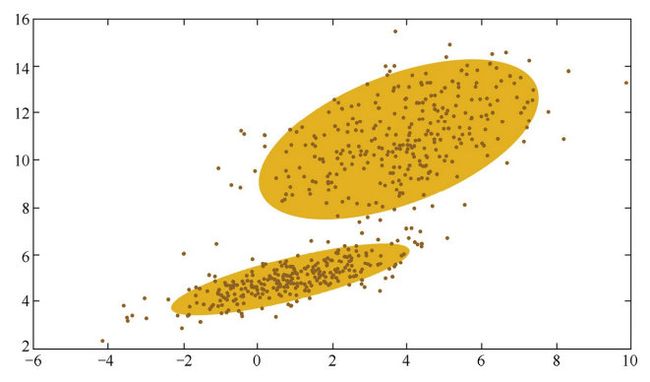
第一张图是一个数据分布的样例,如果只用一个高斯分布来拟合图中的数据,图 中所示的椭圆即为高斯分布的二倍标准差所对应的椭圆。直观来说,图中的数据 明显分为两簇,因此只用一个高斯分布来拟和是不太合理的,需要推广到用多个 高斯分布的叠加来对数据进行拟合。第二张图是用两个高斯分布的叠加来拟合得到的结果。**这就引出了高斯混合模型,即用多个高斯分布函数的线形组合来对数据分布进行拟合。**理论上,高斯混合模型可以拟合出任意类型的分布。
高斯混合模型的核心思想是,假设数据可以看作从多个高斯分布中生成出来 的。在该假设下,每个单独的分模型都是标准高斯模型,其均值 u_i和方差 sum_i是待估计的参数。此外,每个分模型都还有一个参数 pi,可以理解为权重或生成数据的概 率。高斯混合模型的公式为:
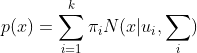
通常我们并不能直接得到高斯混合模型的参数,而是观察到了一系列 数据点,给出一个类别的数量K后,希望求得最佳的K个高斯分模型。因此,高斯 混合模型的计算,便成了最佳的均值μ,方差Σ、权重π的寻找,这类问题通常通过 最大似然估计来求解。遗憾的是,此问题中直接使用最大似然估计,得到的是一 个复杂的非凸函数,目标函数是和的对数,难以展开和对其求偏导。
在这种情况下,可以用EM算法EM算法是在最大化目标函数时,先固定一个变量使整体函数变为凸优化函数,求导得到最值,然后利用最优参数更新被固定的变量,进入下一个循环。具体到高 斯混合模型的求解,EM算法的迭代过程如下。
首先,初始随机选择各参数的值。然后,重复下述两步,直到收敛。
- E步骤。根据当前的参数,计算每个点由某个分模型生成的概率。
- M步骤。使用E步骤估计出的概率,来改进每个分模型的均值,方差和权重。
高斯混合模型是一个生成式模型。可以这样理解数据的生成过程,假设一个最简单的情况,即只有两个一维标准高斯分布的分模型N(0,1)和N(5,1),其权重分别为0.7和0.3。那么,在生成第一个数据点时,先按照权重的比例,随机选择一个分布,比如选择第一个高斯分布,接着从N(0,1)中生成一个点,如−0.5,便是第一个数据点。在生成第二个数据点时,随机选择到第二个高斯分布N(5,1),生成了第二个点4.7。如此循环执行,便生成出了所有的数据点。
也就是说,我们并不知道最佳的K个高斯分布的各自3个参数,也不知道每个 数据点究竟是哪个高斯分布生成的。所以每次循环时,先固定当前的高斯分布不 变,获得每个数据点由各个高斯分布生成的概率。然后固定该生成概率不变,根据数据点和生成概率,获得一个组更佳的高斯分布。循环往复,直到参数的不再变化,或者变化非常小时,便得到了比较合理的一组高斯分布。
3.2 GMM与K-Means相比
高斯混合模型与K均值算法的相同点是:
- 它们都是可用于聚类的算法;
- 都需要 指定K值;
- 都是使用EM算法来求解;
- 都往往只能收敛于局部最优。
而它相比于K 均值算法的优点是,可以给出一个样本属于某类的概率是多少;不仅仅可以用于聚类,还可以用于概率密度的估计;并且可以用于生成新的样本点。
4. 聚类算法如何评估
由于数据以及需求的多样性,没有一种算法能够适用于所有的数据类型、数 据簇或应用场景,似乎每种情况都可能需要一种不同的评估方法或度量标准。例 如,K均值聚类可以用误差平方和来评估,但是基于密度的数据簇可能不是球形, 误差平方和则会失效。在许多情况下,判断聚类算法结果的好坏强烈依赖于主观 解释。尽管如此,聚类算法的评估还是必需的,它是聚类分析中十分重要的部分之一。
聚类评估的任务是估计在数据集上进行聚类的可行性,以及聚类方法产生结 果的质量。这一过程又分为三个子任务。
-
估计聚类趋势。
这一步骤是检测数据分布中是否存在非随机的簇结构。如果数据是基本随机 的,那么聚类的结果也是毫无意义的。我们可以观察聚类误差是否随聚类类别数 量的增加而单调变化,如果数据是基本随机的,即不存在非随机簇结构,那么聚 类误差随聚类类别数量增加而变化的幅度应该较不显著,并且也找不到一个合适 的K对应数据的真实簇数。
-
判定数据簇数。
确定聚类趋势之后,我们需要找到与真实数据分布最为吻合的簇数,据此判定聚类结果的质量。数据簇数的判定方法有很多,例如手肘法和Gap Statistic方 法。需要说明的是,用于评估的最佳数据簇数可能与程序输出的簇数是不同的。 例如,有些聚类算法可以自动地确定数据的簇数,但可能与我们通过其他方法确 定的最优数据簇数有所差别。
-
测定聚类质量。
在无监督的情况下,我们可以通过考察簇的分离情况和簇的紧 凑情况来评估聚类的效果。定义评估指标可以展现面试者实际解决和分析问题的 能力。事实上测量指标可以有很多种,以下列出了几种常用的度量指标,更多的 指标可以阅读相关文献。
轮廓系数、均方根标准偏差、R方(R-Square)、改进的HubertΓ统计。
Demo
import os
import sys as sys
#reload(sys)
#sys.setdefaultencoding('utf-8')
from sklearn.cluster import KMeans
from sklearn import feature_extraction
from sklearn.feature_extraction.text import TfidfTransformer
from sklearn.feature_extraction.text import CountVectorizer
import matplotlib.pyplot as plt
from matplotlib.font_manager import FontProperties
from sklearn.cluster import KMeans
from scipy.spatial.distance import cdist
import numpy as np
def tfidf_vector(corpus_path):
corpus_train=[]
#利用train-corpus提取特征
target_train=[]
for line in open(corpus_path):
line=line.strip().split('\t')
if len(line)==2:
words=line[1]
category=line[0]
target_train.append(category)
corpus_train.append(words)
print ("build train-corpus done!!")
count_v1= CountVectorizer(max_df=0.4,min_df=0.01)
counts_train = count_v1.fit_transform(corpus_train)
word_dict={}
for index,word in enumerate(count_v1.get_feature_names()):
word_dict[index]=word
print ("the shape of train is ")
print (repr(counts_train.shape))
tfidftransformer = TfidfTransformer()
tfidf_train = tfidftransformer.fit(counts_train).transform(counts_train)
return tfidf_train,word_dict
def cluster_kmeans(tfidf_train,word_dict,cluster_docs,cluster_keywords,num_clusters):#K均值分类
f_docs=open(cluster_docs,'w+')
km = KMeans(n_clusters=num_clusters)
km.fit(tfidf_train)
clusters = km.labels_.tolist()
cluster_dict={}
order_centroids = km.cluster_centers_.argsort()[:, ::-1]
doc=1
for cluster in clusters:
f_docs.write(str(str(doc))+','+str(cluster)+'\n')
doc+=1
if cluster not in cluster_dict:
cluster_dict[cluster]=1
else:
cluster_dict[cluster]+=1
f_docs.close()
cluster=1
f_clusterwords = open(cluster_keywords,'w+')
for ind in order_centroids: # 每个聚类选 50 个词
words=[]
for index in ind[:50]:
words.append(word_dict[index])
print (cluster),(','.join(words))
f_clusterwords.write(str(cluster)+'\t'+','.join(words)+'\n')
cluster+=1
print ('*****'*5)
f_clusterwords.close()
def best_kmeans(tfidf_matrix,word_dict):
K = range(1, 10)
meandistortions = []
for k in K:
print (k),('****'*5)
kmeans = KMeans(n_clusters=k)
kmeans.fit(tfidf_matrix)
meandistortions.append(sum(np.min(cdist(tfidf_matrix.toarray(), kmeans.cluster_centers_, 'euclidean'), axis=1)) / tfidf_matrix.shape[0])
plt.plot(K, meandistortions, 'bx-')
plt.grid(True)
plt.xlabel('Number of clusters')
plt.ylabel('Average within-cluster sum of squares')
plt.title('Elbow for Kmeans clustering')
plt.show()
corpus_train = "corpus_train.txt"
cluster_docs = "cluster_result_document.txt"
cluster_keywords = "cluster_result_keyword.txt"
num_clusters = 7
tfidf_train,word_dict=tfidf_vector(corpus_train)
best_kmeans(tfidf_train,word_dict)
cluster_kmeans(tfidf_train,word_dict,cluster_docs,cluster_keywords,num_clusters)
from numpy import *
import numpy as np
import random
import copy
import matplotlib.pyplot as plt
import math
import matplotlib.mlab as mlab
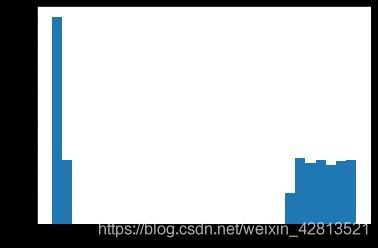
均值不同的样本
EPS = 0.0001
def generate_data():
mu1 = 2
mu2 = 6
sigma1 = 0.1
sigma2 = 0.5
alpha1 = 0.4
alpha2 = 0.6
N = 5000
N1 = int(alpha1 * N)
X = mat(zeros((N,1)))
for i in range(N1):
u1 = random.uniform(-1,1)
X[i] = u1 * sigma1 + mu1
for i in range(N-N1):
u1 = random.uniform(-1,1)
X[i+N1] = u1 * sigma2 + mu2
return X
EM算法
def GMM(X):
k = 2
N = len(X)
mu = np.random.rand(k,1)
print (str('init mu='))
print (mu)
Posterior = mat(zeros((N,k)))
sigma = np.random.rand(k,1)
print (str('init sigma='))
print (sigma)
alpha = np.random.rand(k,1)
dominator = 0
numerator = 0
print (str('init alpha='))
print (alpha)
#先求后验概率
#print (sigma)
for it in range(1000):
for i in range(N):
dominator = 0
for j in range(k):
dominator = dominator + np.exp(-1.0/(2.0*sigma[j]) * (X[i] - mu[j])**2)
#print -1.0/(2.0*sigma[j]),(X[i] - mu[j])**2,-1.0/(2.0*sigma[j]) * (X[i] - mu[j])**2,np.exp(-1.0/(2.0*sigma[j]) * (X[i] - mu[j])**2)
#return
for j in range(k):
numerator = np.exp(-1.0/(2.0*sigma[j]) * (X[i] - mu[j])**2)
Posterior[i,j] = numerator/dominator
oldmu = copy.deepcopy(mu)
oldalpha = copy.deepcopy(alpha)
oldsigma = copy.deepcopy(sigma)
#最大化
for j in range(k):
numerator = 0
dominator = 0
for i in range(N):
numerator = numerator + Posterior[i,j] * X[i]
dominator = dominator + Posterior[i,j]
mu[j] = numerator/dominator
alpha[j] = dominator/N
tmp = 0
for i in range(N):
tmp = tmp + Posterior[i,j] * (X[i] - mu[j])**2
#print tmp,Posterior[i,j],(X[i] - mu[j])**2
sigma[j] = tmp/dominator
#print (tmp)
#print (dominator)
#print (sigma[j])
if ((abs(mu - oldmu)).sum() < EPS) and \
((abs(alpha - oldalpha)).sum() < EPS) and \
((abs(sigma - oldsigma)).sum() < EPS):
print (str('final mu='))
print (str(mu))
print (str('final sigma='))
print (str(sigma))
print (str('final alpha='))
print (str(alpha))
print (it)
break
X = generate_data()
plt.hist(X, 30, normed=True)
plt.show()
GMM(X)