图神经网络用于RNA-蛋白质相互作用的新预测
《De novo p rediction of RNA-protein interactions with Graph Neural
Networks》
- 时间:2021年9月28日
- 作者:Viplove Arora and Guido Sanguinetti
- DOI:https://www.biorxiv.org/content/10.1101/2021.09.28.462100v1
- 发表期刊:还未定
Abstract
RNA结合蛋白(RNAbindingProteins,RBPs)是基因表达的关键协同和转录后调节因子,在许多生物学过程中起着至关重要的作用。像CLIP-SEQ的实验方法已经能够识别选定蛋白质的转录组范围的RNA-蛋白质相互作用,然而这些技术的时间和资源密集性要求开发计算方法来补充他们的预测。
1 Introduction
RNA-蛋白质相互作用是基因表达调控的基础。RNA结合蛋白(RBPs)在RNA的剪接、加工、输出、定位和翻译调控中起着关键作用。尽管RNA-蛋白质相互作用很重要,但与参与转录启动和调控的DNA-蛋白质相互作用相比,RNA-蛋白质相互作用的研究仍然相对较少。许多具有以前未被怀疑的RNA结合特性的蛋白质仍在被发现,超过2000种人类蛋白质已被实验确定与RNA结合。然而,由于在实验上确定单个蛋白质和单个转录本之间的相互作用是困难的,因此已知其相互作用RBPs的数量要少得多。
RNA-蛋白质相互作用研究的一个重大突破是高通量技术的发展,如CLIP-SEQ(交联和免疫沉淀,然后进行下一代测序。CLIP-SEQ能够分离RBPs及其结合的RNA片段,这与CHIP-SEQ用于确定转录因子蛋白结合的DNA区域的方式非常相似。由于技术的改进,如eCLIP协议的开发,大量的RBP结合位点正在得到验证。尽管取得了这些进展,但实践和概念上的障碍意味着,我们距离全面绘制RNA-蛋白质相互作用网络还很远。首先,这种网络本质上依赖于条件(例如,仅仅因为特定的转录本可能在不同的条件下存在或不存在)。其次,实验确定的相互作用不可避免地会有噪音,这意味着假阳性和假阴性结果都有可能。因此,需要计算方法来通过过滤噪声和预测新条件以及新的RBPs的相互作用来补充实验技术。在这里,我们考虑了采用机器学习的观点来预测RNA-蛋白质相互作用(RPI)对的问题,其中一个模型是在当前验证的相互作用上进行训练的,使用RNA和蛋白质序列作为输入。
目前的大多数预测方法都集中在预测RNA中特定蛋白的结合位点这一较窄的问题上,通常结合了目标转录本的序列和二级结构。由于过去缺乏大规模的Clip-Seq数据集,大多数预测RNA-蛋白质相互作用对的方法只在小数据集上接受过培训。RPIseq使用RNA和蛋白质的序列信息,以支持向量机和随机森林作为分类器来预测相互作用。CatRAPID组学使用序列的物理化学性质在全基因组范围内预测RNA-蛋白质相互作用。也有人提出了基于深度学习的方法,但由于数据匮乏,它们没有以端到端的方式进行训练,通常依赖于高级特征工程。
在本文中,我们建议利用新的大规模eCLIP数据集将RNA-蛋白质相互作用的预测问题转移到网络层面, 即试图以端到端的方式预测特定条件下生物体中RNA-RBP相互作用的整个网络。我们使用图神经网络来预测RNA-蛋白质相互作用,从预测单个蛋白质靶标的范式转向利用整个网络信息。我们的模型使用ENCODE项目一部分生成的CLIP-seq数据进行训练,以预测RPI网络中的缺失环节。我们还表明,该模型可以用来预测以前未见过的蛋白质之间的相互作用,以及将知识传输到在不同生物条件下观察到的网络。我们通过利用蛋白质基于序列的特征之间的相似性来引发以前未见过的蛋白质的嵌入来实现这一点。
2 Materials and methods
2.1 Dataset
CLIP-SEQ实验可以提供RPBs的全基因组结合位点。为了检索这些结合位点,首先将CLIP-SEQ读数映射到参考基因组,然后使用峰值调用软件识别读数的峰值。这些峰对应于基于某个预定截止点的RBP结合位点,可以用来识别蛋白质结合的RNA集。
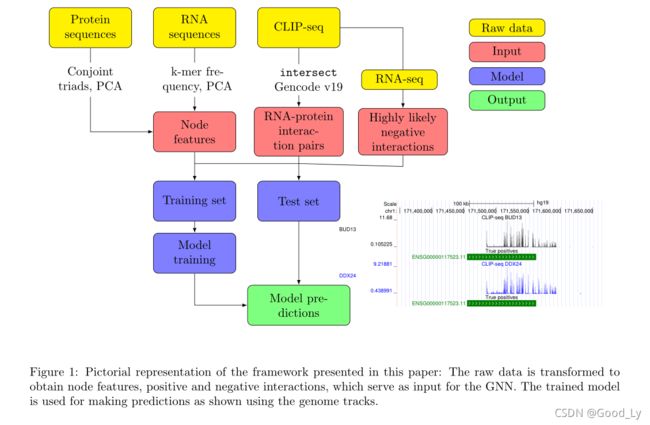
为了构建基准数据集,我们使用了作为ENCODE项目第三阶段的一部分产生的两个细胞系(HepG2和K562)的eCLIP数据集。我们使用不可再现性发现率(IDR)框架从eCLIP数据的两个副本中鉴定出的高度可重复性的峰来获得结合区。对应于结合位点的基因是通过使用床具交集功能(基因组特征之间预先定义的最小重叠,在本研究中使用50%)与人类基因组(使用GENCODE V19)相交获得的。对eCLIP数据集中的每个蛋白质重复这个过程,我们得到了特定细胞系的RNA-蛋白质相互作用的二部网络G=(V,E,X)。节点集合V=R∪P,其中R是RNA集合,P是RBP集合,X是节点特征矩阵。
K562细胞系的最终图由14665个节点(120个蛋白质和14545个RNA)组成,蛋白质和RNA之间有144527个相互作用。蛋白质的平均(出局)度为1204.39,标准差为1304.64,核糖核酸(进出度)的平均和标准差分别为9.94%和10.27%。对于HepG2细胞系,该图包含15018个节点(103个蛋白质和14915个RNA)和145509条边。蛋白质的平均(出局)度为1412.71,标准差为1380.69;核糖核酸(进出度)的平均和标准差分别为9.76%和10.03%。图S1绘制了两个细胞系中RPI网络的蛋白质和RNA度分布。
2.1.1 Negative Interactions
CLIP-SEQ实验提供了来自读数峰值的结合位点的信息,但它们不提供任何关于未结合位点的信息。在CLIPseq数据中,一个众所周知的问题是由于用于实验的细胞系中没有转录本或转录本浓度较低而导致假阴性。适当定义负样本对于训练机器学习算法很重要。负面互动在损失函数的计算中起着至关重要的作用,以及等人最近的研究表明,适当选择负样本可以提高链路预测器的性能。这对于二部网络来说变得更加重要,在这种网络中,随机采样两个未连接的节点可能会产生数据中不存在的edge-type(例如RNA-RNA)。
为了解决这个问题,以下两种策略通常被用来从CLIP-SEQ数据中构建阴性样本:(I)使用没有结合位点的区域作为负样本,或者(II)使用阳性序列中随机洗牌的核苷酸作为负样本。我们通过利用RNA-seq转录本丰度数据来增强第一种策略,以识别在细胞系中具有每百万转录本(TPM)计数超过中值但仍然没有与相应蛋白质的任何峰的RNA来定义阴性样本。这一策略允许模型从真实RNA序列的极有可能的未结合位点学习,并缓解了上述假阴性的问题。使用RNA-SEQ,我们为每个蛋白质识别可靠的非相互作用RNA,并因此使用这些阴性样本来定义训练和测试集。
2.1.2 Node Features
近年来,神经网络已成为将神经网络应用于图领域不可或缺的工具。。GNN体系结构在节点、边和图特征上创建了一个非线性置换不变变换函数,该函数可以被优化以执行下游学习任务。GNN的邻域聚集过程使我们能够捕捉网络数据中的层次非线性,从而学习图中节点的低维嵌入。从整个RNA-蛋白质相互作用网络中学习的能力使我们能够建立单一的端到端模型来预测RNA-蛋白质相互作用。此外,GNN体系结构便于从遥远的邻居(如其他蛋白质)聚集信息,从而学习更好的节点表示。
许多现有的GNN架构可以直接转换为消息传递神经网络的框架,其中每个节点(使用函数Mk(·))发送和接收来自其邻居的消息,并且随后(使用函数UK(·))更新其自己的节点状态:

其中h(K)v是层k中节点v的隐藏表示, M k ( ⋅ ) 和 U K ( ⋅ ) M_k(·)和U_K(·) Mk(⋅)和UK(⋅)是具有可学习参数的函数,N(V)是节点v的邻域集合,和表示可以添加到消息传递过程中的其他功能(如边缘功能)。节点特征(如果可用)可以用作节点的初始隐藏表示 h v ( 0 ) = x v h^{(0)}_{v}=x_v hv(0)=xv。在K个消息传递层之后,为每个节点v生成节点嵌入 z v 作 z_v作 zv作为最终输出,然后可以将其用于节点、链路或图级预测任务。函数 M k ( ⋅ ) 和 U K ( ⋅ ) M_k(·)和U_K(·) Mk(⋅)和UK(⋅)跨节点共享参数,但每个节点与其邻居定义的单个计算图相关联。在本文中,我们使用K=2的图卷积网络(GCN)作为GNN模型。GCN弥合了在基于图的数据上执行卷积的频谱和空间方法之间的差距。GCN的图卷积运算可以写成:
![]()
其中, ˆ A = A + I ˆA=A+I ˆA=A+I是具有自环的邻接矩阵, ˆ D ˆD ˆD是对应于 ˆ A ˆA ˆA的对角度矩阵。 H ( K ) H^{(K)} H(K)是包含层k处节点的隐藏表示的矩阵, H ( 0 ) = X H^{(0)}=X H(0)=X, Θ ( K ) Θ^{(K)} Θ(K)是层k处的模型参数,σ(·)是逐元素激活函数。比较公式1和公式2,对于GCN,消息和更新函数采用以下形式:
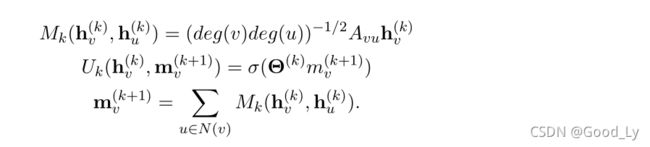
为了向模型添加一些生物学上下文,我们考虑GCN的以下不同设置:
RNA-SEQ实验提供了关于给定生物样本中所有RNA的存在和数量的高分辨率信息。RNA-seq可以告诉我们细胞中哪些基因被激活,以及它们的转录水平是什么。因此,人们会认为RNA的丰度可以很好地反映RNA与蛋白质结合的能力。在GCN的Seq变体中,我们将==log(1+TPM)==附加到GNN层后获得的RNA的最终节点嵌入Z(对于所有蛋白质设置为-1)。然后,修改后的嵌入被用于计算损失和预测相互作用。
如第2.1.1节所述,负抽样在机器学习模型的训练中起着重要作用。在GCN的默认训练机制中,从一组可能的负相互作用中均匀地随机抽样负边缘,这可能导致对于低程度的蛋白质采样更多负相互作用的情况。为了解决这个问题,我们考虑了结构化负抽样(SN),即对蛋白质的负相互作用进行与其程度成比例的抽样。
2.3 Link prediction and evaluation metrics
目前关于生物网络中相互作用的知识往往是不完整的,这使得预测缺失的相互作用成为一项重要的任务。链接预测通常被定义为半监督学习问题,其中网络中的已知链接被用来预测额外的交互。鉴于RNA-蛋白质相互作用的重要性以及与实验方法相关的挑战,使用计算模型预测潜在的相互作用可以补充我们目前的知识。虽然大多数现有的研究都集中在transductive链接预测(两个节点都在图中),但inductive(或样本外)链接预测对于新的蛋白质来说可能被证明是非常有价值的。
在用于链接预测的GNN中,使用第2.2节中描述的消息传递过程来计算单个节点表示 z u z_u zu,随后可以使用函数 p u v = f ( z u , z v ) p_{uv}=f(z_u,z_v) puv=f(zu,zv)来预测链接(u,v)的概率。该模型可以被训练成使用二进制交叉熵损失来最大化重构真实邻接矩阵A的可能性:
![]()
在链路预测问题中,将网络划分为训练子网络和测试子网络并不是一件容易的事。在进行边的训练测试分裂时,我们需要确保训练网络中的每个节点都有一个非空的邻居集,以便GNN可以使用等式1中所示的消息传递过程来学习适当的表示。为了确保这一点,在确保每个RNA保持在训练网络中的连接的同时,对每个RNA进行测试边采样。
链接预测是一项二进制分类任务,可以使用不同的度量来评估算法的性能。这些指标可以分为两大类:固定阈值指标和阈值曲线。在研究背景下,我们通常没有合理的阈值,这就是为什么阈值曲线和总结它们的标量度量在文献中被广泛使用。在本文中,我们使用了接收机工作特性下的面积AUROC和平均精度(Ap)评估链接预测任务中不同方法的性能。接收器操作特征(ROC)曲线表示在不同判决边界阈值下真阳性和假阳性之间的性能权衡。AUROC反映随机选择的正实例出现在随机选择的负实例之上的概率。AP总结了精确度-召回率曲线,对于高度不平衡的数据集来说是一个更好的衡量标准。AP可以使用以下公式计算:
其中βN 和αN是 n t h n^{th} nth临界值的精确度和召回率。
2.4 Prediction tasks
在三种不同的情况下对模型进行了评估:(I)我们假设3.1节中缺少某些百分比的RNA-蛋白质相互作用;(Ii)在第3.2节中,我们进行了遗漏一个蛋白质的实验,假设剩余蛋白质的全部相互作用信息可用;(Iii)在第3.3节中,我们将RNA-蛋白质相互作用的学习从源细胞系转移到目标细胞系。在场景(Ii)和(Iii)中,我们使用蛋白质基于序列的特征之间的相似性来诱导以前未见过的蛋白质的嵌入。使用机器学习术语,我们将场景(I)称为transductive学习(因为需要预测的集合是图的一部分),将场景(Ii)称为Inductive 学习,将场景(Iii)称为迁移学习。
3 Results and discussion
我们的模型是使用从ENCODE项目中提取的RNA-蛋白质相互作用的大规模eCLIP数据集进行训练的(见第2.1节)。我们使用RNAcommender作为基线模型,与我们基于GNN的方法进行比较。RNAcommender是一个推荐系统,它能够利用在其他蛋白质上进行的高通量实验获得的相互作用信息,为未探索的RBPs建议全基因组RNA目标。在我们的评估中,我们使用了2.1.2节中描述的功能,而不是RNAcommender最初实现中使用的高级功能工程(蛋白质结构域组成和RNA预测的二级结构)。对于GCN,我们考虑第2.2节中描述的设置:(I)原版环境中的GCN;(Ii)Seq,具有RNA-Seq的GCN;(Iii)SN,具有结构化负采样的GCN;以及(Iv)Seq.SN,具有RNA-Seq和结构化负采样的GCN。
3.1 Transductive link prediction
对于模型的第一次评估,我们考虑从RPI网络中移除不同百分比的正边缘的情况。根据第2.1.1节中的定义,测试集的相同数量的负相互作用可以随机均匀抽样,也可以与每种蛋白质的程度成正比。第二种设置在实践中要困难得多,因为网络必须隐式地从训练数据中学习学位信息。另一方面,较难的设置可能更能代表真实的生物缺失数据。我们使用验证集上的AUROC来选择最佳模型。在本节中,我们只给出测试集中负交互的“硬”设置的结果。我们将所有实验运行10次,并报告平均结果和标准偏差,在表格中用粗体突出显示最佳结果(使用两个样本t检验确定)。
需要为GCNS调优的一个超参数是节点的最终嵌入维度。在图2中,我们绘制了当最终嵌入维度不同时(隐藏维度保持在50不变)时GCN的三个不同变体的性能。虽然所有嵌入维度的所有方法都提供了明显好于随机预测的结果,但趋势显示所有方法的维度在5到10之间有一个明显的峰值;因此,我们选择10作为不同设置下所有GCN后续评估的最终嵌入大小。
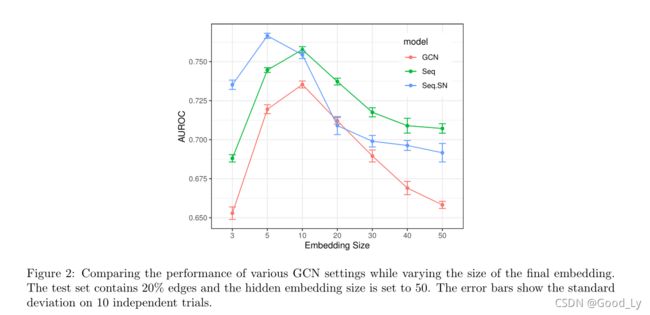
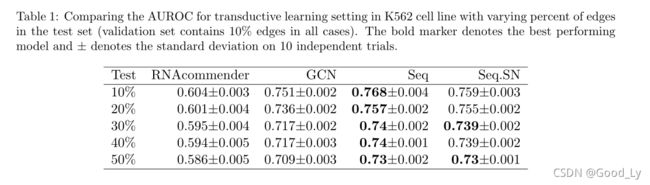
表1和表2中的结果分别显示了不同模型在K562和HepG2细胞的RPI网络上的transductive链接预测任务的性能。执行测试时,测试集中的边缘百分比从10%到50%变化。我们的结果表明,我们的基于GCN的模型在transductive链接预测任务中的表现一直明显优于RNAcommender。正如预期的那样,随着我们增加测试集中的边数(从而减少训练集的大小),性能会下降,然而,对于所有大小的训练集,GCN方法(在其所有变体中)的性能保持在70%AUROC以上。在不同的GCN设置中,我们看到使用RNAseq在所有情况下都一致地提高了模型的预测性能(这种效果在使用基于word2vec的节点特性时更加明显,如表S4所示)。
当随机选择负测试边沿时,较简单设置的结果显示在补充信息中。在这里,我们看到RNACommender和GCN方法都有了很大的改进,后者实现了超过90%的测试准确率(在某些情况下是相当高的)。
3.2 Inductive link prediction
对RNA-蛋白质相互作用进行从头预测的能力是为这个问题开发计算模型的最大动机之一。这就是GNN社区中的Inductive (或样本外)链接预测问题。这种分析特别有价值,因为模型预测可以作为目前无法获得高通量数据的蛋白质的代理。在这种情况下,我们通过删除测试和验证蛋白质的相互作用数据来创建训练网络G,但假设该模型可以访问未知蛋白质的原始特征。然后,训练好的模型使用归一化的方法计算新蛋白质的嵌入率其中P是训练网络G中的蛋白质集合。我们使用测试集合中的单个蛋白质进行实验,同时选择与测试蛋白质特征相似性最高的蛋白质作为验证蛋白质。这可能会在测试和验证集中的正面和负面交互中造成潜在的类不平衡。因此,我们还使用第2.3节中介绍的AP度量,它是不平衡数据集的更好度量。
图3比较了在诱导链接预测设置中不同模型在整个蛋白质集合上的性能。每个框绘制了K562细胞系中蛋白质的平均AUROC/AP分布(每个蛋白质重复10次)。结果表明,在K562细胞系中,平均而言,GCN的所有变异都优于RNAcommender。更具体地说,基于距离相似性的GCN在AUROC和AP上的蛋白质性能分别比RNACommender高93.33%和87.5%。在GCN的不同设置中,我们观察到相似性函数的选择对模型性能的影响非常小,甚至在最终嵌入后添加RNA-seq似乎也没有显著提高模型性能(尽管我们确实在HepG2细胞系中看到了一些改善,见图S2)。
图4显示了在K562细胞系的诱导链预测任务中,我们的模型对两种蛋白质BUD13和DDX24的预测标注的基因组轨迹。图4a和4b示出了对应于预测的真阳性和真阴性的示例性区域;正如预期的那样,真阳性对应于具有强结合信号的区域,而真阴性显示完全没有信号。图4d和4e显示了错误预测的示例(分别为假阳性和假阴性):这两个示例都显示了适度的绑定,可能表示在高峰呼叫过程中处于边界情况的区域。这表明模型可能对应于潜在的噪声区域。这一点使用图4c和4f进行了全局说明,图4c和图4f绘制了四种结果的每个转录的读数分布。我们观察到,与其他情况相比,真阳性和真阴性预测分别具有显著更高和更低的读取次数,而该模型的假阳性和假阴性预测具有中等读取量,潜在地对应于噪声区域。
3.3 Transfer learning
ENCODE数据集包括223个eCLIP实验,涉及两个不同的细胞系(K562和HepG2)上的150个蛋白质。这提供了执行转移学习的机会,其中可以使用从源细胞系的eCLIP数据学习的模型来预测目标细胞系中的相互作用。这可能是我们方法中最有趣的方面,因为它将允许研究人员基于关于目标条件的最小信息,在新的条件下获得对RPI网络的合理预测。
为了训练转移学习的模型,我们通过将固定百分比的随机选择的蛋白质中的所有相互作用分配到验证集来分离来自源细胞系的相互作用。以这种方式拆分数据可以让我们选择一个对以前未见过的蛋白质有更高预测能力的模型。为了使用目标细胞系创建测试集,我们只考虑与源细胞系中已经存在的RNA的相互作用。这使得我们可以专注于蛋白质的迁移学习。测试集中的负交互作用(与正交互作用的数量相同)是从目标细胞系中极有可能的负交互作用中随机均匀抽样的(3.1节中的“Easy”设置)。这是一个合理的假设,因为要采样与蛋白质程度成正比的负相互作用(“硬”设置),我们需要对其在目标细胞系中的相互作用有一个先验的了解。
据我们所知,我们是第一个探索RNA-蛋白质相互作用预测的迁移学习问题的人,这就是为什么我们在本节中只比较不同GCN设置的性能。与第3.2节中一样,我们使用公式4来计算测试和验证集中蛋白质的嵌入量。我们特别关注RNA-seq在转移学习中的使用,因为它可以提供关于目标细胞系中RNA丰度的信息。对于迁移学习,我们在训练过程中将源细胞系的TPM计数附加到RNA嵌入中,但替换为目标细胞系中的TPM计数,以做出最终预测。

表3和表4中的结果表明,即使在迁移学习模式下,GCN方法也提供了良好的预测精度,大多数情况下AUROC值都在70%以上。此外,在迁移学习中使用RNA丰富的数据似乎有一个明显的优势,因为GCN的序列变体在大多数情况下是性能最好的模型。这是直观地吸引人的,因为它表明RNA-SEQ数据清楚地传达了与RPI网络的预测相关的关于细胞状态的一些信息。然而,仍然非常令人惊讶的是,即使没有RNA-SEQ信息,GCNS也提供了良好的预测性能。为了将这一观察结果联系起来,在图5中,我们将GCN与RNA-SEQ数据的ROC曲线与我们仅假设两个RPI网络在两个eCLIP实验共享的一组蛋白质/RNA上相同(幼稚转移)得到的预测进行了比较。值得注意的是,这种朴素的传输方法的性能仅略好于随机,而在相同的误报率下比GCN预测的性能要差得多。这表明,GCN主要学习在多种不同环境中可以看到的强大的相互作用,并且这些相互作用可能是硬连接到蛋白质-RNA序列特征中的。
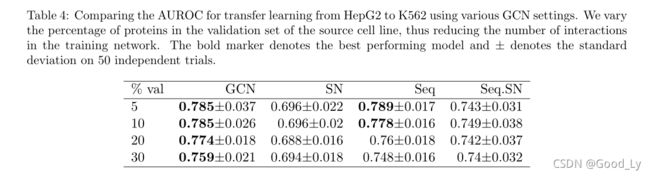
我们还观察到GCN的性能随着验证集大小的增加而降低。这意味着,通过查看来自源细胞行的更多数据,而不是仅仅过度拟合训练数据,模型可以学习得更好。
4 Conclusion
在过去的十年中,RPI的实验发现一直是研究的一个主要焦点。在经历了一段仍在开发新技术的开创期之后,最近几年人们努力实现该技术的规模和标准化,结果在去年出版了第一本大规模的人类细胞系RNA-RBP相互作用汇编。
这些技术发展要求改变RPI数据的建模方式。早期的方法中进行了审查侧重于预测单个RBPs的靶标(或结合位点),将潜在的靶标转录本视为I.I.D.。观察结果使机器学习方法的部署成为可能,例如CNN。现在,来自数百个RBP的绑定数据的可用性导致了数百个相关的预测任务,这就需要能够有效地利用这些结构的方法。本着这种精神,我们的GNN模型在不同的RBPs结合数据,将RBP结合目标的预测问题转化为对整个RPI网络的预测。
我们的实验在这一尝试中显示出相当大的希望。除了在链接预测的经典任务上大大优于最先进的竞争对手外,我们的GNN方法还在对看不见的RBP的目标进行样本外Inductive 预测方面提供了强大的预测性能。此外,我们的经验表明,GNN方法也能够执行迁移学习,即从特征良好的条件下的RPI数据开始预测不同(相关)条件下的RPI网络,这是我们所知从未尝试过的任务。
虽然我们相信GNN在RPI网络预测问题上有很大的希望,但一些未来需要改进的领域显然是开放的。首先,在我们的方法中,蛋白质(和转录本)的特征仅仅是它们的序列和它们的结合伙伴,这使得预测新蛋白质的结合伙伴的完整补充(诱导链预测)的任务变得困难。原则上,额外节点信息的可用性(例如,以蛋白质-蛋白质相互作用或本体论注释的形式)可以很容易地合并到GNN框架中,潜在地导致重大改进。另一个令人非常感兴趣的领域当然是可以改进的,那就是迁移学习。这里的问题是找出合适的协变量,这些协变量可以用来衡量不同领域之间的相似性。在本文中,我们表明,RNA-seq数据的使用有助于迁移学习任务,可能是因为它概括了细胞状态的一些信息,然而,可能会找到更合适的任务描述符,并将其集成到框架中。