疾病负担研究(GBD)——如何使用joinpoint软件计算AAPC
如需要加入GBD数据交流群,请加小编微信“Endoscopy_1991”,小编拉你进群
关于GBD数据库的实操视频已经陆续更新,如需要,大家可以关注B站的“小明学习室”。
这次是GBD相关的第10篇推文。
后台有朋友反应如何使用joinpoint软件计算AAPC。这次我们将讲解GBD数据计算AAPC的软件操作过程。
首先我们需要下载好软件,具体到官网下载(https://surveillance.cancer.gov/joinpoint/download),填写好信息后即可下载桌面软件。
接着我们准备好用于joinpoint的数据,我们同样以食管癌为示范进行操作
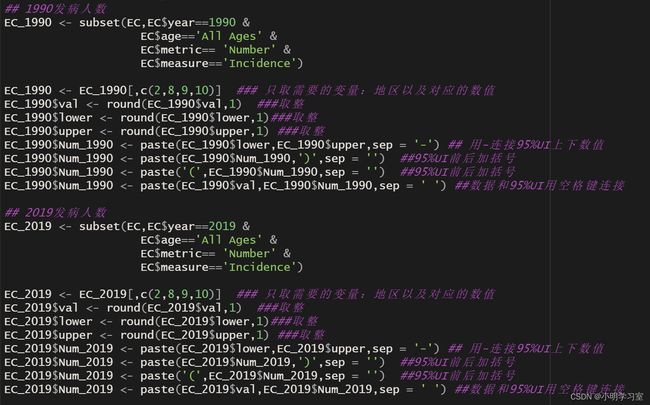
打开R语言,读取食管癌1990到2019年的发病率数据
由于joinpoint需要发病率以及标准误数据,因此我们需要得到标准误数据,我们根据可信区间的定义公式95%UI=mean+/-1.96*standard error,因此standard error= (up-lower)/(1.96*2),而且joinpoint软件要求年份按照升序排列,且一个组放在一起,因此我们计算出standard error后对EAPC数据location,year进行升序排列,并输出结果
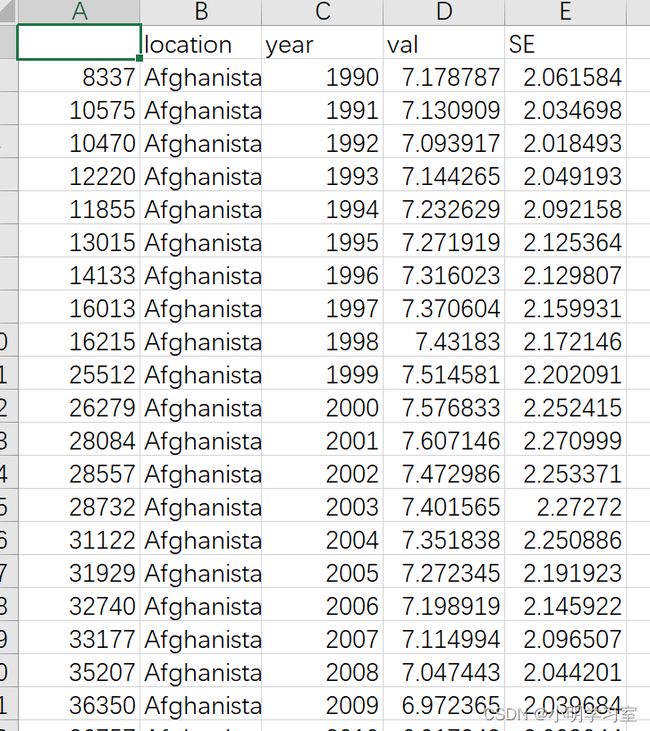
我们打开数据文件,可以看下格式
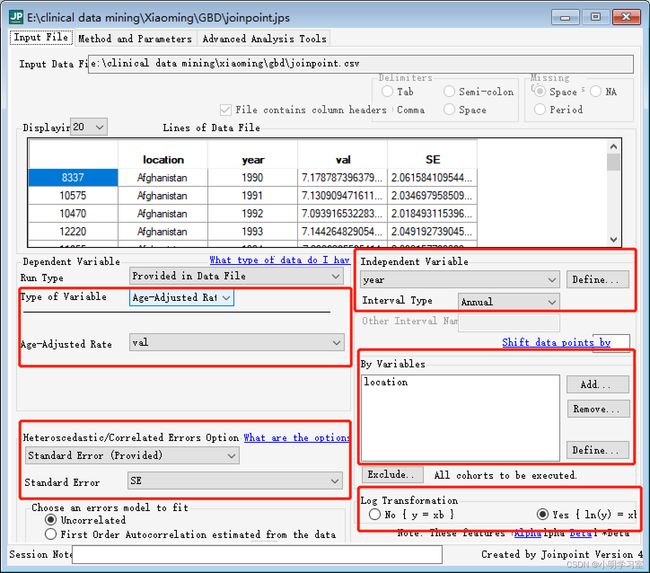
我们打开joinpoint软件,选择file目录的new session,找到joinpoint.csv文件打开
打开后即可跳出参数设置界面,按照图示中的设置:
需要说明的一点是:Joinpoint回归模型有线性模型(y=xb)和对数线性模型(ln y=xb)两种,如果因变量服从正态分布(或近似正态分布)且数据样本量较大(通常大于100)时选择线性模型;如果因变量服从指数分布或泊松分布,则宜选用对数线性模型。分析以人群为基础的肿瘤发病率和死亡率趋势时一般选择对数线性模型。GBD的数据由于每个国家的年份时间不多,数据分布多不符合正态分布,因此一般选用对数线性模型。
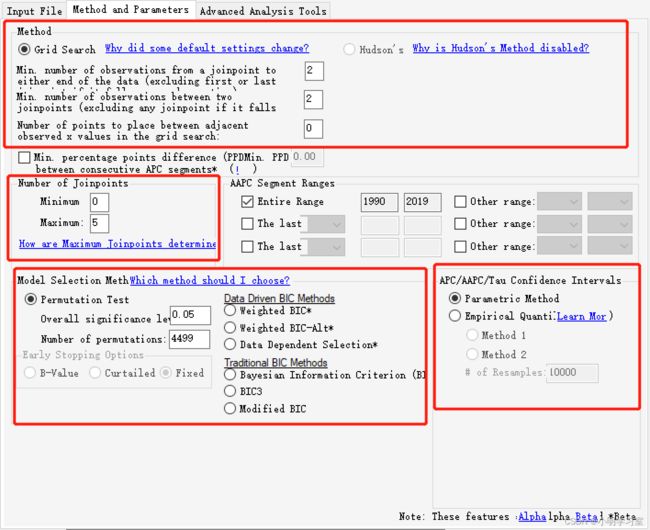
Method and Parameters设置按照默认参数设置即可。
这里讲几个计算原理:
- 建模方法:目前软件只采用网格搜索法(grid search method, GSM),Hudson′s 法的备选项目前已取消。建模原理大概是通过GSM法建立所有可能存在的区间分段函数连接点(即Joinpoint点),并计算每种可能的情况下所对应的误差平方和(sum of squares errors,SSE)和均方差(mean squared errors, MSE), 选择MSE最小的网格点为分段函数连接点,并根据选定的连接点和区间函数拟合β0、β1、δ1、... δk等方程参数。
- 模型优化:Monte Carlo置换检验(permutation test)是Joinpoint软件默认的模型优选方法。除Monte Carlo置换检验外,软件还提供了贝叶斯信息准则(bayesian information criterion,BIC)、校正贝叶斯信息准则(modified bayesian information criterion,MBIC)及相关衍生方法进行优选模型的筛选,但主要面向特殊需求的高级用户使用。我们一般选择默认的permutation test。
- 有些文章设置number of joinpoint成2个,但一般设置上限最多为5个,如果太多会导致计算时间过长。这里我们选择number of joinpoint为5个
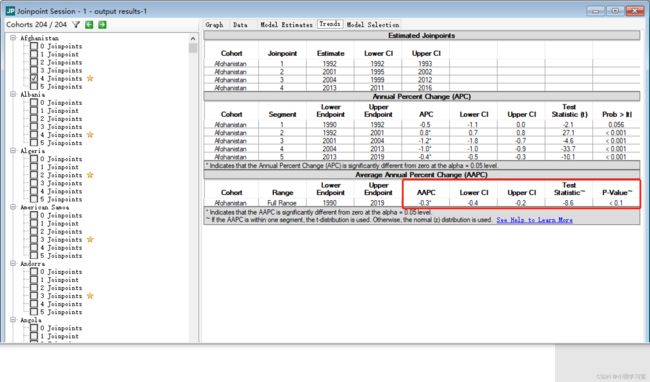
- 指标计算:年度变化百分比(annual percent change, APC)和平均年度变化百分比(average annual percent change, AAPC)及95%CI是Joinpoint模型主要结果指标。APC为因变量平均每年变化的百分比。APC用于评价分段函数各独立区间的内部趋势,或者连接点数量为0的全局趋势,如果要综合评价包含多个区间的全局平均变化趋势时就需要用AAPC。AAPC的参数计算方法是通过分段区间的跨度宽度w对各区间的回归系数进行加权计算而来
这个参数设置页面主要说明需不需要进行设置,主要用于高级分析:如果需要比较两组系列数据的分段回归方程是否等价或趋势一致性,可选用模型中的“ 成对比较分析" ( pairwise comparison ) 方法; 如果由于疾病/ 死因编码系统的改变等原因, 引起监测数据发生系统性的“ 跃变" , 但并不影响所研究“ 事件" 的潜在变化趋势,可选用其中的“ 跃变" 模型/ 可比性比例模型分析( jump model/comparability ratio(CR) model )方法。
我们点击下图的这个按钮后程序开始计算分析
运行前提示这个信息,我们直接选择确定即可
由于我们设置的最大joinpoint比较大,花的时间比较长,大家去喝杯茶回来就可以看到结果~
下图软件运行结束后的界面,左边是每个国家的发病率变化趋势,右边展示的是第一个国家发病率的趋势图,这个国家趋势图最终选择4个转折点作为计算AAPC的最终模型,
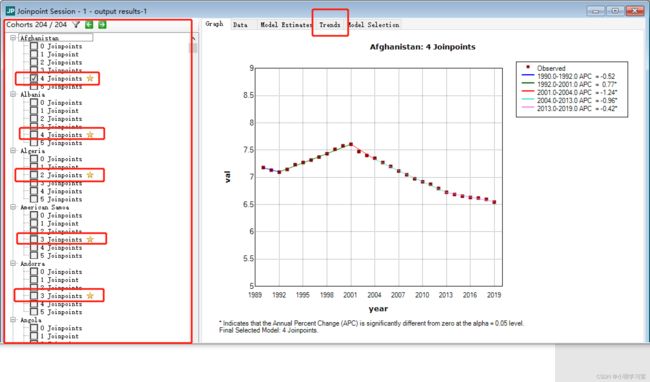 我们点击界面右侧的trends按钮后可查看APC以及AAPC结果
我们点击界面右侧的trends按钮后可查看APC以及AAPC结果
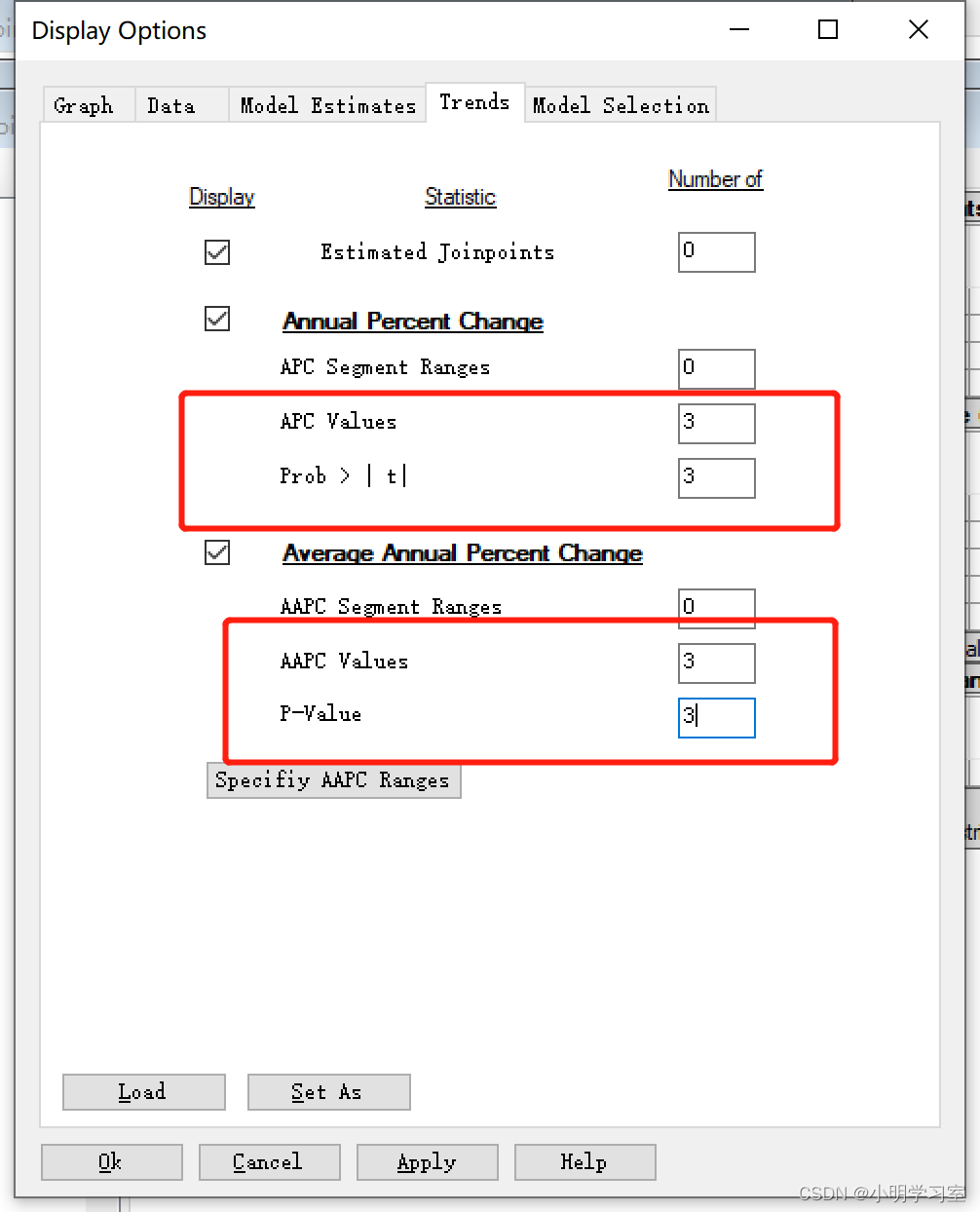
由于软件默认只保留1个小数点,因此我们设置参数,选择output菜单的options按钮,按照如图所示修改,APC、AAPC和P值保留3个小数点
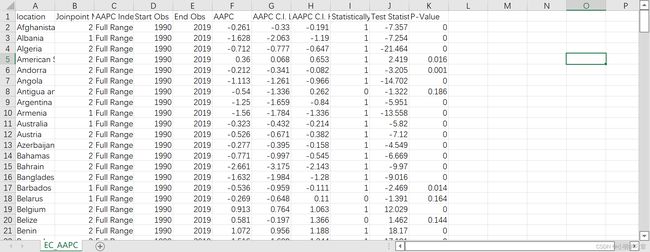
修改后的数据展示如下
我们选中output中的export,导出txt文件,只导出我们想要的AAPC结果,并勾选红框中的选项即可。
然后打开结果,看下数据形式
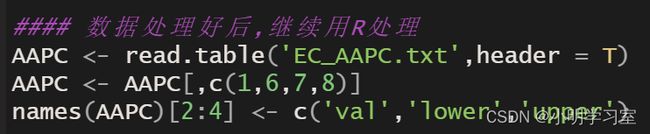
我们再次打开R软件,读取这个文档。
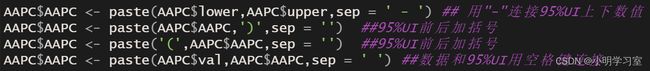
然后运行下述代码,把AAPC与95%CI连接起来,
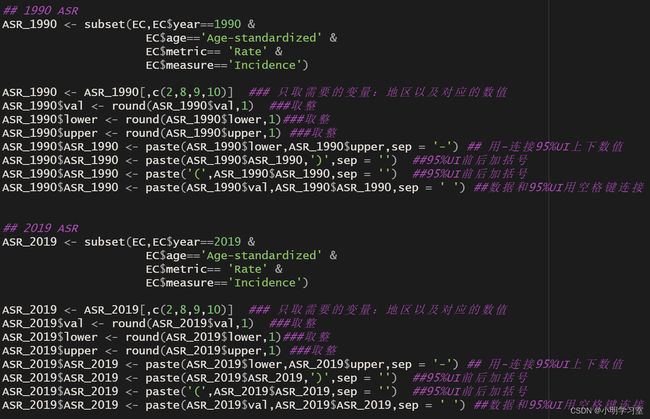
后面我们按照第三篇推文里的方法整理好1990与2019年的食管发病数以及标准发病率
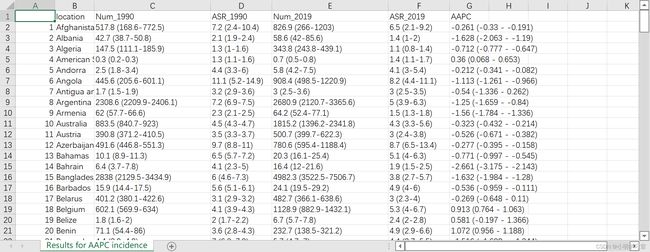
最后进行数据整合,输出文件
最后我们打开文件查看下
可以看到数据合并准确无误,大家就直接可以把内容黏贴至word中,制作成Table 1即可。
下次推文我们讲解如何直接使用R语言调用joinpoint进行分析,大家可以先把软件下下来,进入官网(https://surveillance.cancer.gov/joinpoint/callable/)下载joinpoint的command-line文件即可。
好了,大家可以打开R语言,下载好joinpoint软件实际操练起来,如果需要上述代码和数据,可以关注公众号——小明学习室,回复关键词“GBD”即可获取~
因为微信公众号修改规则,如果不标星或点在看,你可能会收不到我公众号文章的推送,请大家将本公众号标星,看完文章后记得点在看,谢谢。